摘要:本文介绍了Flare的PLIF聚类分析方法,它可以用来快速识别配体数据集中具有相似结合特征的化合物。通过根据配体的相互作用特征进行聚类,PLIF能够高效地支持虚拟筛选的初步过滤,并为SAR提供关键见解。无论是在指导基于片段的药物发现,还是在探索化学空间方面,这一新功能都具有显著潜力,有助于简化并加速药物研发项目。
作者:Sylvie Sciammetta/2025-04-30
编译:肖高铿
分子指纹能够高效地描述分子结构并快速比较相似性,在药物发现中具有重要价值。虽然2D指纹已被广泛使用,但是3D结构相互作用指纹能提供更详细的蛋白质-配体相互作用信息,在药物发现项目中尤其有用。Flare™ 支持生成此类指纹,为研究人员提供一种直观且高效的手段,用于分析复杂的蛋白质-配体相互作用——即蛋白质-配体相互作用指纹(Protein-Ligand Interaction Fingerprint, PLIF)技术。利用PLIF,科学家可以简化工作流程,并深入理解分子与靶标之间的相互作用,从而助力以下项目:
- 深入了解构效关系(Structure-Activity Relationship, SAR)——通过使用相互作用指纹(interaction fingerprints)能够识别在已知配体系列中与特定结合位点所发生的相似相互作用。对这些相似相互作用的分析有助于揭示影响分子生物活性的关键结构特征。
- 提升虚拟筛选的效率——虚拟筛选通常会选择一组结构多样的化合物,以探索更广泛的化学空间。而通过相互作用指纹聚类,则可以在化合物选择中进一步确保蛋白质-配体相互作用的多样性,从而在筛选集合中保留更少但更具代表性的化合物,使需要购买和实验验证的化合物数量最小化。
- 基于片段的药物发现——通过对片段与其在蛋白质结合位点上的相互作用进行聚类分析,可以识别出多个片段结合的“热点”区域(例如,在未被占据的结合位点内)。这有助于后续通过整合来自不同聚类的结构特征,指导更具活性分子的设计。
- 高效地探索化学空间——通过使用相互作用聚类方法,可以高效地从一个化合物库中识别出具有多样性的分子子集。这有助于研究人员在药物发现过程中更全面地探索不同的结构特征,从而覆盖更广泛的化学空间。同时,这种方法还能帮助识别化学空间中某些被过度代表(即已有太多类似结构)或表示不足(即缺乏相关结构)的区域。这对于指导后续的化合物选择和优化、避免重复研究以及发现新的活性分子具有重要意义。
什么是蛋白-配体相互作用指纹?
Flare的PLIF可以快速识别分子数据集中存在的相互作用模式。在基于配体或片段与靶标蛋白的相互作用聚类分析中,PLIF具有重要的作用。
PLIF由一串数字组成,其中每个数字代表基于每个残基特定类型的相互作用,比如氢键、阳离子-π作用、硫原子-孤对电子相互作用、卤键、芳香环-芳香环相互作用、盐桥、金属配位作用、疏水相互作用以及空间位阻冲突。这些字符串(即指纹)随后可通过层级聚类方法进行聚类分析(见图1)。
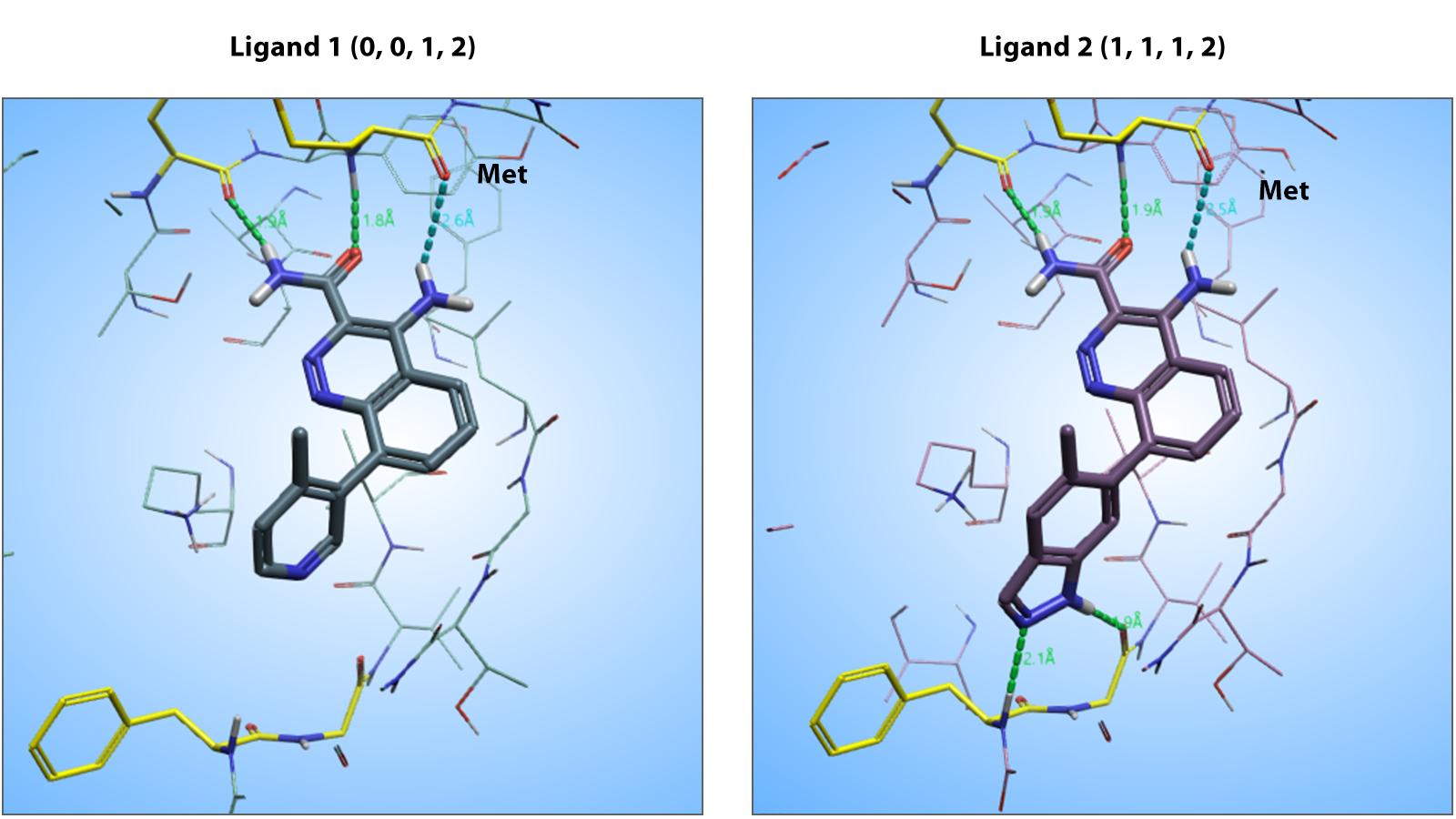
图1. 配体1与配体2的 PLIF。针对每个残基的各种相互作用类型均会生成一个计数,并组合成一串数字,构成指纹。在本例中,所有参与相互作用的残基均形成一个氢键,唯独标注的蛋氨酸残基形成了两个氢键。配体1相比配体2少了两个发生相互作用的残基,因此其指纹中对应位置出现了两个“0”。
如何在Flare里生成PLIF?
在Flare项目中,只需在“3D Pose”标签页下选择“PLIF Clustering”,即可轻松生成PLIF,见图2。该操作会打开“PLIF Clustering Calculation”面板,在此可以:
- 选择需要聚类的配体;
- 指定使用配体的最佳构象(preferred pose)还是所有构象进行分析;
- 选择哪些蛋白质-配体相互作用,这些相互作用将包含在用于聚类的PLIF中。
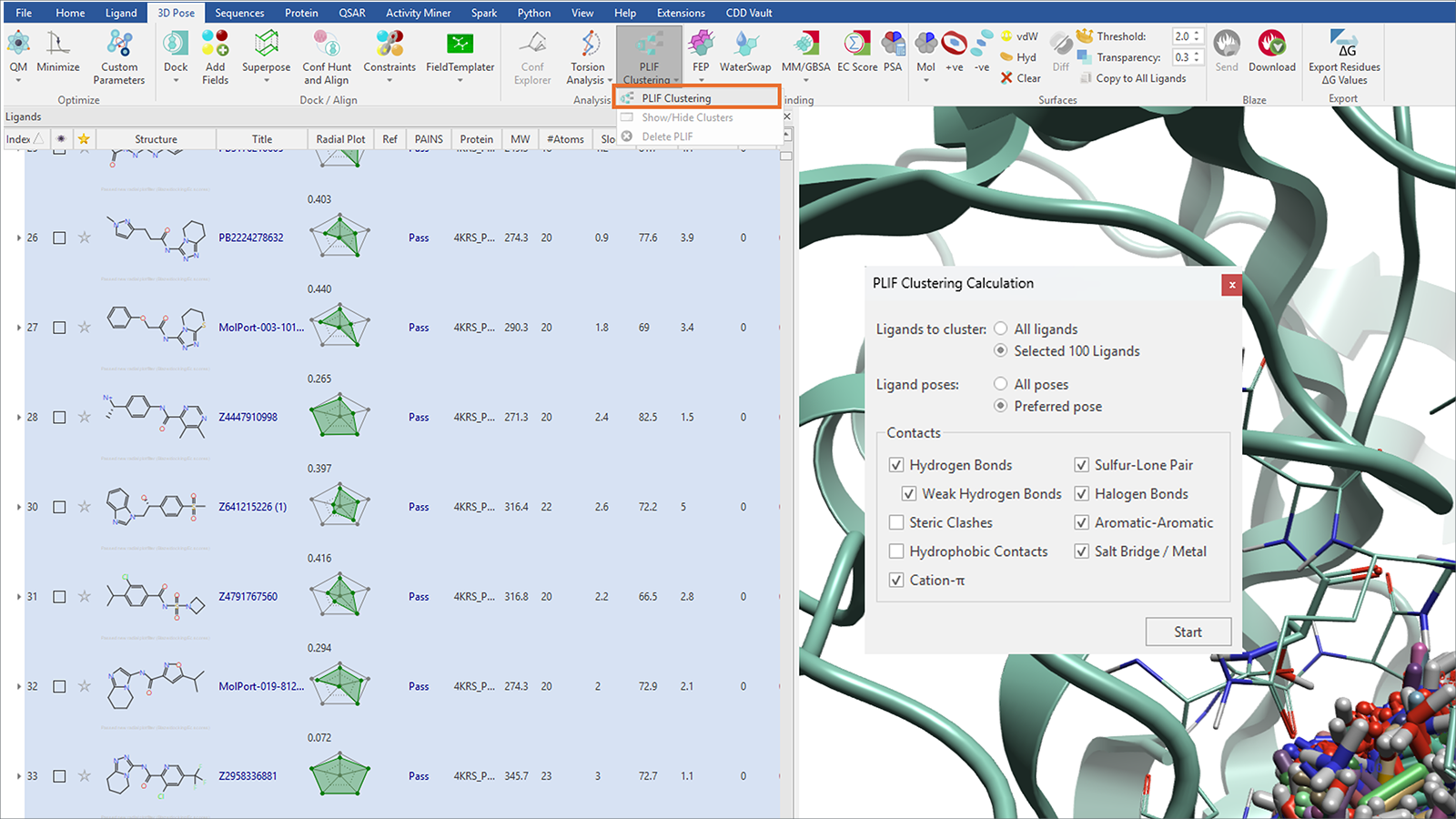
图2. 对TNKS2 Blaze™虚拟筛选的苗头化合物进行PLIF聚类的示例。
PLIF聚类分析会生成一个新的“PLIF聚类视图(PLIF Cluster View)”窗口,其中显示了分子的树状图,见图3。通过右键点击该树状图,可以将聚类结果以标签(Tags)形式或包含聚类成员ID的新列形式汇总到配体表单中。此外,还可以调整距离阈值(默认值为0.5;对于Blaze虚拟筛选的结果,可能更适合使用更高的阈值,例如0.8)。

图3. 在“PLIF聚类视图(PLIF Cluster view)”窗口中生成并显示的分子树状图,该结果可以轻松地在配体表单中的“Cluster ID”列或“Tag”列中进行汇总。
如果选择生成列数据,则会创建两列:“PLIF Cluster ID“ 和 “PLIF Cluster Size”,后者表示该聚类中的分子数量(见图4)。PLIF提供了指纹信息,可以通过将 [data] 占位符复制/粘贴到文本编辑器或Flare日志文件中来访问这些信息。
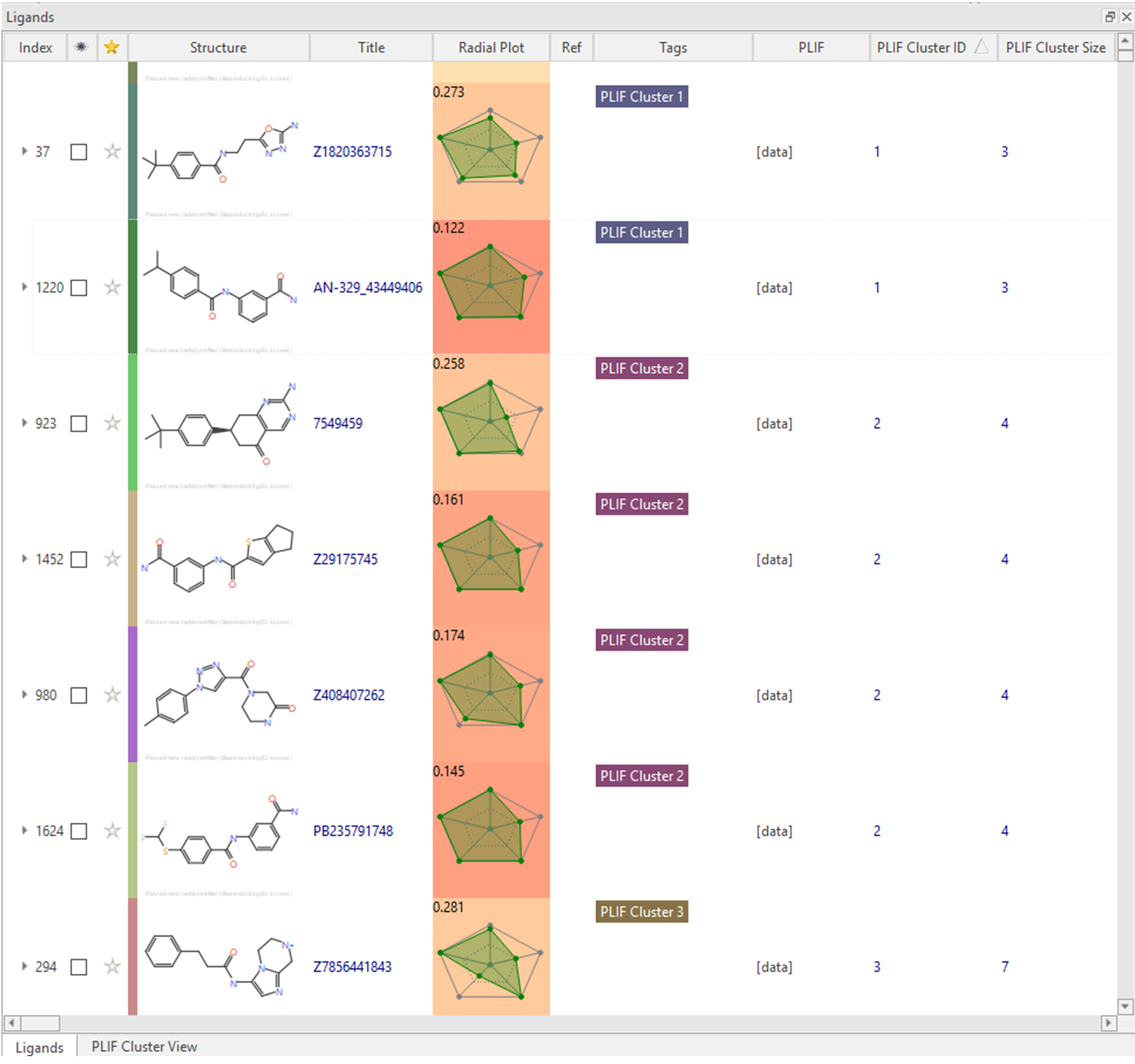
图4. 生成“PLIF Cluster ID”、“PLIF Cluster Size”列以及“Cluster ID Tags”用于轻松识别与靶标蛋白发生相似相互作用的、属于同一聚类的分子,它们与靶标蛋白发生相似的相互作用。
在你自己的项目中试用PLIF聚类
Flare的PLIF聚类可以快速识别配体数据集中具有相似结合特征的化合物。通过根据配体的相互作用特征进行聚类,PLIF能够高效地支持虚拟筛选的初步过滤,并为SAR提供关键见解。无论是在指导基于片段的药物发现,还是在探索化学空间方面,这一新功能都具有显著潜力,有助于简化并加速药物研发项目。
如果您希望在您的项目中试用PLIF聚类功能,请立即申请免费评估版的Flare。
- 电邮:info@molcalx.com
- 电话:020-38261356
关于作者

PhD. Sylvie Sciammetta
Sylvie在英国利兹大学获得有机化学博士学位后,在瑞士日内瓦大学担任博士后研究研究员。2000年,西尔维加入BioFocus(现为查尔斯河实验室的一部分),领导并推进了多个G蛋白偶联受体(GPCRs)的药物化学项目,成功达成一系列合作里程碑。2017年,Sylvie加入Cresset公司,担任应用科学家,为北美地区的客户提供科学与技术方面的支持。2021年,她转入Cresset Discovery团队,目前继续为制药与生物技术行业的客户开展支持工作,负责交付基于结构与基于配体的药物设计项目。



