摘要:本文用二肽基二肽酶IV抑制剂(DPP-IV) 的侧链替换来说明如何用SPARK的片段数据库和高级过滤功能来控制设计出来化合物的理化性质空间,您还可以了解到如何用SPARK快速地生成新的DPP-IV抑制剂,并设计出各种性质良好的、具有自主知识产权的新化合物。
前言
SPARK的片段数据库和高级过滤功能可以用来控制结果的理化学性质的空间。在本文中,我们用二肽基二肽酶IV抑制剂(Dipeptidyl Peptidase IV, DPP-IV) 的侧链替换来说明这些功能特性,您可以了解到如何用SPARK快速地生成新的DPP-IV抑制剂,并设计出各种性质良好的、具有自主知识产权的新化合物。
实验部分
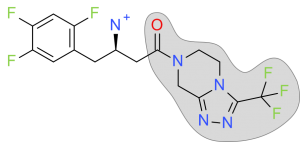
Figure 1. 起始结构
下载PDB结构1X70,读入SPARK,蛋白导入工具将文件被分成为配体和蛋白的排斥体积(仅保留其中A链)两个部分。我们选择配体的头部作为要被替换的部分(Figure 1),然后选择chembl_common、Very Common, Common, Less Common与Rare Database数据库开始进行基团替换。
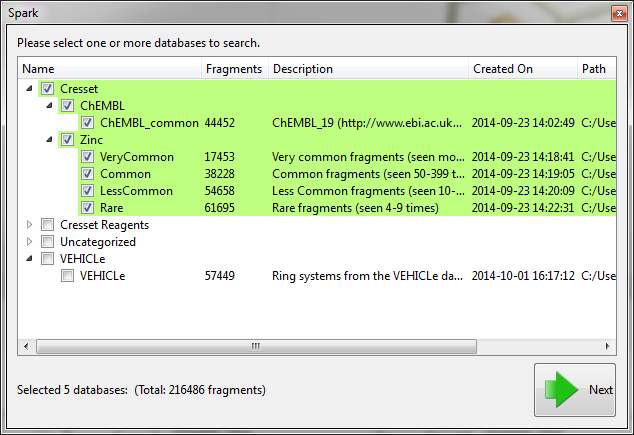
Figure 2. 选择片段数据库
在开始“Accurate but slow”实验之前,我们修改了几个参数。在Score标签页限制输出结果的数量为200,将梯度截断值设为0.1kcal/mol/A以改善最终分子的优化结果。接着,在’Size Criteria’标签页将片段允许的旋转键数设为3。
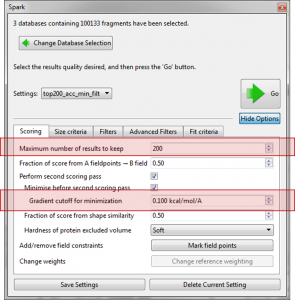
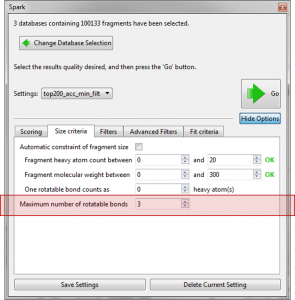
Figure 2. 设定生成的化合物数量、优化的梯度与可旋转键的数量
最后,用”Advanced Filter”标签来控制最终生成分子的性质以便我们仅要那些logP为1-3.5,TPSA为40-90的分子。还可以将设置保存下来以便以后还继续用同样的设置,比如保存为“top200_acc_min_filt”
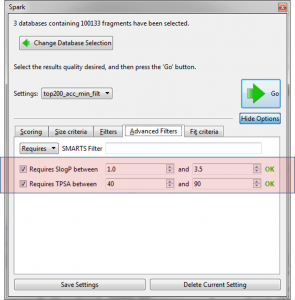
Figure 3. 控制生成分子的性质比如logP与TPSA取值范围以及保存设置
计算
用Cresset Engine Broker将计算从笔记本分布到Linux集群上。用20个CPU不到30分钟计算完毕。
结果
我们用平铺视图来查看结果。将打分值、分子量、LogP、TPSA与2D相似性用雷达图展示出来,并展示了排名、数据库来源以及不稳定标记,下图展示了前三十个结果的屏幕截图。
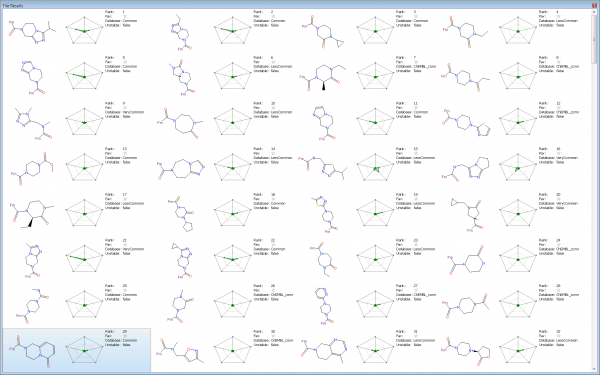
Figure 4. 结果展示:Tile风格
头两个结果与提问式属于同系物(其中一个为已知的nM活性级别的抑制剂),这是一个很好的验证,这说明计算实验设置合理。接下来的几个3-oxo-piperazine也是已知DPP-IV活性的骨架。
令人感兴趣的是,SPARK推荐、排名第6的3-oxo-tetrahydro-imidazopyrazine与起始分子非常不一样,它具有优良的性质(在雷达图里,阴影区域面积小)。这也是一个已知的pIC50=5.9的DPP-VI抑制剂。排名第9的分子为初始分子的开环化合物,排在第10的1,4-diazepanone,这又是一个已知的DPP-IV活性化合物。接下来的几个结果与已知的DPP-IV活性化合物骨架相似。
接着往下看,排序29的一个化合物具有新的骨架,3,4-dihydro-1h-pyrido [1,2-a] pyrazin-6-one,它在DPP-IV活性化合物里没有出现过。这个化合物看起来很有趣,因为它具有良好的性质以及进一步改进的空间。根据表单,可以发现,该片段赖在”Common”数据库,并且可以买的到。
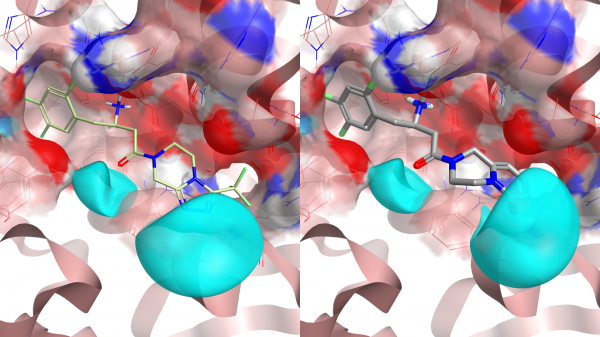
Figure 5. SPARK计算结果截屏:比较排名29的化合物(右边)与初始化合物(左边)会发现杂环部分有负的静电势出现。
结论
结果表明:SPARK只需30分钟不仅可以找到许多”已知“的骨架替代化合物、创造出性质理想的活性化合物,而且还能生成结构全新具有自主知识权的新化合物。通过平铺视图可以轻而易举、迅速地找到最理想的骨架或侧链替换结果。



