摘要:抑制FABP4的小分子可能是治疗代谢综合征的有用候选药物。在本文中,作者将Field-Based 3D-QSAR方法与Spark骨架跃迁(scaffold hopping)相结合,使得设计能够与结合位点相互作用并抑制FABP4的新型强效分子成为可能。在这个方法中,包含了三个关键步骤:1)开发一个3D-QSAR模型,用于阐明构效关系(SAR);2)用Spark进行骨架跃迁,生成FABP4抑制剂虚拟库;3)用第1步的3D-QSAR模型来预测第2步新设计分子的FABP4活性。最后合成了三个活性预测值排名靠前的骨架跃迁提出的配体,并进行了体外活性测试,结果发现它们的体外IC50值在3.70-5.59 μM之间,这与3D-QSAR模型的预测值非常一致,这证明了该方法的有效性。
前言
脂肪细胞脂肪酸结合蛋白(A-FABP, aP2 或 FABP4)在脂肪细胞中高表达,并且受过氧化物酶增殖活化受体-γ(PPAR-γ)激动剂以及胰岛素和脂肪酸水平的调控1。对FABP4基因敲除小鼠的研究表明,FABP4在代谢综合征的许多方面起着关键作用2,3,显示出缺乏这种蛋白质可以部分防止小鼠胰岛素抵抗和肥胖的发展。抑制FABP4功能的药物可以模拟FABP4缺陷小鼠的表型,而抑制FABP4的小分子可能是治疗代谢综合征的有用候选药物。因此,抑制FABP4功能的药物可以模拟FABP4缺陷小鼠的表型,而抑制FABP4的小分子可能是治疗代谢综合征的有用候选药物。
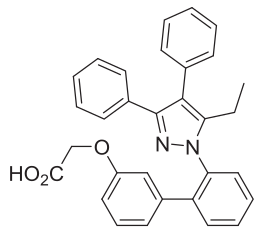
图1. BMS309403的化学结构式
虽然人们已经发现了多种有效的FABP4抑制剂,但是目前还没有任何一种进入临床研究阶段,只有BMS309403(图1)在体外和体内动物模型中进行了系统性的研究4-6。人们为发现结构新颖的苗头化合物持续努力,最近Floresta等人7报道了一种将3D-QSAR模型与骨架跃迁分析相结合的方法,使得设计能够与结合位点相互作用并抑制FABP4的新型强效分子成为可能。在Floresta等人7的研究中,包含三个步骤:1)使用Forge软件开发了一个3D-QSAR模型,用于阐明构效关系(SAR);2)用Spark进行骨架跃迁,生成FABP4抑制剂虚拟库;3)用第1步的3D-QSAR模型来预测第2步新设计分子的FABP4活性。最后合成了三个根据骨架跃迁提出的配体,并进行了合成与体外活性测试,结果发现它们的体外IC50值在3.70-5.59 μM之间,这与3D-QSAR模型的预测值非常一致。
本文的主要目的是简要描述Floresta等人7研究的方法与结论,展示Forge 3D-QSAR(现在是Flare 3D-QSAR)与Spark联合使用作为高效骨架跃迁的强大能力,证明了该方法在真实世界骨架设计中的重要作用。
结果与讨论
建立3D-QSAR模型
使用Forge的Field-Based 3D-QSAR来建立回归模型,采用的方法是偏最小二乘法(PLS),具体算法为SIMPLS,详细的建模过程见《 Data in Brief》文章8。最初尝试了两种不同的分子叠合方法(即基于结构的和基于配体的)。通过多个统计测试确认了生成的QSAR模型的预测能力。交叉验证回归系数(q²)是基于PRESS(预测误差平方和)和SSY(实验值与其平均值偏差的平方和)计算得出的:
$$
q^2 = {1 – PRESS \over SSY} = 1 – {{\sum_{i=1}^{n}(Y_{exp} -Y_{pred})^2} \over {\sum}_{i=1}^{n}(Y_{exp} -Y_{mean})^2}
$$
其中:
- Yexp = 训练集化合物的实验活性值
- Ypred = 训练集化合物的预测活性值
- Ymean = 训练集化合物的活性平均值
两个模型的统计结果见表3和图2。
表3. 3D-QSAR模型的性能指标
| Alignment protocols | Leave-one-out q2 | Training set r2 |
|---|---|---|
| based on docking pose (structure-based) | 0.38 | 0.90 |
| based on Forge alignment (ligand based) | 0.64 | 0.92 |
如图2与表3所示,基于配体的方法结果显示比基于结构的叠合方法更为可靠(r² = 0.92, q² = 0.64),后者具有较高的噪音(r² = 0.90, q² = 0.38)。
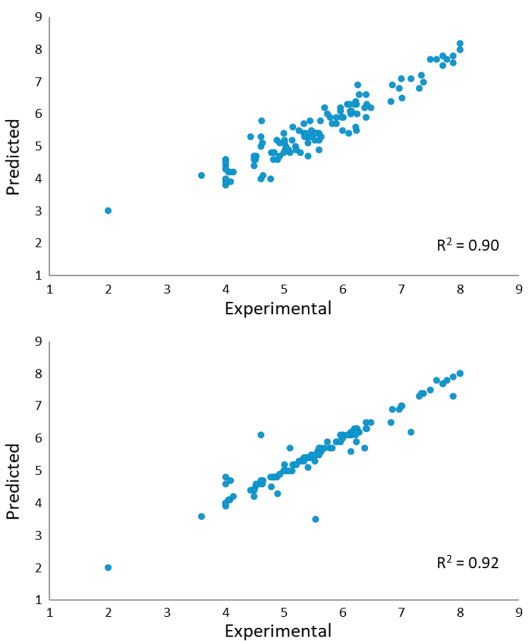
图2. Field Based 3D-QSAR模型——实验值 vs 预测值。上:基于对接结合模式(基于结构);下:基于Forge叠合(基于配体)
在此基础上,使用一组外部化合物(即测试集)进一步验证了基于配体的3D-QSAR叠合模型。实际上,更可靠的稳健性评估来自于独立的训练集和测试集。在120个分子中,随机选取了96个分子(覆盖了所有化合物活性的全范围)作为训练集来构建模型,而剩下的24个化合物则作为测试集来评估模型。
该模型的统计可靠性还通过确定r²_test来验证,使用以下公式:
$$
q^2 = 1 – {{\sum_{i=1}^{n}(Y_{predtest} -Y_{test})^2} \over {\sum}_{i=1}^{n}(Y_{test} -Y_{mean})^2}
$$
其中:
- Ypredtest = 测试集化合物的预测活性值
- Ytest = 训练集化合物的实验活性值
- Ymean = 训练集化合物的活性平均值
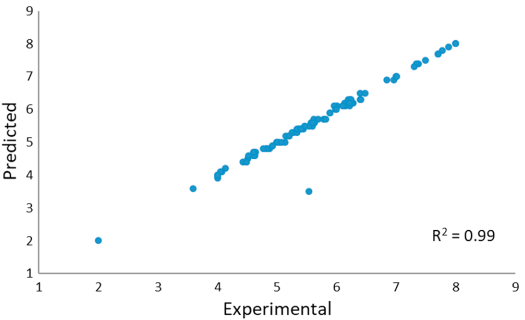
图3. 11个主成分的3D-QSAR模型——训练集化合物的实验值 vs 预测值。
结果如图3和图4所示。一个含有11主成分的模型展示了良好的预测能力和描述能力,这从训练集和交叉验证训练集的良好r²(0.99)和q²(0.69)值可以看出来。训练集和交叉验证训练集(q² = 0.69)中化合物的实验活性与预测活性之间的对比图显示了合理的数值分布。测试集中化合物的实验活性与预测活性之间的对比图仍然相当不错,仅有少数离群点,并且具有良好的交叉验证r²值0.73。
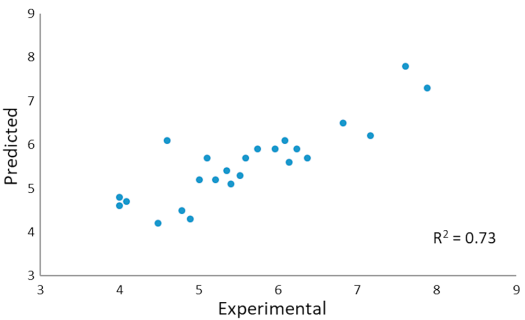
图4. 11个主成分的3D-QSAR模型——测试集化合物的实验值 vs 预测值。
模型的图形解释如图5所示,其中3D-QSAR模型的结果与BMS309403的结构叠合在一起。在这个模型中,点越大,则该位置的静电/立体场之间的相关性越强。这个3D-QSAR模型明显由立体效应和静电效应共同描述,并很好地描述了不同类别的FABP4抑制剂在FABP4结合口袋内的行为。
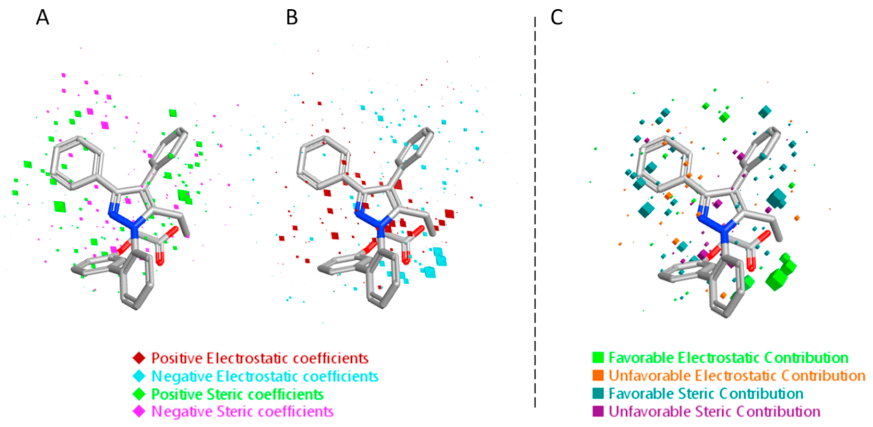
图5. FABP4模型的静电与立体效应系数图与BMS30943的叠合. (A) 立体系数 (B)静电系数 (C) BMS309403亲和力预测值的贡献来源
为了揭示所研究系列的关键特征,还通过Forge的Activity atlas9的活性悬崖总结分析对结构-活性关系(SAR)进行可视化研究。图6展示了Activity atlas计算的结果,其中活性悬崖总结分析图与BMS309403先导化合物叠加在一起。图上的不同颜色突出了静电、疏水性和形状特征对活性的影响。在红色区域,更正的静电场会增加受体亲和力;而在蓝色区域,更负的静电场则会增加亲和力。绿色和紫色区域代表立体效应和体积/疏水性相互作用。在绿色区域,立体/体积相互作用可以改善结合亲和力。相反,在紫色区域,立体/体积相互作用则会降低亲和力。
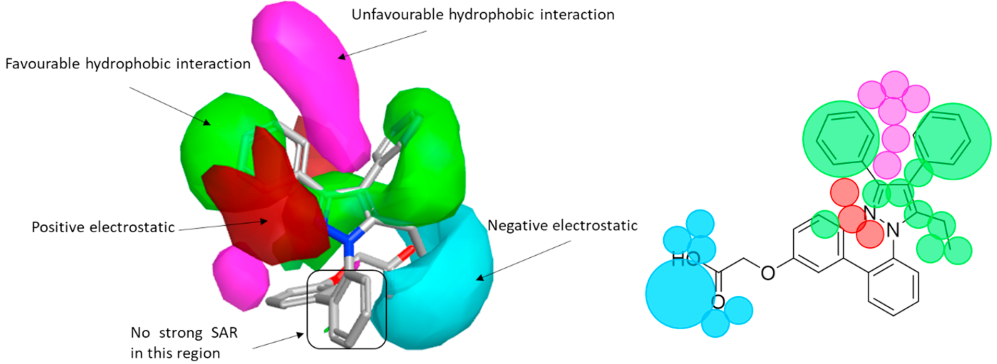
图6. FABP4抑制剂的Activity Atlas活性悬崖总结分析图与BMS309403的叠合。红色:正静电场对活性有利的区域,蓝色:负静电场对活性有利的区域。绿色:形状/疏水对活性有利的区域,紫色:形状/疏水对活性不利的区域。
Spark骨架跃迁实验
为了设计具有FABP4抑制活性的新苗头化合物,Floresta等人7决定利用上述从QSAR模型获得的信息,采用生物电子等排体和片段替换软件工具Spark10进行骨架跃迁实验,以生成一个虚拟的FABP4配体库。如图7所示,对BMS309403的不同片段用Spark进行生物电子等排替换实验研究。
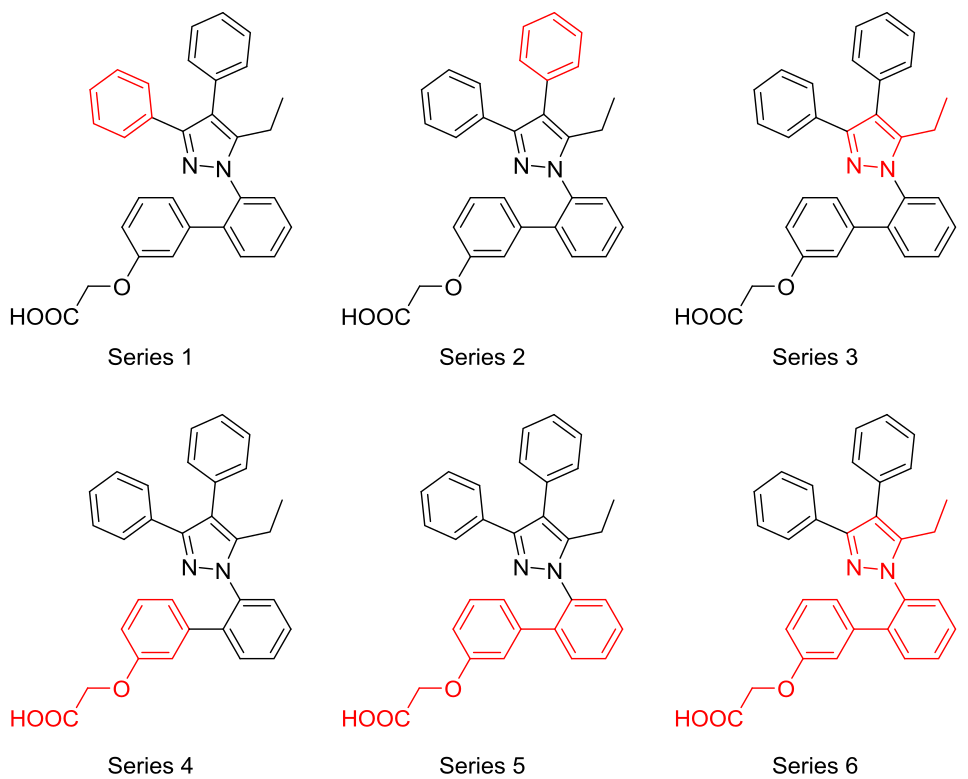
图7. 先导化合物BMS309403的6种生物等排体替换策略
对骨架跃迁结果进行活性预测、识别新的化合物
一旦创建了虚拟库,新设计的分子通过前面建立的Field-Based 3D-QSAR模型来预测其潜在的生物活性,假设如果它们计算/叠合的场与3D-QSAR模型中原有化合物的高度相似,那么预期的生物活性也会相似。对于每个片段,生物电子等排替换是通过相同的178,558个片段库完成的,这些片段来源于ChEMBL和Zinc数据库,具体细节参见《Data in Brief》文章8。每次生物等排体替换实验生成500个化合物,总共产生了3000个苗头化合物,随后这些化合物用3D-QSAR模型进行了活性预测。
系列1至6中生物电子等排体替换后打分最靠前的化合物(预测的最佳pIC50值)如图8所示,而所有化合物的完整集合以SMILES格式见《Data in Brief》文章8。总体而言,结果表明,通过生物电子等排体替换以及随后的3D-QSAR模型评估所生成的化合物具有适合抑制FABP4的化学结构。
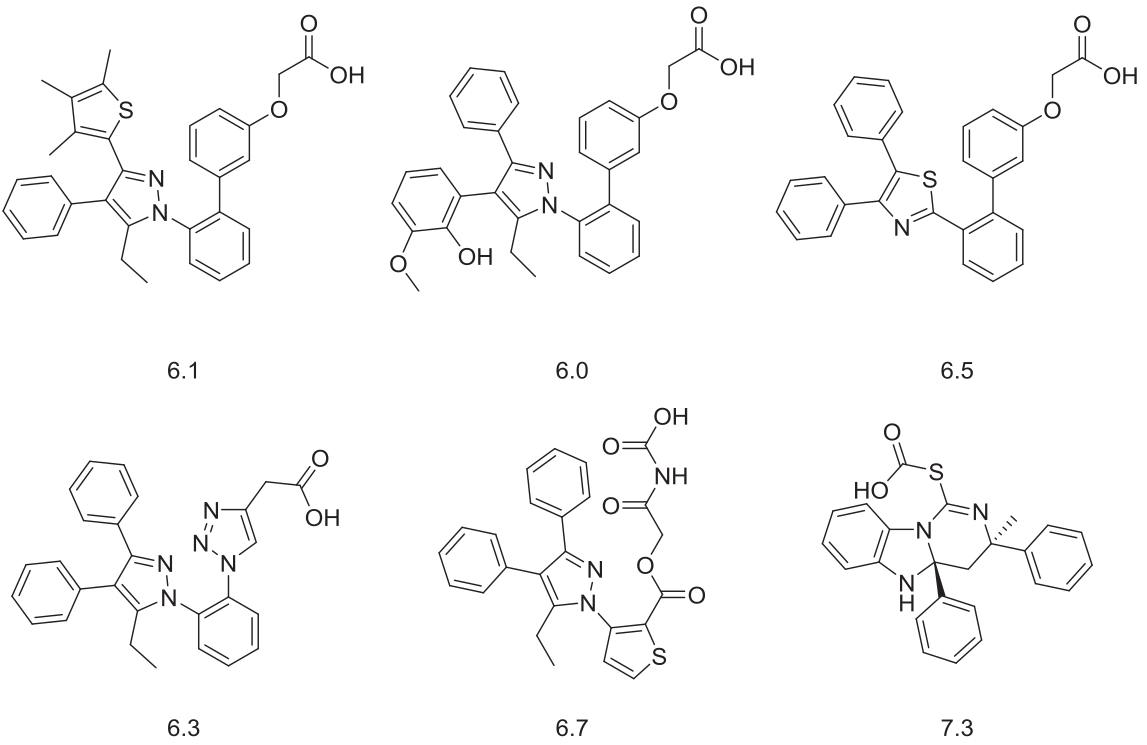
图8. 首批在系列1至6的骨架跃迁结果中FABP4 pIC50预测值靠前的部分化合物
骨架跃迁分析的结果被用于设计一系列基于BMS309403的新衍生物。特别地,将注意力集中在系列3和系列4的结果上。在系列3中,预测最具活性的化合物是BMS309403的一个噻唑衍生物,其中中心的吡唑环被噻唑环取代。在系列4中,预测最具活性的化合物含有一个三氮唑环作为芳香连接臂,该基团可以通过点击化学反应轻松引入。利用这些信息,设计了新的化合物系列AST_1-3(图9)。将这些分子导入Forge,并用3D-QSAR模型进行叠合并预测活性。
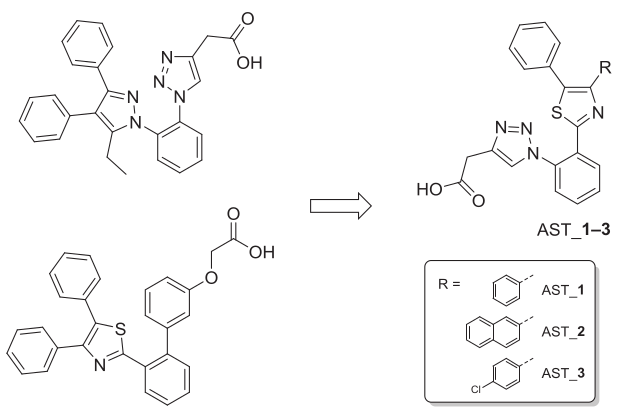
图9. AST_1–3的化学结构式
令人感兴趣的是,AST_1-3预测的pIC50值分别为5.4、5.9和5.6,其对应的IC50值分别为3.98、1.25和2.51 μM。
合成与活性评估
为了证明模型的有效预测能力,建立一条能够合成AST_1-3的合成路线,并进行体外FABP4活性测试。抑制活性数据以IC50(µM)表示,AST_1-3的结果见表4。已知的强效FABP4配体——花生四烯酸用作阳性对照,显示的IC50值为3.06 µM。三个新的化合物,即AST_1-3,分别显示出IC50值为5.59、3.70和4.31 µM,这与前面的预测值基本一致。
表4. 花生四烯酸与AST_1–3的FABP4 IC50值
| Compound | IC50 (μM) |
|---|---|
| Arachidonic acid | 3.06+/-0.22 |
| AST_1 | 5.59+/-0.79 |
| AST_2 | 3.70+/-0.34 |
| AST_3 | 4.31+/-0.67 |
结论
FABP4在多种动物模型中被证明是一个有吸引力的治疗和诊断靶点。在本研究中,作者用已知的FABP4抑制剂建立了Field-Base 3D-QSAR模型,并通过Spark骨架跃迁实验建立一个庞大的虚拟分子库,以寻找可能的FABP4抑制剂。QSAR模型的有效预测能力得到了证实,这通过合成三种新的FABP4抑制剂并使用从QSAR方程获得的信息来证明。新设计的化合物(AST_1–3)对FABP4的结合亲和力通过体外试验测定,结果显示它们是低微摩尔范围内的FABP4抑制剂(即IC50值介于3.70至5.59 μM之间)。此外,还有很多不同的分子被3D-QSAR理论模型预测为与这三个合成的分子一样有效,可以作为设计其他FABP4抑制剂的有用起点。结果表明,在金标准BMS309403的不同位点进行简单修饰可以保留甚至提高类似物的FABP4抑制活性。当然,这些发现需要通过更多实验验证,但可作为该领域研究的一个合理起点。毫无疑问,这些发现将引导未来在FABP4抑制剂开发领域的研究。
文献
- M.I. Queipo-Ortuno, X. Escote, V. Ceperuelo-Mallafre, L. Garrido-Sanchez, M. Miranda, M. Clemente-Postigo, R. Perez-Perez, B. Peral, F. Cardona, J.M. Fernandez-Real, F.J. Tinahones, J. Vendrell, FABP4 dynamics in obesity: discrepancies in adipose tissue and liver expression regarding circulating plasma levels, PLoS One 7 (2012) e48605.
- B.R. Thompson, A.M. Mazurkiewicz-Munoz, J. Suttles, C. Carter-Su, D.A. Bernlohr, Interaction of Adipocyte Fatty Acid-binding Protein (AFABP) and JAK2 AFABP/aP2 AS A REGULATOR OF JAK2 SIGNALING, J. Biol. Chem. 284 (2009) 13473–13480.
- A. Adida, F. Spener, Adipocyte-type fatty acid-binding protein as inter-compartmental shuttle for peroxisome proliferator activated receptor gamma agonists in cultured cell, Bba-Mol. Cell Biol. L 1761 (2006) 172–181.
- A. Bosquet, J. Girona, S. Guaita-Esteruelas, M. Heras, P. Saavedra-Garcia, N. Martinez-Micaelo, L. Masana, R. Rodriguez-Calvo, FABP4 inhibitor BMS309403 decreases saturated-fatty-acid-induced endoplasmic reticulum stress-associated inflammation in skeletal muscle by reducing p38 MAPK activation, Biochim. Biophys. Acta Mol Cell Biol. Lipids 2018 (1863) 604–613.
- C. Look, I. Morano, M. Ehrhart-Bornstein, S.R. Bornstein, V. Lamounier-Zepter, BMS309403 directly suppresses cardiac contractile function, Naunyn Schmiedebergs Arch. Pharmacol. 384 (2011) 255–263.
- M.Y. Lee, H. Li, Y. Xiao, Z. Zhou, A. Xu, P.M. Vanhoutte, Chronic administration of BMS309403 improves endothelial function in apolipoprotein E-deficient mice and in cultured human endothelial cells, Br. J. Pharmacol. 162 (2011) 1564–1576.
- Floresta, G. et al. (2019) “3D-QSAR assisted identification of FABP4 inhibitors: An effective scaffold hopping analysis/QSAR evaluation,” Bioorganic Chemistry, 84, pp. 276–284. Available at: https://doi.org/10.1016/j.bioorg.2018.11.045.
- Floresta, G. and Cilibrizzi, A. (2019) “Data in Brief FABP4 inhibitors 3D-QSAR model and isosteric replacement of BMS309403 datasets,” Data in Brief, 22, pp. 471–483. Available at: https://doi.org/10.1016/j.dib.2018.12.047.
- Activity Atlas. https://cresset-group.com/software/activityatlas
- Spark. https://cresset-group.com/software/spark
联系我们
想在自己的项目中亲自使用Field-Based 3D-QSAR与Spark骨架跃迁,请联系我们获取免费的试用版;或者联系我在线演示功能;你还可以采购软件或委托研究与我们进行项目合作:info@molcalx.com。
原创文章,作者:小墨,如若转载,请注明出处:《3D-QSAR与Spark生物等排体替换联合使用——高效骨架跃迁设计新型FABP4抑制剂》http://blog.molcalx.com.cn/2025/01/15/3d-qsar-assisted-identification-of-fabp4-inhibitors.html



