摘要:基于片段的药物发现是一个不断发展的研究领域,将苗头化合物转化为先导分子是其独特的挑战。在本案例研究中,我们用生物等排体解决方案Spark™来演示基于结构的虚拟筛选如何高效地识别用于结核病(TB)治疗的新型InhA还原酶抑制剂。我们演示了如何使用Spark中的对接打分功能将片段往活性位点的新区域生长以发现新的化合物。
作者:Scott Midgley†, Nathan Kidley†
单位:†Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
前言
结核病(tuberculosis,TB)仍然是21世纪的主要死亡原因。烯酰-[酰基载体蛋白]还原酶(Enoyl-[acyl-carrier-protein] reductase,InhA)是结核病小分子治疗研究中的一个热门靶点,然而,许多领先的治疗化合物需要通过NAD通路激活才能抑制这种酶。阻断这种激活是已知的抗结核药物耐药机制,因此,在没有代谢激活的情况下抑制InhA的治疗是一个令人感兴趣而且活跃的研究领域。这是Sabbah等人的目标1,他们进行了基于片段的筛选研究,然后优化了碎片苗头化合物。在本研究中,我们证明了Spark2使用对接方法可以将片段生长到InhA的活性位点,独立地识别出在实验中命中的化合物。
Spark中对结果进行打分的传统方法主要是基于配体的方法。Cresset的场相似性技术与形状相似性度量相结合,将结果与起始分子进行比较,以识别生物等排体。或者,Spark™在打分过程中使用Lead Finder™对接引擎3直接从蛋白和配体开始计算对接打分。这种方法可以让用户能够将配体生长到活性位点未探索的区域,并识别出与靶蛋白产生新相互作用的结果。
在本案例研究中,PDB 6SQ5共晶配体结合到活性位点中与Site I及Site II(图 1),我们以这个配体为起始分子,替换其羧酸片段,同时保留了其与蛋白结构的相互作用,进行往“Site III”方向的分子生长实验。这么做是因为将配体往Site II生长的空间范围有限,但往Site III生长具有增加配体亲和力的潜力。
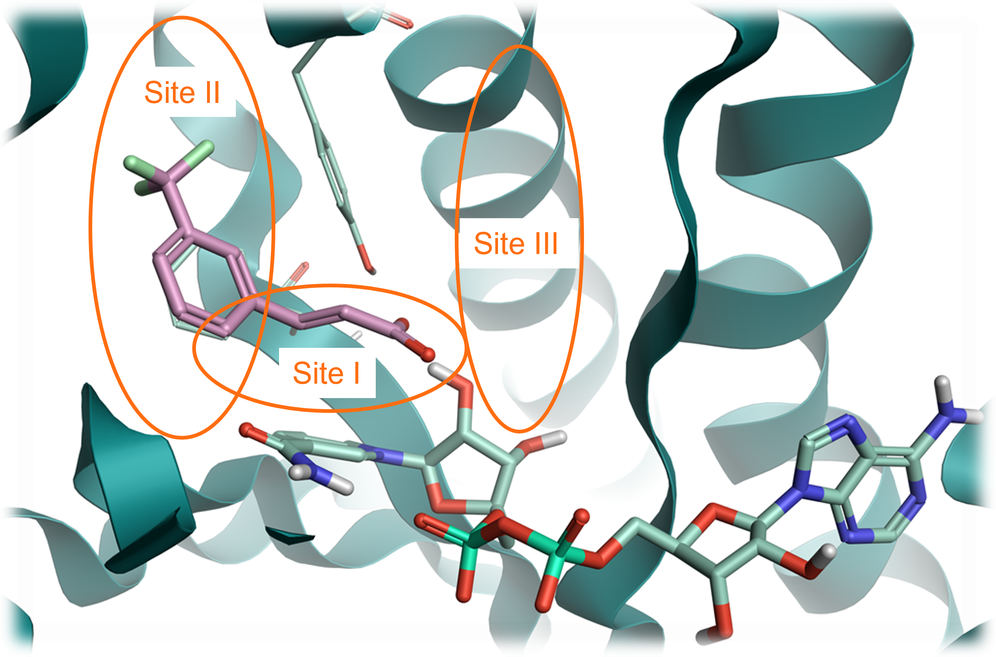
图1. 从PDB 6SQ5可以识别出InhA蛋白结构的三个关键结合位点。Site I:催化位点; Site II: 疏水部位; Site III:溶剂暴露区域。粉红色:进行生长的片段结构。淡绿色:NAD辅因子。绿丝带:InhA蛋白。
在SPARK中进行计算片段筛选
在开始Spark对接实验之前,进行了传统的Spark场与形状相似性搜索,以识别图1中片段羧酸基团的潜在生物等排体。这使得化学搜索空间得以聚焦,为 Spark对接实验做准备。
该搜索的结果识别出了一系列生物等排体,例如亚砜、砜、磺胺、亚砜酰胺、咪唑、吡唑以及三氮唑等等片段。我们专注于羧酸基团的生物等排体砜和磺酰胺,因为合成这些衍生物便捷且容易。这些片段还提供了合适的载体以将配体往Site II 或 III方向生长。
然后采用逐步的方法进行Spark对接实验,包括三个不同的阶段:
- 羧基的生物等排体替换。在这里,对接方法用于起始分子的生长,替换X-衍射共晶配体中的羧基。对片段空间进行过滤使之包含SO2子结构。 此外,应用自动片段大小约束来限制替换片段的大小,使其与羧基的大小相似。
- 从第一阶段打分最高的结果开始进行片段筛选实验。Spark对接实验使用第一阶段打分最高的砜类结果作为起始分子进行进一步的片段生长。该实验没有使用子结构过滤器,但使用了自动片段大小限制。
- 使用蛋白质相互作用约束,对第一阶段打分最高的砜进行片段筛选实验。对接计算时采用了约束以确保结果必须包含与活性位点中NAD分子关键氧原子发生氢键相互作用。
阶段1:羧基的生物等排体替换
我们用对接方法来识别羧基的生物等排体,目的是使配体在活性位点里生长。对接网格是从共晶配体开始创建的,大小设置为8 Å,这足够大,使得片段往Site III生长。用来搜索的片段数据库包括ChEMBL4、 Commercial Very Common与Common5数据库,共包含约100,000 个片段。
在高级搜索选项添加了一个子结构过滤器以便被搜索的片段中必须含有SO2基团,还添加自动片段大小限制以将替换片段的大小限制为羧基的大小相似。这将搜索空间聚集在相对较小的含SO2 的片段上。
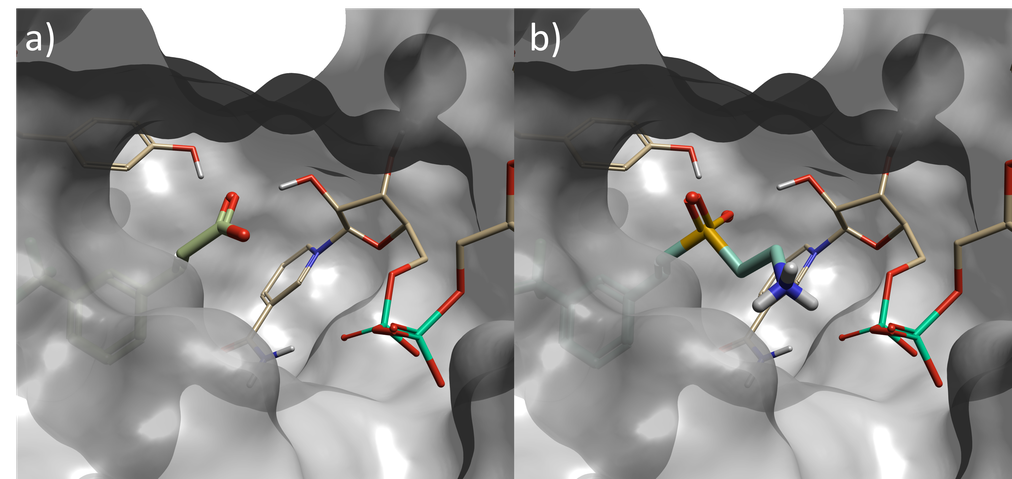
图2. a)PDB 6SQ5蛋白结构的共晶配体作为起始分子,b)在第一阶段 Spark实验中确定的打分最高的砜类化合物,该片段与NAD磷原子的接触比起始分子更紧密。
第1阶段打分最高的结果里产生了多个苗头化合物,其中SO2基团直接取代了原始羧基。还有其他的结果,其中SO2占据了活性位点的不同部位,或有一个原子远离羧基。出现这些变化是因为SO2过滤器不受空间约束,这可通过附着点约束得到改善。
打分最高的结果如图2所示。替换片段中的一个SO2氧原子取向为与活性位点中的酪氨酸残基形成氢键;以及与NAD糖上的羟基形成第二个氢键,模拟了起始分子中羧基的相互作用。此外,乙胺基从砜延伸以与NAD辅因子的磷酸酯多出一个氢键相互作用。这是一种用传统的基于配体的方法无法发现的新相互作用,因为起始配体在活性位点的这个区域没有原子。
阶段2:对已识别的砜进行二次片段筛选实验
在第一阶段识别的打分最高的砜-乙胺化合物作为第二个实验的起始分子(图 3)。选择乙胺基作为要替换的片段(图3中的蓝色晕圈),附着点原子(图 3 中的红色圆圈)被限制为氮原子,以确保本实验的结果仅包原始羧基片段的磺酰胺生物等排体。在本实验中还在高级选项中设置了自动片段大小约束。
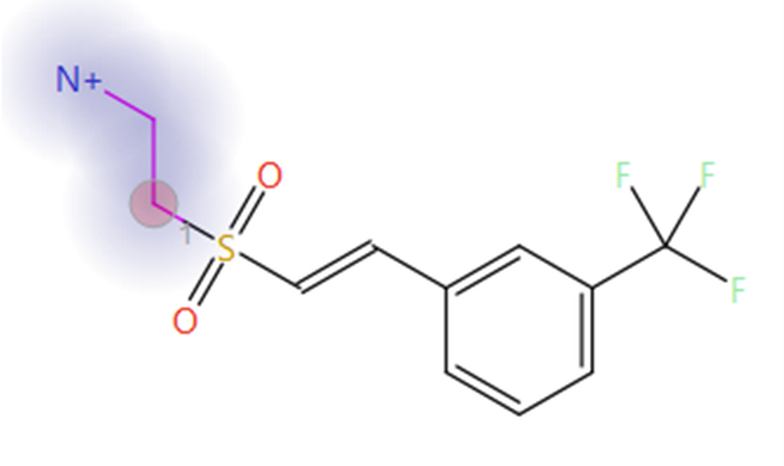
图3.砜-乙胺化合物是第一阶段Spark实验中打分最高的结果,并被用作第二阶段的新起始分子。蓝色光晕:要替换的片段。红圈:附着点。
与第1阶段一样,对Very Commond与Common ChEMBL以及Commercial片段数据库进行了搜索,在本次计算中,在新的起始分子周围设置一个5Å的网格。选择较小的网格是为了降低对接实验的计算费用,而更新的参比分子有助于在实验期间进入到Site III(图 1)。
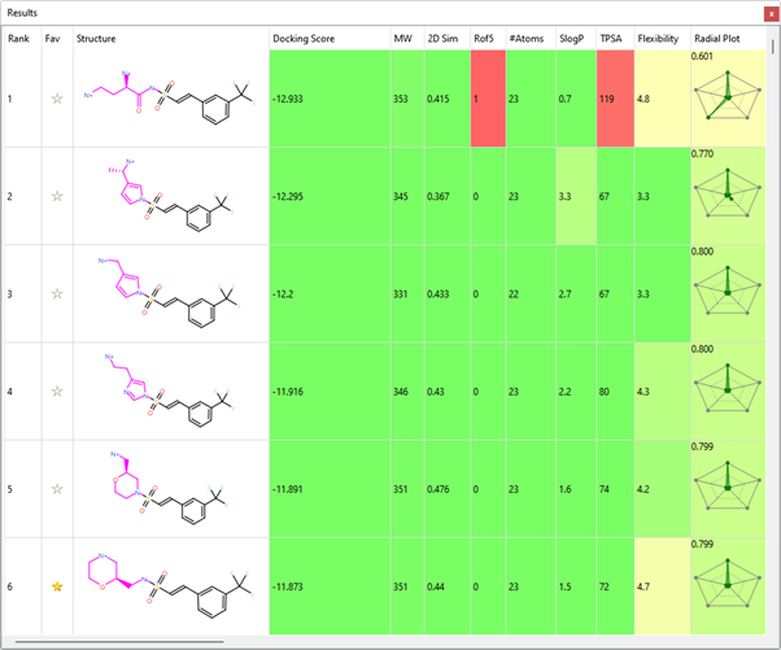
在第2阶段(表 1)中识别出了几个令人感兴趣的磺酰胺替代物,包括图4a和4b中所示的咪唑和吗啉结构,与位于远离参比分子的蛋白和NAD(棕色的碳原子)形成相互作用。
表1. Spark结果表单显示了在第二阶段确定的前6个片段。
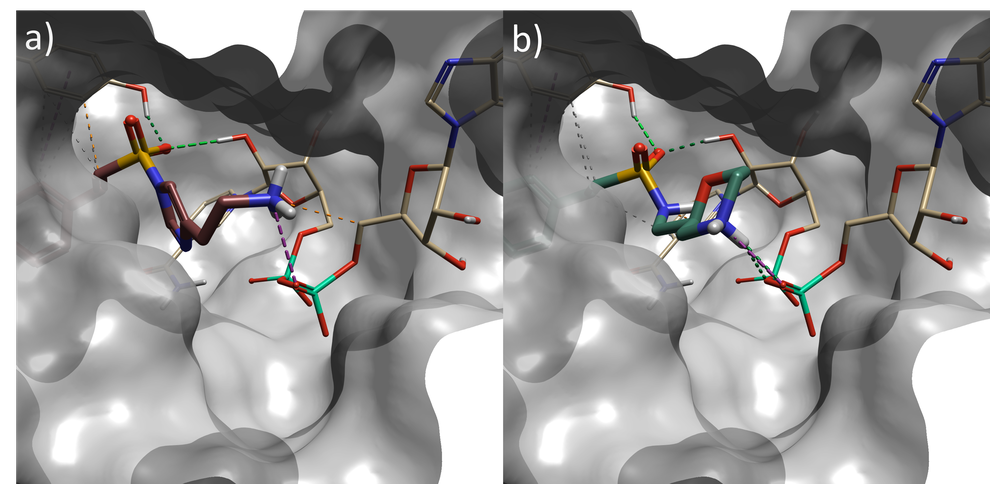
图4. a) 具有咪唑磺酰胺的SPARK结果(result 4),b) 具有吗啉磺酰胺的SPARK结果(result 6)。棕色原子:NAD辅因子;灰色表面:蛋白质活性部位表面。
阶段3:对已识别的砜进行应用蛋白质接触约束进行第三次片段筛选实验
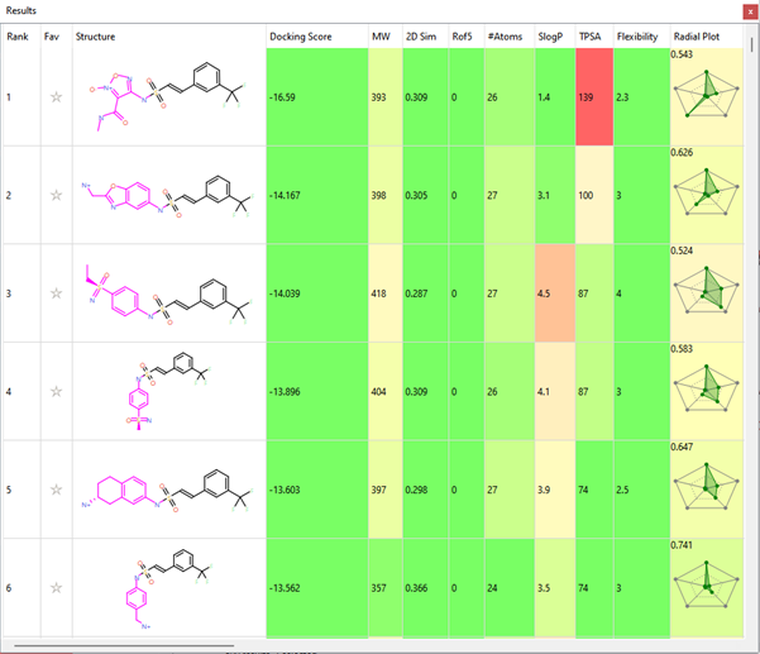
第1阶段和第2阶段进行的Spark实验识别了许多有令人感兴趣的设计。为了补充这些结果,我们进行了一项搜索Spark试剂数据库的实验,以从常见反应中快速识别出可合成处理的化合物。磺酰胺类化合物是通过搜索伯芳香胺片段(保留胺片段)生成的,共大约24,000 个片段4可供搜索。在这个实验中没有用到过滤器。对接计算使用默认的5Å网格,并在NAD氧原子上添加对接约束以优先考虑可以与辅因子形成氢键的化合物。
本次实验的结果识别出了更多令人感兴趣的磺酰胺类化合物(表 2),它们与受约束的NAD氧以及其他蛋白残基形成氢键相互作用。
有趣的是,打分值排名第6的化合物与Sabbah 等人确定为该口袋的最佳片段的是一样的。如图5所示,该化合物的Spark结果表明,末端胺基与在对接过程中受约束的NAD氧原子以及附近的主链羰基形成氢键相互作用。
表2. Spark结果表单显示了在第三阶段确定的前6个片段。
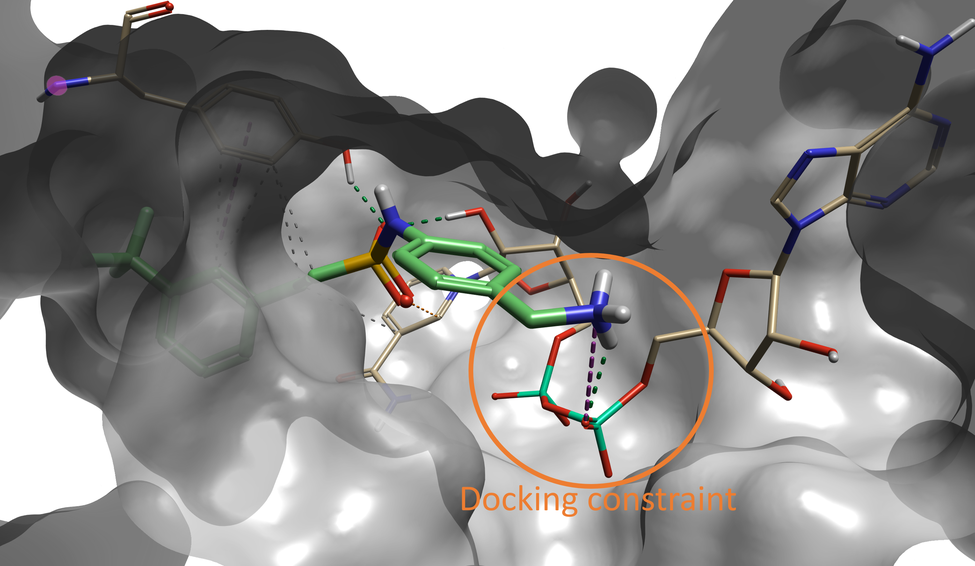
图5. Spark中的化合物#6(共 500 个)是Sabbah等人1在论文中确定的先导化合物。
结论
在本案例研究中,我们用Spark中的对接方法以PDB 6SQ5共晶配体为起始分子往InhA蛋白活性位点的未占用区域进行分子生长,识别出具有与InhA TB靶标公开的实验结果一致的新型配体-蛋白结合特性的设计理念。
对接方法扩展了Spark的能力,即使在缺乏与活性位点发生所需相互作用的已知配体信息的情况下,也能够运行片段生长实验。
过滤器或约束条件的使用极大地有助于将Spark结果聚焦到所需的物理化学和反应空间。在本案例研究中,过滤器用于对化学合成的便利性优先性排序以及与活性位点形成新的相互作用以提升内在活性。
本案例研究使用的工作流仅关注活性位点内的一个区域和两个相关的化学子系列以生成潜在的先导化合物。它可以通过搜索替代试剂来进一步扩展,或者从初始片段苗头化合物的酸基开始替换生物等排体来进一步扩展。此外,可以探索靶向活性位点其他区域的化合物,通过计算探测结合位点以识别可选的化学系列以扩大片段苗头化合物以供进一步开发,这是实时药物发现项目中的实用方法。
文献
- M. Sabbah, V. Mendes, R. G. Vistal, D. M. G. Dias, M. Záhorszká, K. Mikušová, J. Korduláková, A. G. Coyne, T. L. Blundell and C. Abell; Fragment-Based Design of Mycobacterium tuberculosis InhA Inhibitors, J. Med. Chem., 2020, 63, 4749–4761
- Spark™ V10.7. https://www.cresset-group.com/software/spark
- Lead Finder™. https://www.cresset-group.com/software/leadfinder. accessed 12 May 2022
- ChEMBL. https://www.ebi.ac.uk/chembl. Accessed 12 May 2022
- Products: Screening Compounds – eMolecules. https://www.emolecules.com/products/screening-compounds. Accessed 12 May 2022