摘要:诺华生物医学研究院使用骨架跃迁软件Spark对图1化合物6的吲唑母核进行骨架替换以发现代替品。经过对最有希望的、打分靠前的Spark结果进行的视觉选择并进一步优化后发现了新型的LpxC抑制剂,该LpxC抑制剂对LpxC具有亚nM的结合亲和力、对铜绿假单胞菌和其它革兰氏阴性细菌具有体内抗菌活性。
前言
诺华生物医学研究院(Novartis Institutes for Biomedical Research,NIBR)最近在J Med Chem上发表了关于虚拟筛选在发现新化学类型LpxC抑制剂噁唑烷酮和异噁唑啉中的应用的论文[1]。该文报道了使用Cresset的骨架跃迁软件Spark对图1化合物6的吲唑母核进行骨架替换以发现代替品的研究过程。经过对最有希望的、打分靠前的Spark结果进行了视觉选择并进一步优化后发现了新型的LpxC抑制剂,该LpxC抑制剂对LpxC具有亚nM的结合亲和力、对铜绿假单胞菌和其它革兰氏阴性细菌具有体内抗菌活性。
骨架跃迁(Scaffold hopping)
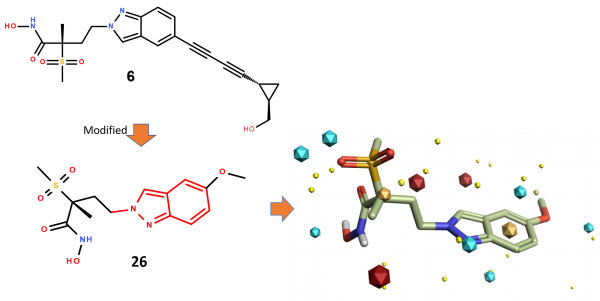
将化合物26(最初由Actelion报道的化合物6的简化形式)的生物活性构象作为Spark骨架跃迁实验的起始分子。研究的目的是确定吲唑母核的合适替代品(图1)。
图1. 上:LpxC抑制剂6。下:化合物26的生物活性构象作为Spark骨架跃迁实验的起始分子。
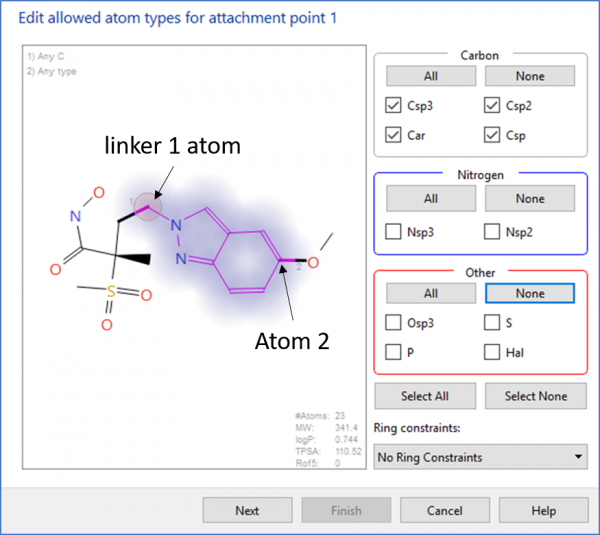
“linker 1 atom”和”atom 2″的允许原子类型(参见图2)分别设置为”任何碳原子”和”任何原子”。还规定,所有SPARK设计必须包含至少一个环,且不应包括任何反应性官能团。SPARK实验对ZINC[2]、ChEMBL[3]、和理论环系的VEHICL[4]的片段数据库进行了搜索。
使用场和形状相似性占比分别为50%来计算SPARK结果对化合物26的相似性打分。手动审查了排序靠前的100个类的合成可行性、在吲唑区域引入亲水性基团的情况以及计算物理化学性质。
图2. 设定‘linker 1 atom’和‘atom 2’允许的原子类型
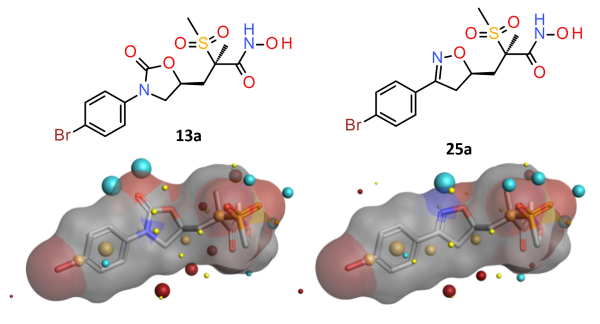
噁唑烷酮13和异噁唑啉25骨架出现在”link 1 atom”和”atom 2″与亲水性连接臂的几个建议中(图3)。衍生物13和25,是对位的甲氧基被溴原子取代(图3)物,显示出对铜绿假单胞菌的最低抑制浓度(MIC)值低于4μg/mL的细胞活性。因此,选择该系列化合物进一步研究。
图3.噁唑烷酮13a和异噁唑啉25a的MIC小于4ug/mL
SAR与优化
进一步对噁唑烷酮和异噁唑啉系列进行了研究、并对其进行扩展最后发现了对铜绿假单胞菌和其它革兰氏阴性细菌具有强体外活性的化合物。代表性化合物13f(图4)在小鼠中性粒细胞减少性大腿感染模型中显示出对铜绿假单胞菌的优异效果。
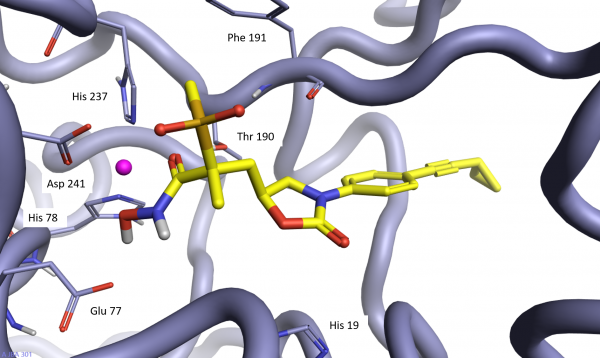
与铜绿假单胞菌LpxC酶(pdb:6mae)共晶的13f的复合物晶体结构表明,异羟肟酸部分与活性位点中的锌原子结合,并与H78、H237、T190、E77和D241的相互作用(图4)。疏水尾(苯基)与几个疏水侧链相互作用,并且环丙基进一步延伸到溶剂暴露区域。砜氧原子与明确定义的结晶水分子(图4中未示出)相互作用,并且与砜官能团连接的甲基与F191发生疏水相互作用。噁唑烷酮的羰基氧与H19的C2CH基团形成有利的极性相互作用。
因此,13f的晶体结构证实了:噁唑烷酮基团可将抑制剂的砜/异羟肟酸部分摆出相对于疏水性苯基有利的取向,同时保持低能构象,与SPARK所预测的一致。
图4. 13f与铜绿假单胞菌LpxC酶(pdb:6mae)复合物的X射线晶体结构
结论
NIBR公司的研究表明,SPARK可以用于指导新药候选药物的合理设计和发现。在本研究中,传统的骨架跃迁发现了对革兰氏阴性细菌具有强抗菌活性的新型噁唑烷酮和异噁唑啉LpxC抑制剂。
文献
- Lee, P. S.; Lapointe, G.; Madera, A. M.; Simmons, R. L.; Xu, W.; Yifru, A.; Tjandra, M.; Karur, S.; Rico, A.; Thompson, K.; et al. Application of Virtual Screening to the Identification of New LpxC Inhibitor Chemotypes, Oxazolidinone and Isoxazoline. J. Med. Chem. 2018, 61 (20), 9360–9370. https://doi.org/10.1021/acs.jmedchem.8b01287.
- Irwin, J. J.; Sterling, T.; Mysinger, M. M.; Bolstad, E. S.; Coleman, R. G. ZINC: A Free Tool to Discover Chemistry for Biology. J. Chem. Inf. Model. 2012, 52 (7), 1757–1768.
- Gaulton, A.; Bellis, L. J.; Bento, A. P.; Chambers, J.; Davies, M.; Hersey, A.; Light, Y.; McGlinchey, S.; Michalovich, D.; Al-Lazikani, B.; Overington, J. P. ChEMBL: A Large- Scale Bioactivity Database for Drug Discovery. Nucleic Acids Res. 2012, 40 (D1), D1100–D1107.
- Pitt, W. R.; Parry, D. M.; Perry, B. G.; Groom, C. R. Heteroaromatic Rings of the Future. J. Med. Chem. 2009, 52 (9), 2952–2963