摘要:PickR最早由诺华与Cresset合作开发挑选DNA编码库(DEL)的砌块(Building Block)而设计的。PickR将Novartis的方法规范化为一个新的计算工具,用于选择结构多样的试剂并合并到化合物库中。本文展示PickR设计多样氨基酸侧链库的算例以展示其性能。
PickR的由来
在2016年的Cresset用户会议上,来自诺华的Nik Stiefl和Finton Sirockin的演讲讨论了使用静电和形状多样性作为关键描述符来选择DNA编码库(DNA Encoded Libraries,DEL)砌块(Building Block,BB)。这项工作由Cresset编写的可执行程序和脚本提供支持。在过去几年中,该方法广泛地应用于多种化合物库设计,为不同类型的化合物库设计提供了新的选择方法而赢得了良好声誉。
PickR将我们与Novartis合作开发的方法规范化为一个新的计算工具。它是一个命令行可执行程序,用于选择结构多样的试剂并合并到化合物库中。与其他方法不同,PickR使用分子的静电和形状性质来生成描述符矩阵并作为结构多样性选择的基础。
使用3D性质产生多样性并不是直接的。为了充分了解R-基团的性质分布,有必要探索R-基团的构象,并围绕定义的连接点将它们进行转动。这在Nik和Finton的演讲(幻灯片12-15)中有详细的描述。更详细的讨论留给PickR正式发布之后进行。
PickR算例:氨基酸侧链的设计
我应用了一个包含大约1000个氨基酸的数据集,这些氨基酸可以从eMolecules购买。首先对原料试剂(Raw reagent)进行预处理,将侧链转化为与C-α 原子连接在一起的R-基团,其中C-α 原子用碘代替(将所有其它含碘试剂变为含Br试剂)。使用PickR生成了侧链的3D相似性矩阵,这些侧链按C-alpha-C-beta键叠合对齐,初步进行聚类,生成100组不同的类(cluster),见Figure 1。
Figure 1. 氨基酸侧链生成100个多样类的3D叠合结果,片段之间按I-C键互相叠合对齐。
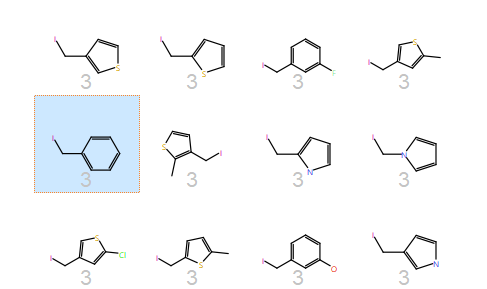
在给出的结果里,可以观察到同一个类的成员有非常好的关系。例如在类2(Cluster 2,见Figure 2)中,与酪氨酸(高亮的那个)同在一个类中的除了各种含有酚的侧链之外,还包含了具有供体-受体模式的吲唑侧链。
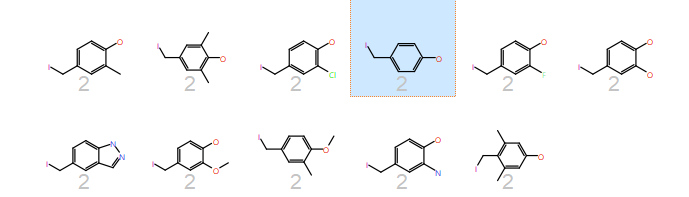
Figure 2. 与酪氨酸相关的类2
除色氨酸吲哚外,还有其他取代吲哚、pyropyridine和苯并呋喃等衍生物。与亮氨酸的异丁基侧链同组的有许多环状类似物(见Figure 3),我预计它们会引起许多2D相似性的问题。
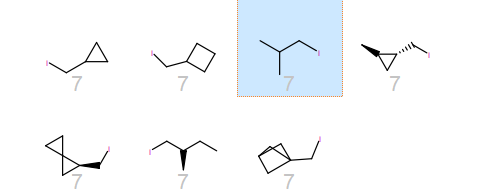
Figure 3. 与亮氨酸相关的类
令人感兴趣的是,二氢吲哚(indoline)与高苯丙氨酸(homo-phenylalanine)在同一个类里(见Figure 4)。
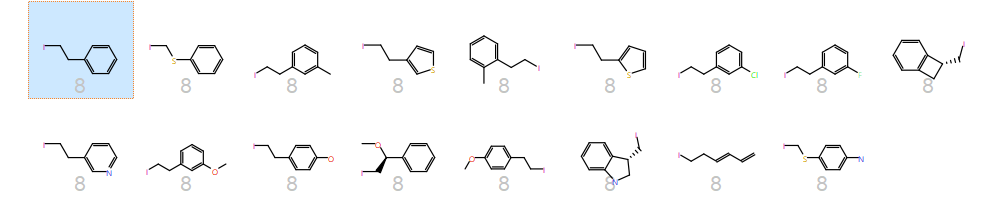
Figure 4. 与高苯丙氨酸相关的类
含有苯丙氨酸侧链的化合物突出了PickR与其它方法的主要区别:R基团用3D静电相关的性质聚类。因此,与苯丙氨酸侧链同在一组的化合物,你可以看到有有噻吩和吡咯(见Figure 5),但很少有其他芳香族化合物。吡啶和嘧啶各自归属到去自己的类里,这是因为使用静电,它们与普通的苯环是相当不同的。
Figure 5. 苯丙氨酸类侧链的3D与2D视图