摘要:1H-吲唑-3-甲酰胺类化合物1是全新的ATP-竞争性GSK-3β抑制剂,它具有良好的激酶选择性、PK特性和药效,但其hERG抑制作用也很强。本文用Spark中的“docking”功能来快速识别化合物1双氟苯基部分的可替代R-基团,用于在靶标蛋白的未被占据的口袋中进行配体生长或片段生长,以便发现那些现有起始化合物或参比分子尚未与蛋白活性位点发生相互作用的新化合物。更具体地说,我们将寻找与GSK-3 β 活性部位直接相互作用以增强GSK-3β活性的替代R-基团,以实现减少亲脂性、降低hERG抑制作用。
原文:https://www.cresset-group.com/science/resources/docking-Spark
编译:肖高铿/2021-01/23
在本算例中,我们使用Cresset生物等排体替换与骨架跃迁工具SPARK的对接功能,直接从GSK-3β活性位点挑选配体蛋白相互作用可供选择的R-基团替换,并进行优先排序。
前言
GSK-3β是一种丝氨酸/苏氨酸激酶,参与多种细胞过程;特别是,它在成人大脑中的广泛表达表明它在神经元信号通路中起着基础性的作用。 GSK-3β也与情绪障碍有关,如双相情感障碍、抑郁和精神分裂症,人们对发现新的GSK-3β抑制剂作为一种潜在的治疗方法越来越感兴趣。
1H-吲唑-3-甲酰胺类化合物(1H-indazole-3-carboxamides)是最近发现的全新的ATP-竞争性GSK-3β抑制剂[1,2]。Cmpd 1(图1-左和表1)是一种有效的酶和细胞GSK-3β抑制剂,也显示出良好的激酶选择性,在躁狂症动物模型中PK特性和药效令人鼓舞,但hERG抑制作用也很强。
为解决hERG的抑制作用,对Cmpd 1进行了先导化合物优化[2]包括用氧杂环烷基替换(2-甲氧基乙基)-4-甲基哌啶部分以降低化合物的碱性,以及用极性更大的基团替换二氟苯基片段以降低亲脂性。这一努力发现了Cmpd 14(图1-右与表1),其GSK-3β活性得以提高,并且显著降低了hERG的抑制作用。
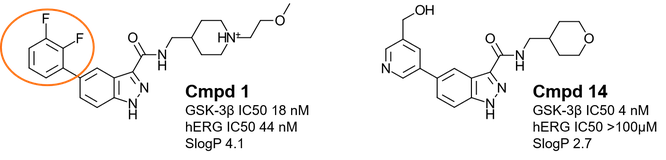
图1. 左:Cmpd 1是一种强效、选择性的GSK3β抑制剂,但却受到hERG不利的影响;右:优化后的Cmpd 14的GSK3β活性得以提高,并且降低了hERG的不利作用。
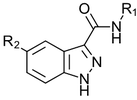
Cmpd 1衍生物的SAR数据如下表1所示。
表1. 1H-吲唑-3-甲酰胺母核R1与R2位的SAR探索[1,3]
本文我们用Spark中的“docking”功能来快速识别双氟苯基部分的可替代R-基团(图1-左中的橙色圆圈)。该方法可用于在靶标蛋白的未被占据的口袋中进行配体生长或片段生长,以便发现那些现有起始化合物或参比分子尚未与蛋白活性位点发生相互作用的新化合物。更具体地说,我们将寻找与GSK-3 β 活性部位直接相互作用以增强GSK-3β活性的替代R-基团,以及减少亲脂性并降低hERG抑制作用的替代R-基团。
蛋白-配体复合物
在Spark中使用对接方法时,为了加氢、优化内部氢键网络、消除原子碰撞、并分配最佳的质子化状态,强烈建议从精确准备的蛋白结构开始进行实验。使用Cresset基于结构的设计平台Flare来准备Cmpd 1与GSK-3β结合的蛋白-配体复合物结构(PDB:6TCU)。
对蛋白质-配体接触的目视检查(图2)表明,吲唑甲酰胺骨架与GSK3的铰链结构域Asp133的主链羰基、Val135的主链羰基以及NH发生三个氢键相互作用。哌啶部分完全暴露于溶剂,而二氟苯环靠近催化位点Lys85。
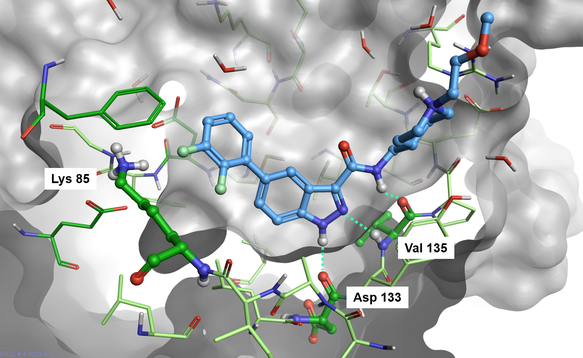
图2. Cmpd 1结合到GSK3β活性位点
SPARK docking方法
首先,SPARK将起始分子中你希望被替换的部分删除掉(图3,A-B)。然后将新片段连接到在上一步被截短的起始分子上,并在Cresset XED力场计算的蛋白静电势引导下以合理的取向放置到蛋白的结合位点里(图3,C)。 然后,用Lead Finder的“score only”对接模式来优化新分子的pose(图3,D)。最后,用Lead Finder的“Rank score”打分函数对最佳结果进行优先级排序,经优化后打分函数可以正确重现已知配体的结合模式,并在Spark结果表单中按对接打分值升序进行排序(打分越负更好)。

图3. Spark的Docking工作流(PDB code:6TCU) A)在起始分子中选择要被替换部分;B)删除所选择的片段; C)将新的片段接到在上一步被截短的起始分子上,并以有意义的取向放置好;D)使用 LeadFinder优化新结果分子的pose
“Scaffold Hopping or R-group Replacement”向导(见图4-左)提供了易于遵循的界面进行实验设置。将事先用Flare准备好的6TCU蛋白-配体复合物以using the input protonation state模式导入到Spark中,选择Cmpd 1作为起始分子,并删除所有结晶水分子。选择二氟苯基部分作为起始分子中要被替换的区域(见图4-右)。
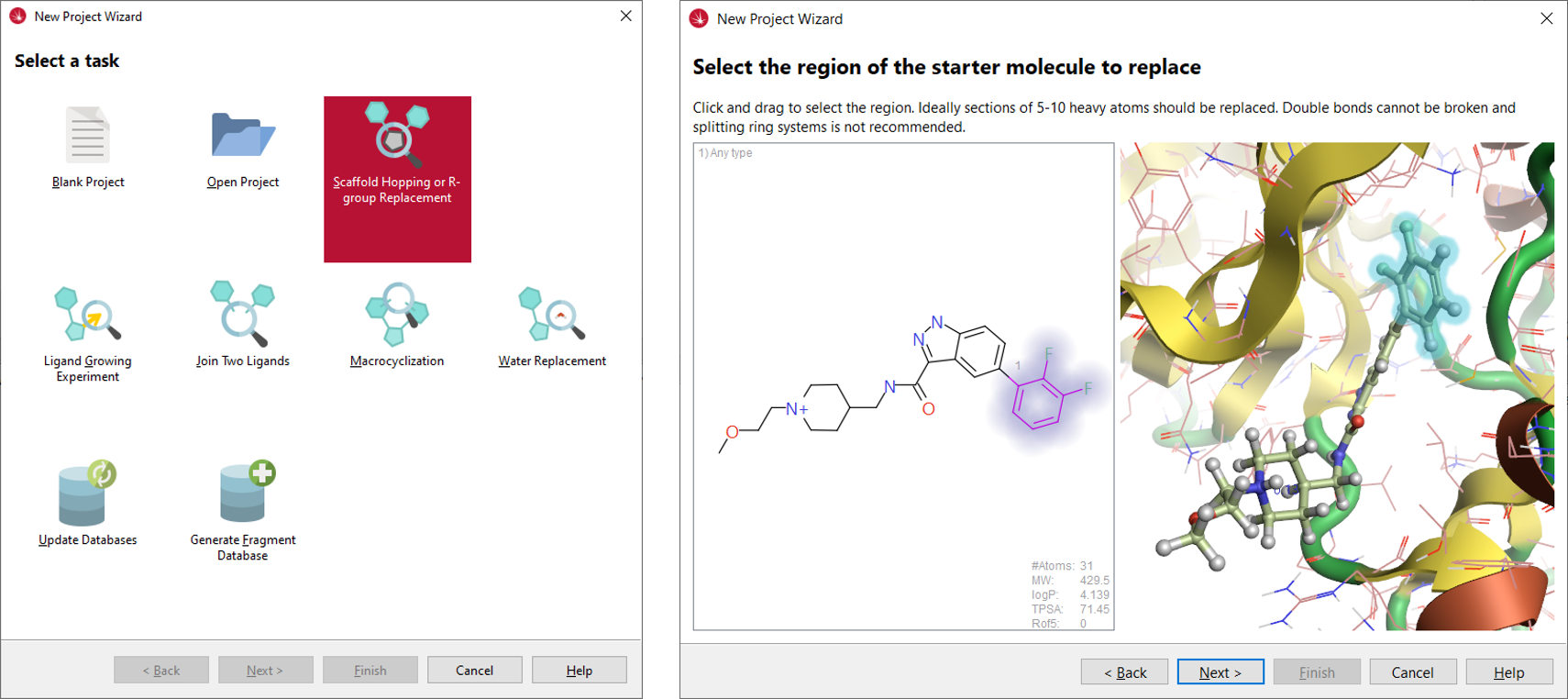
图4. Spark的项目向导(左)以及起始分子替换区域的选择(右)
退出向导后,Spark计算方法设置为“Docking”。由于这种方法比传统的基于配体相似性的Spark搜索方法对计算量要求更高,因此建议设置适当的物理化学过滤器来限制搜索空间并减少计算时间、将实验重点放在所需的结果上。在本实验中,以起始分子(MW 429.5,SlogP 4.1)的性质作为参考,将潜在结果的分子量设定为低于466,并将SlogP设定为低于3.5。并使用SMARTS过滤器将取代苯基和2-羟基吡啶从潜在结果列表中过滤掉(图5)。
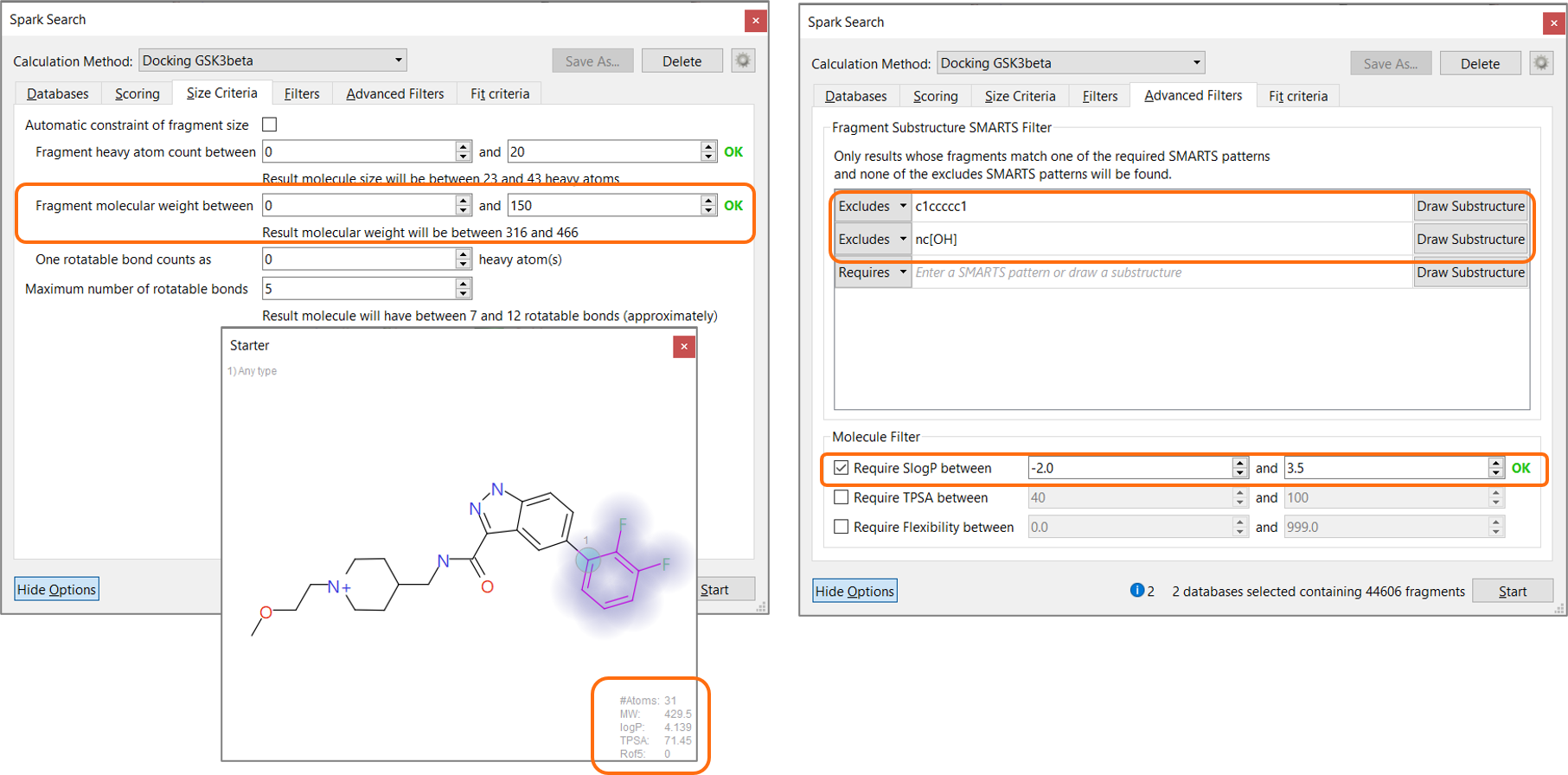
图5. 在SPARK搜索时设置合适的理化性质过滤器以便将实验聚焦于所需的结果上并减少计算时间
为了重现原始文献中使用的化学方法[2], 本次实验仅对来自eMolecules砌块[4]的4.5K芳香族硼酸和40K卤代芳香族数据库进行虚拟筛选。
结果
Spark的对接实验中正确地将吡啶3-基取代基识别为得分最高的结果(图6,左)之一,包括与表1中报道的相同R-基团或相似的R-基团(图6-中间)。在得分最高的Spark结果中,我们还发现了分子量和亲脂性受限的各种双环杂环化合物,它们可作为吡啶-3-取代的替代品以作为先导化合物系列进行进一步的探索。图6(右)展示了一些化合物示例。
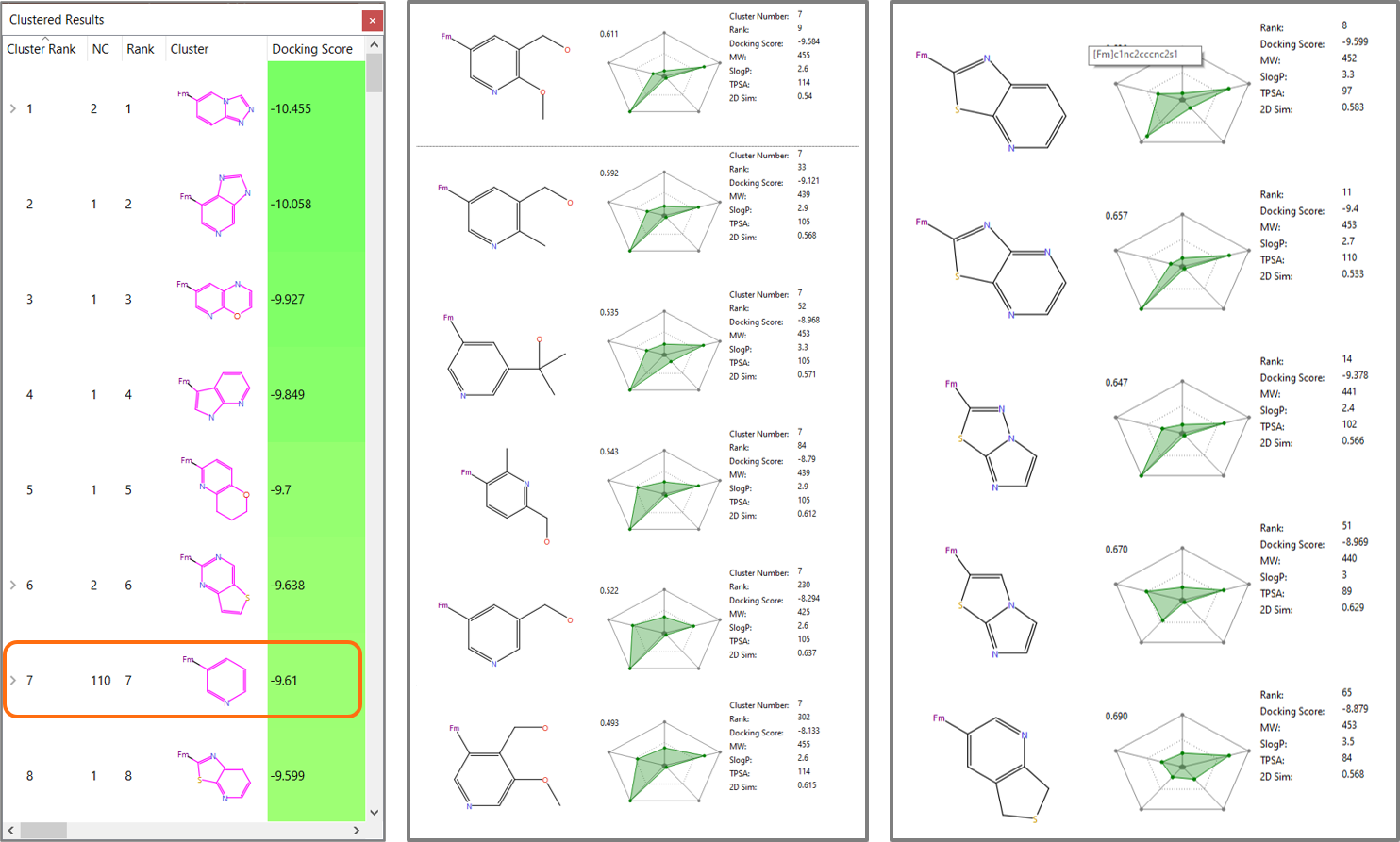
图6. 左:吡啶-3-取代基[2]在Spark结果中排名第7; 中:吡啶-3-取代基各个成员的与表1的结构相似;右:SPARK发现的双环杂环其分子量与亲脂性受到限制。
有趣的是,Spark给出的吡啶-3-衍生物的结合模式涉及与Lys85的氢键相互作用,有些还与Phe67发生的芳香-芳香相互作用,这与化合物OH84的晶体学结合模式一致( PDB:6Y9R,图7)。 然而,并不令人惊讶的是,对接方法倾向于取代的(例如羟基)3-吡啶,从而与活性位点里的残基(例如Asp 200)发生额外的氢键相互作用。
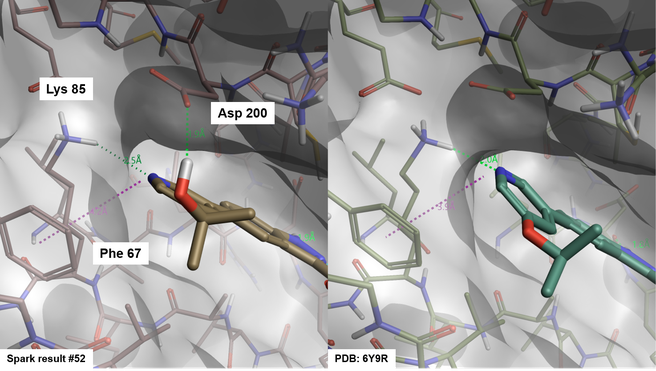
图7. SPARK结果#52(左边)与PDB 6Y9R的化合物OH8(右边)的结合模式
结论
对接功能使Spark可以直接从蛋白活性位点中找到配体-蛋白相互作用相符的片段。用这种方法可以使配体和/或片段往靶蛋白未占据口袋方向生长,因此找到的新化合物与蛋白活性位点的相互作用模式也是现有的起始分子或参考分子未曾有过的。
文献
- Ombrato, R.; Cazzolla, N.; Mancini, F.; Mangano, G. Structure-Based Discovery of 1 H -Indazole-3-Carboxamides as a Novel Structural Class of Human GSK-3 Inhibitors. J. Chem. Inf. Model. 2015, 55 (12), 2540–2551. https://doi.org/10.1021/acs.jcim.5b00486.
- Prati, F.; Buonfiglio, R.; Furlotti, G.; Cavarischia, C.; Mangano, G.; Picollo, R.; Oggianu, L.; di Matteo, A.; Olivieri, S.; Bovi, G.; Porceddu, P. F.; Reggiani, A.; Garrone, B.; Di Giorgio, F. P.; Ombrato, R. Optimization of Indazole-Based GSK-3 Inhibitors with Mitigated HERG Issue and In Vivo Activity in a Mood Disorder Model. ACS Med. Chem. Lett. 2020, 11 (5), 825–831. https://doi.org/10.1021/acsmedchemlett.9b00633.
- Buonfiglio, R.; Prati, F.; Bischetti, M.; Cavarischia, C.; Furlotti, G.; Ombrato, R. Discovery of Novel Imidazopyridine GSK-3β Inhibitors Supported by Computational Approaches. Molecules 2020, 25, 2163. https://doi.org/10.3390/molecules25092163
- https://www.emolecules.com/info/products-building-blocks.html
想在项目中使用Spark吗?
SPARK现有用户会收到一封关于如何升级到V10.6的电子邮件;若您当前还不是SPARK用户的话,可以联系我们获取试用版。



