摘要:本文介绍了如何在 Cresset 药物设计平台Flare™ 中利用梯度提升(Gradient Boosting)机器学习方法构建稳健的定量构效关系(QSAR)模型,以应对分子描述符间的多重共线性问题。首先概述了 QSAR 建模中常用的分子描述符类型(如理化性质、2D 指纹、拓扑指数和 3D 场描述符),随后重点阐述了在缺乏生物活性 3D 构象信息时,如何基于 2D 描述符构建机器学习 QSAR 模型。针对传统 QSAR 模型易因高维或相关描述符导致过拟合的问题,本文强调梯度提升模型凭借其决策树架构,能自动识别关键特征、抑制冗余信息,从而提升模型泛化能力。通过 hERG 心脏毒性预测案例,展示了该方法在 ToxTree 数据集上的优异表现:模型 R² \(>\) 0.5,训练与测试性能差距小,表明无明显过拟合。此外,本文还介绍了 Flare Python API 支持的两种描述符筛选策略——基于方差与相关性的简单剔除法和更智能的递归特征消除(RFE)法,为用户提供了灵活的建模工具。研究表明,结合梯度提升算法与合理的特征工程,可显著增强 QSAR 模型在药物发现中的预测可靠性。
Boyli Ghosh. 2025-08-27. Enhancing QSAR Models: tackling descriptor intercorrelation with robust Gradient Boosting Machine Learning models in Flare™.
在典型的药物发现场景中,我们通常需要处理大量化合物及其相关的理化性质和活性数据,这些数据往往来源于文献和/或内部实验。为此,我们常构建定量构效关系(QSAR)模型——这是化学信息学中的一项关键技术,能够基于分子描述符预测化合物的性质或活性。这使得我们能够在超大规模的化学空间中实现超快速的性质/活性预测,适用于有结构信息支持和无结构信息支持的分子设计项目。
QSAR 建模中的特征表示
构建有效 QSAR 模型的第一步,是将分子输入格式(例如一维 SMILES 字符串或二维/三维分子构象)转化为有意义的特征,即“描述符”(descriptors)。这些描述符包括:
- 理化性质:由分子物理特性导出的标量值,如分子量、logP、极性表面积、氢键性质(供体/受体)等
- 二维指纹(2D fingerprints):以比特向量形式编码分子子结构,反映特定子结构、路径或片段的存在与否;
- 拓扑与连接性指数:基于分子二维图表示计算得出,用于刻画分子大小、形状、分支程度及原子连接方式(例如 Kier-Hall 连接性指数);
- Cresset XED 三维场描述符:分子静电场与形状场中的局部极值点,从蛋白质“视角”模拟配体的形状与静电特性
QSAR 建模通过挖掘分子描述符(如三维描述符、拓扑指数、二维指纹、理化性质等)与目标终点之间的线性或非线性关系,构建预测模型,从而支持化合物的虚拟筛选与优先级排序。
Flare中的2D-ML-QSAR建模
Flare 是一个强大的平台,支持构建3D和2D QSAR 模型。利用 Cresset 的 XED 力场生成的静电与体积描述符,可构建 Field QSAR1 和基于机器学习 QSAR 模型,二者在预测生物活性方面均表现出色。然而,构建3D-QSAR 模型需依赖一个已知的生物活性3D构象作为参比,以便对所有化合物进行叠合——这是生成3D场描述符和“去噪”QSAR 模型的关键步骤。当参比配体的生物活性3D构象未知时,可转而采用基于2D描述符的机器学习 QSAR 模型。这些2D描述符既可在 Flare 中实时隐式计算,也可从 RDKit 或其他外部性质服务中导入。
QSAR 建模面临的一大挑战是避免过拟合。使用过多描述符的模型可能在训练集上表现优异,却难以准确预测训练集之外的新化合物。为此,可采用对共线性(collinearity)和多重共线性(multi-collinearity)具有内在鲁棒性的模型,如梯度提升(Gradient Boosting)机器学习模型;或者,需谨慎选用相关描述符。
Flare 中的梯度提升模型(Gradient Boosting Model)
Flare 进一步扩展了其 QSAR 能力,引入了梯度提升机器学习模型2。该类模型能够捕捉分子描述符与活性之间复杂的非线性关系,自动优先考虑重要描述符,同时抑制对无关描述符的过拟合。得益于其基于决策树的架构,梯度提升模型天然具备对共线性与多重共线性的鲁棒性——它会优先选择信息量大的分裂节点,并自动降低冗余描述符的权重。因此,该模型非常适合处理高维描述符集,无需借助递归特征消除(RFE)等方法进行预筛选即可实现高效建模。
案例研究:hERG 离子通道活性预测
我们以构建基于描述符的梯度提升机器学习 QSAR 模型为例,用于预测化合物因抑制 hERG 通道而引发的心脏毒性——这在药物研发中评估心脏安全性至关重要。本实验采用 ToxTree hERG 数据集3进行模型训练与评估。该数据集包含 8,877 个以 SMILES 字符串形式存储的化合物,涵盖多样化的化学结构,每个化合物均关联一个 hERG pIC50 值。数据集经标准化处理(转换为规范 SMILES),并使用 RDKit 计算了 208 个理化、拓扑及连接性描述符。
所有 208 个 RDKit 描述符均经过标准化缩放,并通过 Python 脚本4 生成相关性矩阵,以初步评估描述符空间中多重共线性的程度(见图1)。在图1中,每个单元格表示两个描述符之间的 Pearson 相关性系数,颜色深浅反映相关性强弱:红色区域代表高度相关,提示可能存在冗余,易导致模型过拟合;绿色/浅蓝色区域则表明描述符彼此独立,提供互补信息。在本案例中,强相关性的描述符对并不普遍,但仍有个别描述符值得进一步监控。若在最终建模过程中出现过拟合迹象,剔除部分高度相关的描述符或将有助于提升模型泛化能力。
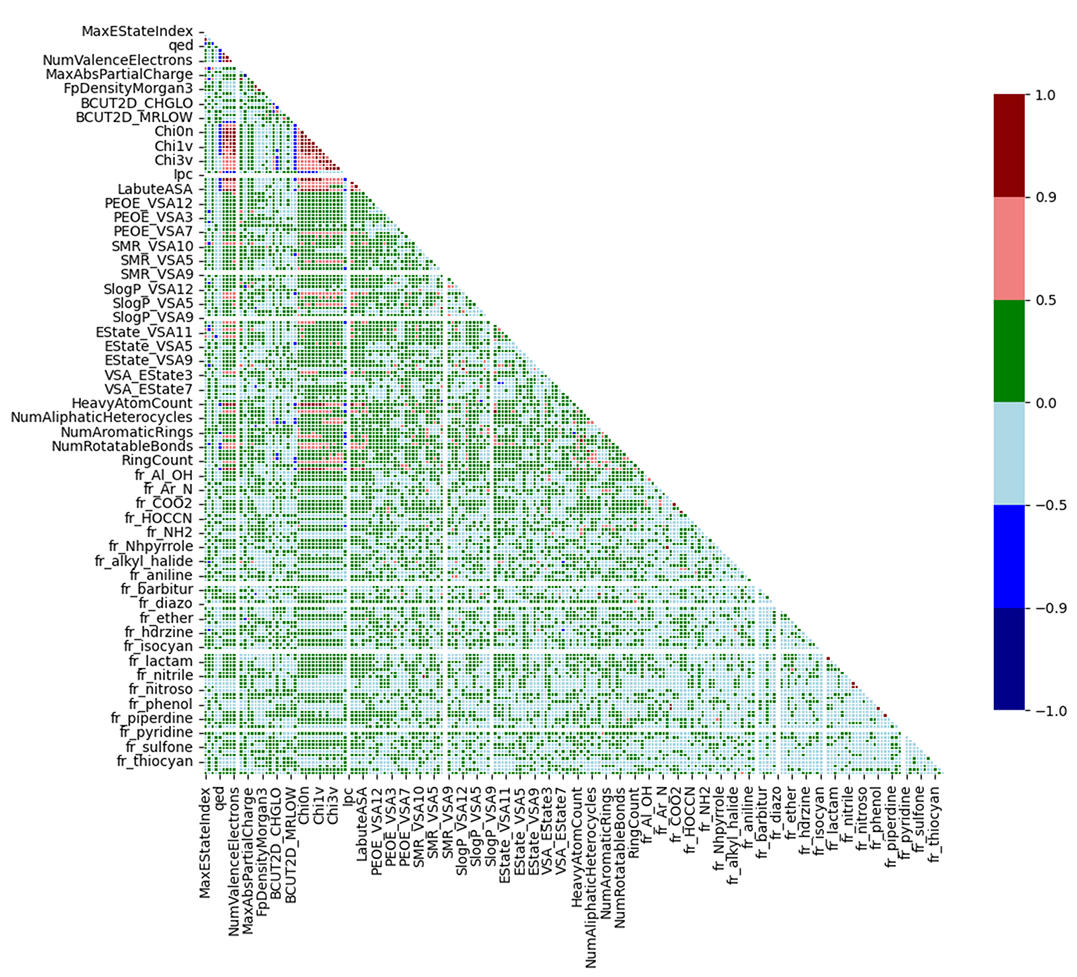
图1. ToxTree hERG 数据集2的特征相关性矩阵,为清晰起见仅展示部分描述符(208个RDKit描述符中的部分未显示)。颜色标度表示描述符之间的相关程度:深红色(1.0):强正相关(描述符高度相似);深蓝色(-1.0):强负相关(一个描述符增大时,另一个减小);绿色(≈0.0):弱相关或无相关性(描述符相互独立)。
作为评估分子描述符与 hERG pIC50 之间关系本质的诊断步骤,我们首先在完整数据集上分别训练了一个五折交叉验证的线性回归模型和一个梯度提升(Gradient Boosting, GB)模型。此步骤旨在判断数据底层结构主要是线性的还是非线性的,或是否存在多重共线性问题(即两个或多个预测变量高度相关,导致难以区分各自对因变量的独立影响)。结果显示,GB 模型显著优于线性模型,其均方根误差(RMSE)明显更低,表明描述符与活性之间的关系具有非线性特征,或受到多重共线性的影响。基于这一认识,我们在 Flare V10 中采用梯度提升方法推进完整的建模流程,包括超参数优化与模型验证(见图2)。测试集的决定系数(R2)大于 0.5,表明该模型具备良好的预测能力。交叉验证训练集与测试集 R2 的差值(R2 delta)为 0.041,同时 RMSE 差值(RMSE delta)为 6.59%,进一步说明模型未出现对训练数据的过拟合。
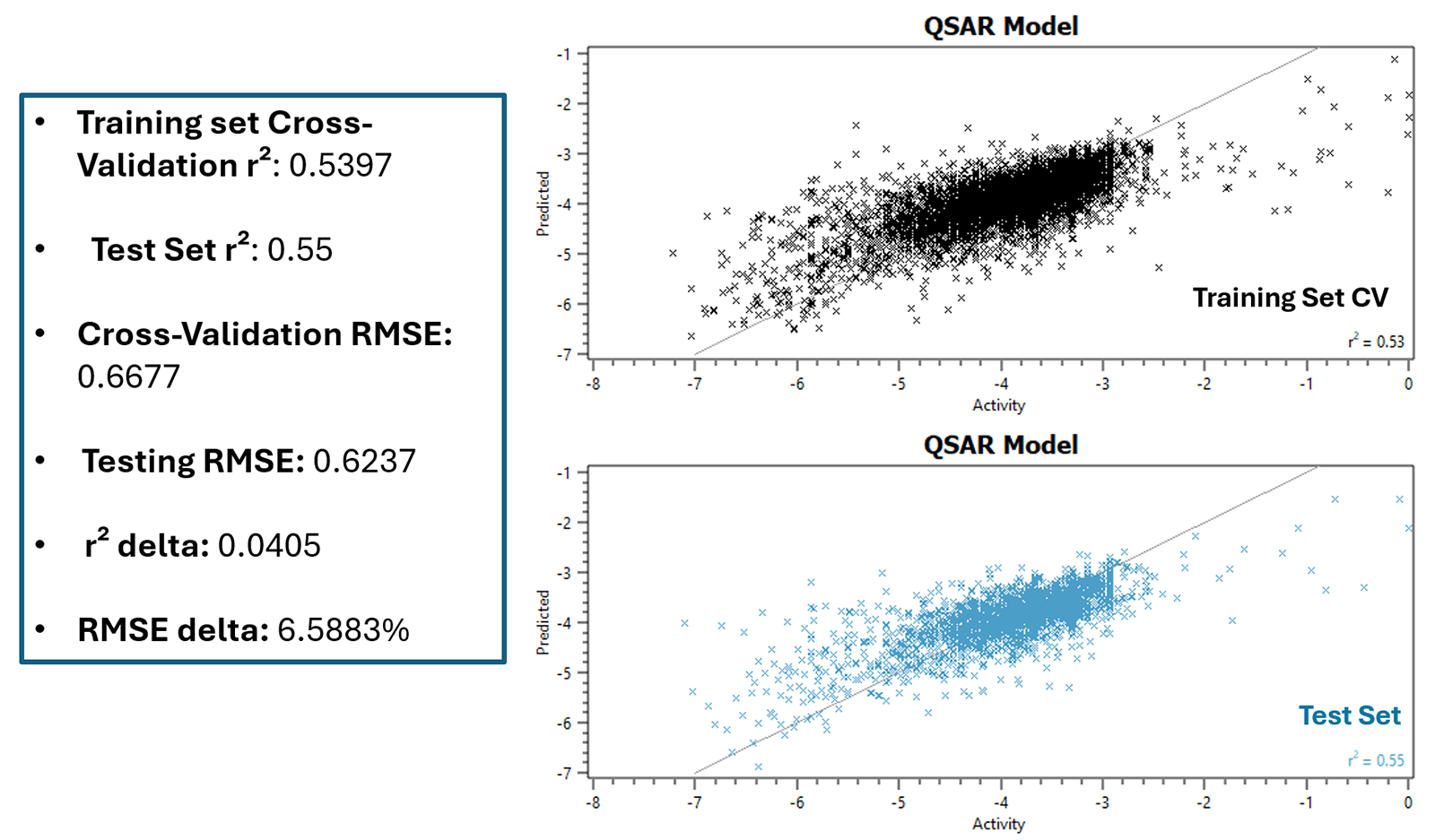
图2. 梯度提升模型性能(左);训练集交叉验证(CV)与测试集的预测活性 vs. 实验活性散点图(右)。
利用 Flare Python API 避免过拟合并进行描述符筛选
避免过拟合的一种简单替代方法是剔除以下几类描述符:
- 含缺失值的描述符(Flare 中已自动处理);
- 在整个数据集中取值恒定的描述符;
- 具有多重共线性的描述符,即彼此高度相关的描述符。
尽管该方法直观且易于实现,但仅通过剔除低方差或高相关性的描述符存在局限性——最显著的问题是忽略了这些描述符与目标性质之间可能存在的因果关系,从而可能无意中丢弃了在与其他描述符组合时具有重要预测价值的特征。另一种虽更复杂但更优的策略是递归特征消除(Recursive Feature Elimination, RFE)。RFE 将描述符筛选过程整合到模型训练中,提供了一种更智能的剔除方式:它通过迭代方式,依据各描述符对模型性能的影响,逐步移除重要性最低的描述符,最终仅保留那些在完整描述符空间中真正具有预测能力的特征。这种有监督的筛选方法在降低过拟合风险的同时,有效保留了最具信息量的特征。
Cresset 通过 Flare Python API 提供了两个 Python 脚本,支持用户采用上述两种策略:
- 基于方差和多重共线性阈值的简单描述符剔除;
- 基于 RFE 的描述符筛选。
结论
有效管理描述符间的相关性和方差,对于构建稳健的 QSAR 模型至关重要。Flare V10 提供的先进功能——包括梯度提升 QSAR 机器学习模型,以及基于 Flare Python API 开发的描述符筛选脚本——为提升药物发现中模型的性能与可靠性提供了强有力的工具。
作者介绍

Boyli Ghosh 应用科学家
Boyli Ghosh 于印度科学培育协会(Indian Association for the Cultivation of Science)取得计算化学博士学位。随后,她先后在班加罗尔的Prescience Insilico与韩国的PharmCadd两家计算软件开发公司担任研究科学家,累计工作两年半。Boyli Ghosh 在小分子体系的多种量子力学方法应用、酶体系的QM/MM计算,以及基于配体的药物设计所涉及的各类计算机模拟方法方面拥有丰富经验。目前,作为Cresset公司的应用科学家,她主要负责客户培训与技术支持,协助用户通过Cresset基于配体与基于结构的解决方案达成科研目标。
参考文献
- Flare™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/software/flare/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation J. Chem. Inf. Model. 2006, 46 (2), 665-676
- (a) A. Knoll, A. Natekin, Gradient Boosting Machines, a Tutorial. Front. Neurorobotics 2013, 7. (b) A. Ferrario, R. Hämmerli, On Boosting: Theory and Applications. Social Science Research Network
- Arab, I.; Barakat, K. ToxTree: descriptor-based machine learning models for both hERG and Nav1.5 cardiotoxicity liability predictions. arXiv, 2021, arXiv:2112.13467, ver. 1. DOI: 10.48550/arXiv:2112.13467v1
- Contact support@cresset-group.com for access to the Python script



