摘要:本文用一个包含76个SARS-COV-2 Mpro抑制剂的数据集为例,演示了Cresset Discovery团队如何利用他们专业的计算知识与Flare强大的机器学习与Field QSAR建模能力相结合,构建稳健的QSAR模型来理性理解SAR,并用来指导新化合物的设计、并对新设计的化合物进行优先级排序。
前言
病毒主蛋白酶Mpro是严重急性呼吸综合征冠状病毒-2(SARS-CoV-2)复制的关键酶,该病毒是新冠肺炎全球大流行的原因。除了既定的新冠肺炎疫苗接种计划外,抗病毒药物被视为控制未来不可避免的冠状病毒流行的关键。由于其关键作用,Mpro作为新型治疗药的潜在靶点受到了广泛关注1-6。
开发稳健、预测性的抗SARS-CoV-2 Mpro活性的定量结构活性关系(QSAR)模型,可以阐明活性、为新分子设计提供信息。Cresset Discovery CRO团队使用一个包含76个已知实验活性、常见结合模式的化合物数据集,在分子建模解决方案FlareTM中构建预测性机器学习(ML)和Field 3D-QSAR方法模型7。
所有模型的统计性能与实验结果一致,所有模型的描述性和预测能力相当。此外,Field 3D-QSAR方法突出了驱动活性的分子区域。
方法
数据集
在本案例研究中,将一个具有76个不同化学型和活性范围(pIC50:4.00-7.74)均匀分布的非共价抑制剂数据集按26%的比例划分为训练集(56个分子)和测试集(20个分子)。
2D-QSAR
最初选择的2D描述符(一些从RDKit8导入)通过线性Pearson相关矩阵进行交叉相关,以减少冗余描述符。使用6个物理化学描述符(MW,TPSA,#RB,NumHAcceptors,NumHDonors和RingCount)以及指纹(RDKit,Morgan和MACCS Keys),用监督机器学习(ML)方法包括支持向量机(SVM)9,高斯过程回归(GPR)10,随机森林(RF)11和多层感知器(MLP)12来建立2D-QSAR回归模型。
3D-QSAR
构建3D-QSAR模型对构象搜索和分子叠合非常敏感。Flare用于进行高质量的叠合,特别是基于最大公共子结构(MCS)算法的高质量叠合,结合Cresset Discovery的专业知识来解释结果,产生了具有低噪音、有意义的分子叠合结果(图1)。这些化合物通过MCS叠合到PDB 7L131、7L141、7QBB5和8SXR6的共结晶配体上,这些共晶配体被用作参比分子(加权平均贡献),并使用7L13蛋白作为“软”硬度的排除体积。使用的构象搜索参数是标准的“very accurate and slow”,设置能量窗口为2.5 kcal/mol。
3D-QSAR回归模型使用Field 3D-QSAR方法(标准的“Normal with Y scrambles”方案)和机器学习方法进行开发7,其中机器学习方法包括k-Nearest Neighbors(kNN)、SVM、GPR、RF、MLP和Consensus。这些方法使用Cresset XED力场获得的场点直接确定的探针位置来对训练集中每个分子的静电势和体积/形状进行采样,然后将其用作QSAR模型的描述符。例外的是kNN方法,它使用Cresset XED力场作为相似性度量7。
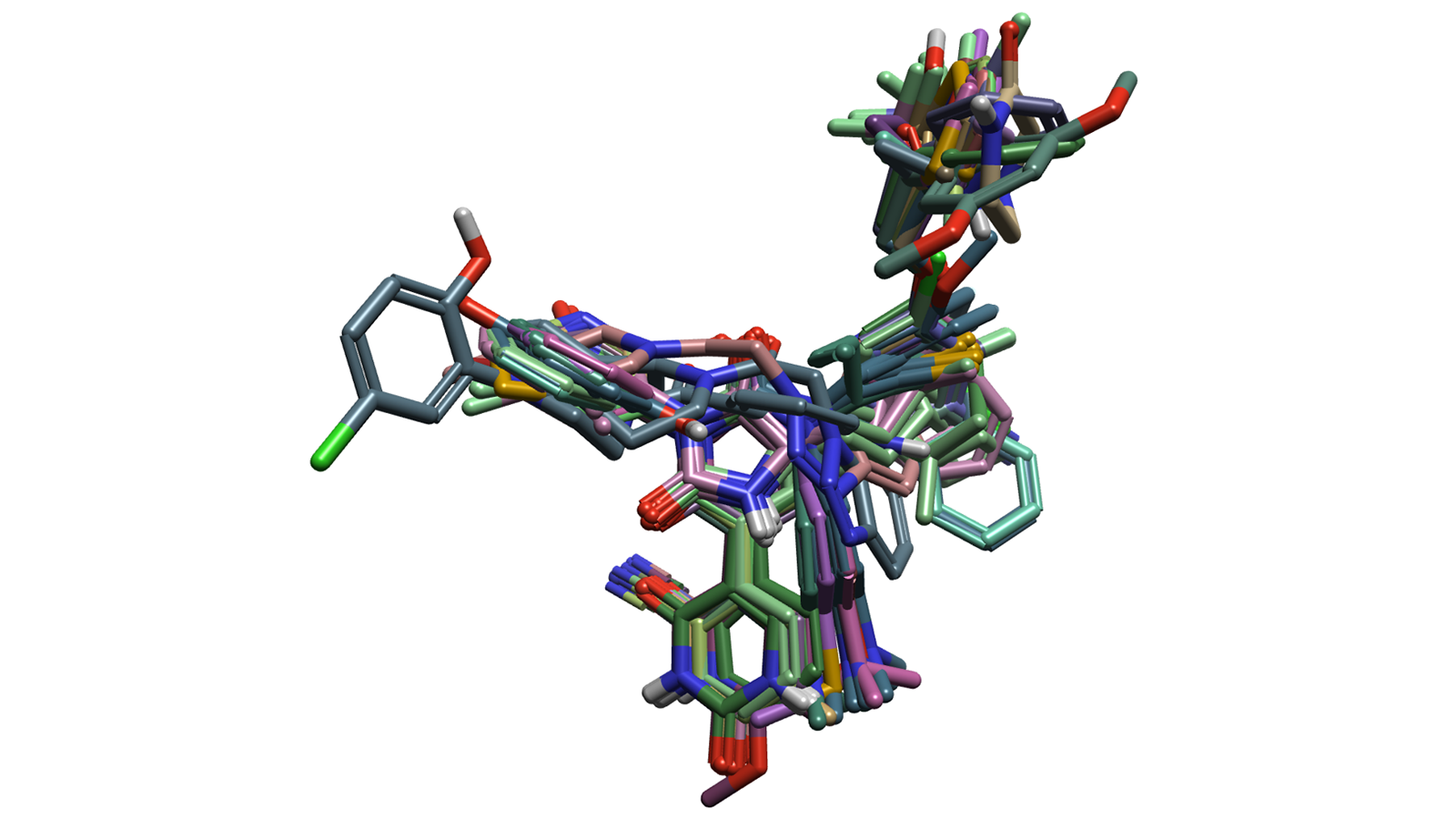
图1. 数据集化合物的叠合结果
结果
统计分析
表1. 不同QSAR模型预测值与实验值的统计数据比较
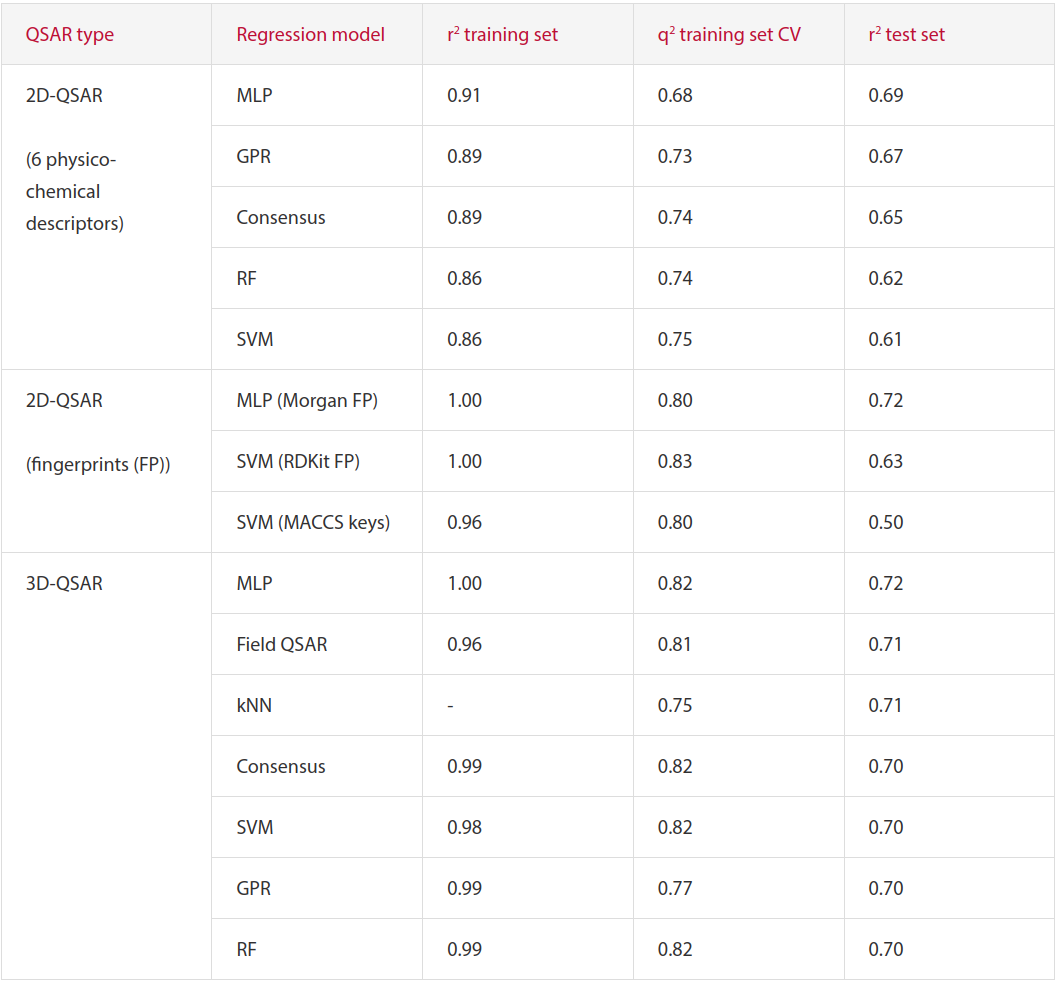
表1显示了各种模型的性能,根据所有评估的QSAR方法的预测统计数据排名。图2显示了Morgan FP MLP 2D-QSAR和MLP 3D-QSAR模型的实验与预测活性图,这两个模型具有最高的总体测试集r2,为0.72。除了MACCS-keys SVM 2D QSAR之外,所有测试集r2确定的模型置信度都很高。因此,这些模型中的任何一个都可以准确地预测新化合物的活性。2D和3D模型之间良好的一致性表明,该数据集中的化合物是通过类似的机制起作用的。还可以看出来RDKit 2D描述符和指纹可作为Cresset 3D描述符的良好替代方案来构建预测性ML模型。然而,与Cresset分子场点相反,这些模型不能进一步提供建模的潜在关键区域的见解以改进识别、结合和随后测试分子的活性。
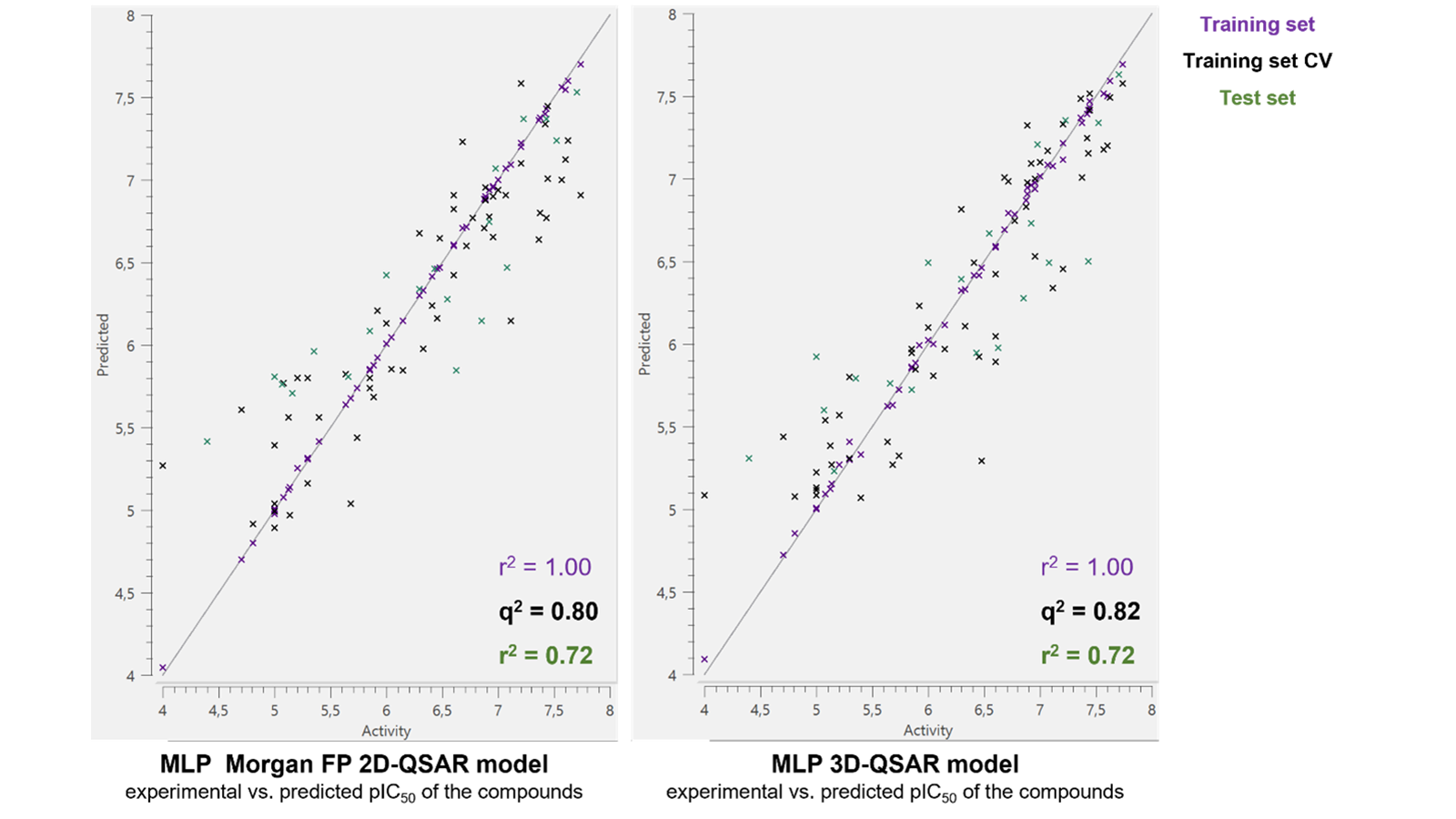
图2. MLP Morgan FP 2D-QSAR(左)和3D-QSAR(右)模型 – 训练集(紫色)、训练集交叉验证(CV)(黑色)和测试集(绿色)中化合物的实验活性和预测活性。
模型可视化与结果解释
独特的Cresset Field 3D-QSAR方法比ML方法更有优势,因为其模型系数的可视化检查可用来确定模型预测对活性有强烈影响的区域。图3显示了叠合在活性最强分子37(pIC50 = 7.74)上的静电和立体模型系数。在母核环的酰胺羰基和吡啶单元的氮原子上观察到对活性有利的负静电系数区域,这意味着这些区域的正电荷较少,可以提高活性。此外,大绿色点指出了2-氯苄基部分附近具有对活性有利的立体系数区域,这与高的立体效应方差相结合,提示了这是建模的最佳部分,增加体积可以提高活性。利用Cresset Discovery团队从模型中学习到的知识和计算化学知识,提出了在2-氯苄基单元的3位上引入氰基或甲基等取代基的可以提高活性,其活性预测值优于化合物37的预测值,pIC50预测值分别为7.70和7.60。除了立体效应之外,这些基团还优化了与酶的分子间相互作用,CN允许与Q192主链NH形成氢键,而甲基与P168和Q192侧链的烷基区域形成疏水接触,从而可以提高了抑制剂的活性。
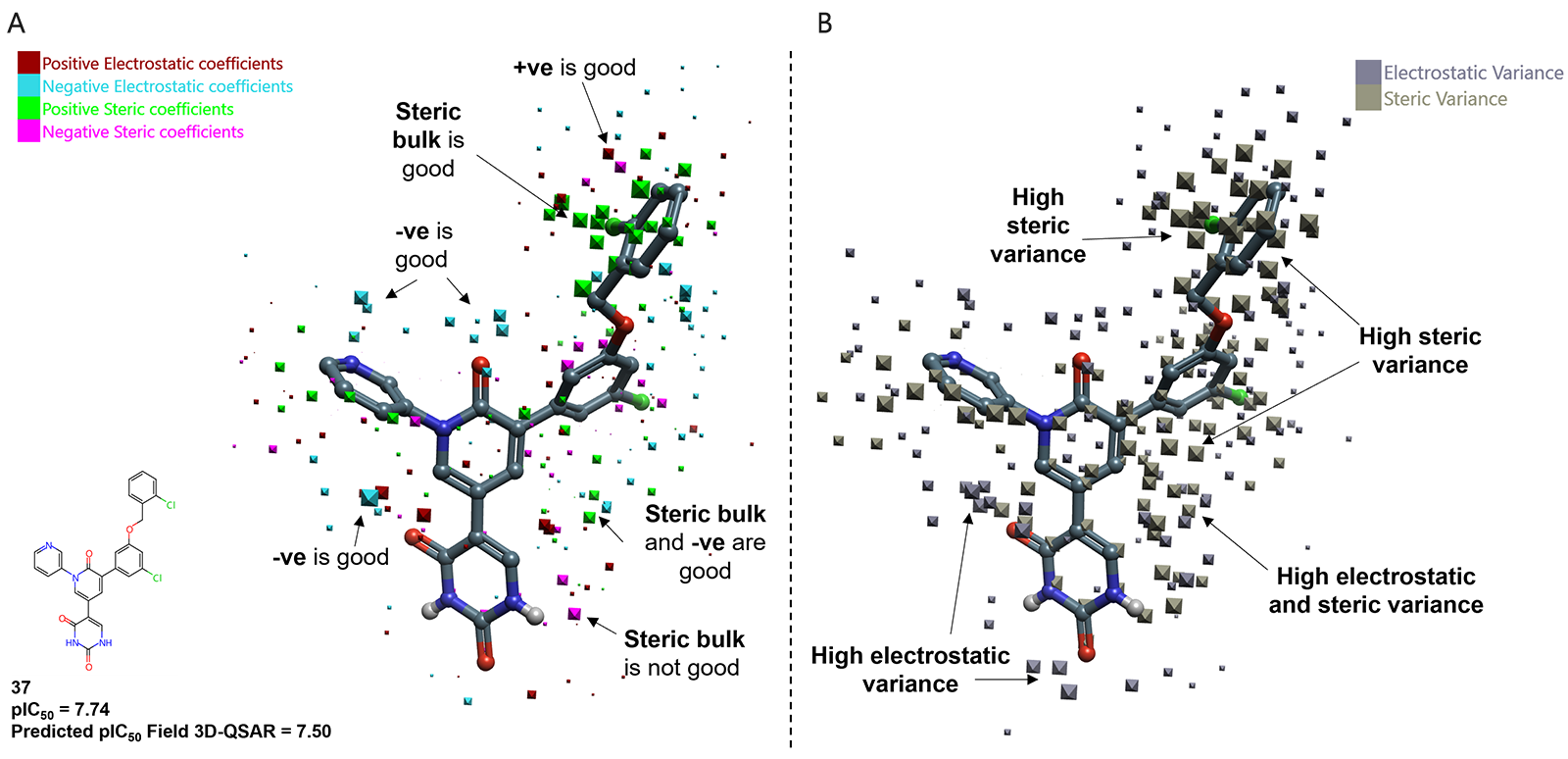
图3. SARS-CoV-2 Mpro Field QSAR模型的模型系数图。(A)静电和空间系数;(B)静电和空间方差,使用最强活性的分子37作为参照。化合物根据专利 WO2022/150584A14进行编号。
此外,通过比较化合物37与相似分子的场贡献,可以突显2-氯苄醇基团的意义(图4)。化合物8中缺少该基团会产生不利的静电贡献,导致活性降低约2.5个对数单位;而芳香环被烷基链或饱和环取代后,观察到较大的、温和的不利静电和空间贡献,导致活性降低约1个对数单位。同样,化合物28中羟基的存在会产生强烈的不利静电贡献,降低其预测的活性。
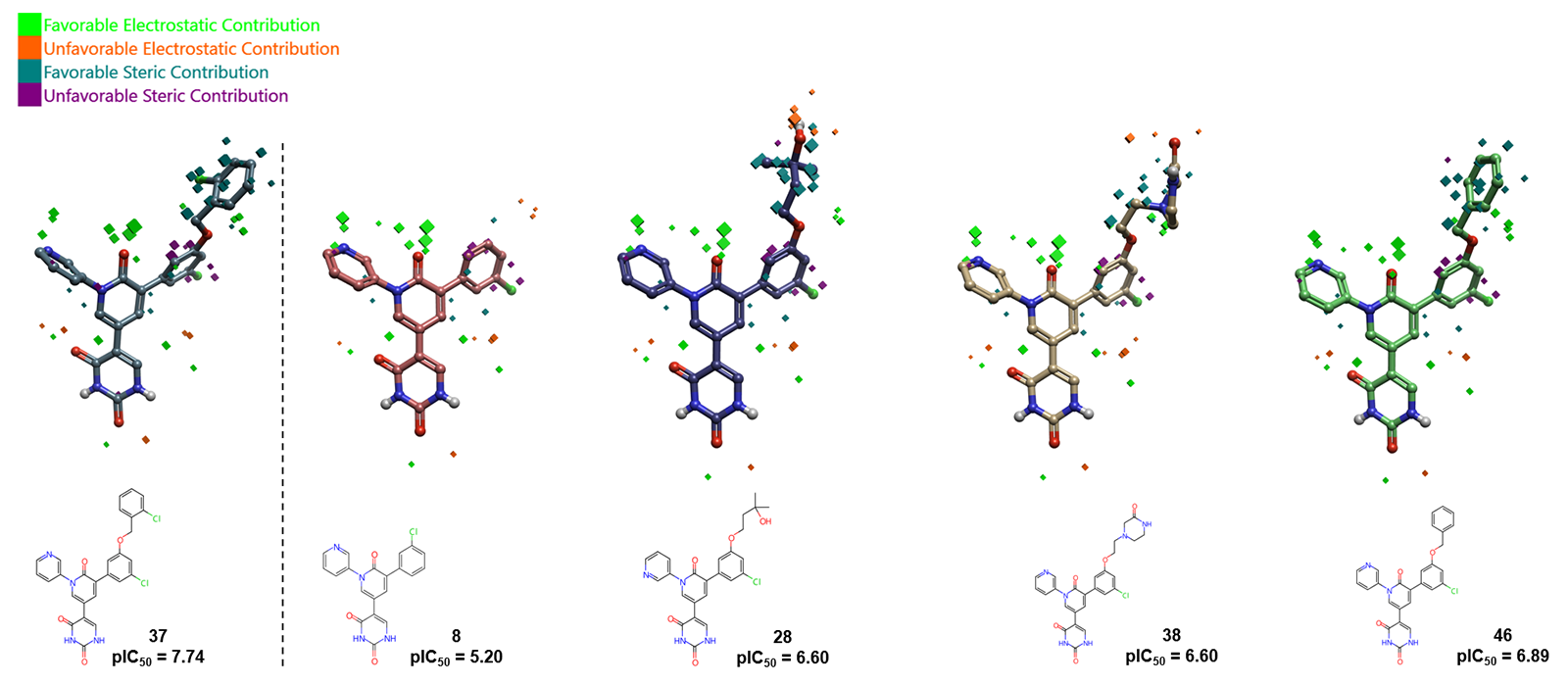
图4. SARS-CoV-2 Mpro 3D-QSAR场对化合物37、8、28、38和46预测活性值的贡献。
结论
Cresset Discovery的科学家成功地构建了稳健的2D-QSAR和3D-QSAR回归模型,以描述、预测非共价SARS-CoV-2 Mpro抑制剂的活性。我们阐明了2D-和3D-QSAR模型之间的一致性,Field 3D-QSAR模型的性能表现优于机器学习方法。团队的专业知识进一步促进了静电和空间系数分析,理性地理解抑制剂地活性,可用于指导新分子的设计并对之进行优先级排序。
Flare强大的QSAR方法,结合Cresset Discovery CRO在定量构效关系方法方面的知识,为加速创新药物的设计提供了优质的服务。请联系我们,了解Cresset Discovery CRO团队如何支持您的QSAR项目。
参考文献
- Chun-Hui Zhang, et al., ACS Cent. Sci. 2021, 7, 467–475, https://doi.org/10.1021/acscentsci.1c00039
- Chun-Hui Zhang, et al., ACS Med. Chem. Lett. 2021, 12, 1325–1332, https://doi.org/10.1021/acsmedchemlett.1c00326
- Maya G. Deshmukh, et al., Structure 2021, 29, 823–833, https://doi.org/10.1016/j.str.2021.06.002
- William L. Jorgensen, Patent WO 2022/150584 A1
- Andreas Luttens et. al., J. Am. Chem. Soc. 2022, 144, 2905–2920, https://doi.org/10.1021/jacs.1c08402
- Jimena Perez-Vargas et. al., Emerg. Microbes Infect. 2023, 12, 2246594, doi.10.1080/22221751.2023.2246594
- Flare™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/software/flare/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation J. Chem. Inf. Model. 2006, 46 (2), 665-676.
- RDKit: Open-source cheminformatics. https://www.rdkit.org
- Harris Drucker et. al., Support Vector Regression Machines, Advanced in Neural Information Processing Systems 9, 1996, 155-161.
- C. E. Rasmussen, C. K. I. Williams, Gaussian processes for machine learning 2016, The MIT Press, ISBN 026218253X.
- L. Breiman, Random Forests, Machine Learning, 2001, 45, 5-32.
- F. Rosenblatt, Principles of Neurodynamics: Perceptrons and the Theory of Brain Mechanisms, Washington: Spartan Books, 1962.



