超大化学空间3D虚拟筛选新模式
摘要:虚拟筛选的局限性是由数据存储和处理所带来,任何超过对1亿个化合物的筛选都会带来问题。本文描述了一种全新的方法——Ignite技术,它使用基于场的方法解决数十亿化合物的3D虚拟筛选问题。我们认为,在理想情况下,打开这些超大化学空间应该可以为寻找新药发现所急需的新化学起点提供更多的机会。
作者:Martin Slater, Mark Mackey, Rosemary Mantell
编译:肖高铿/2024-07-17
前言
随着知识的增加,蛋白质在疾病中的作用日益有新的发现,并且具有被小分子靶向的潜力。如果第一个挑战是设计一个合适的检测方法来测试干预措施,那么下一个挑战就是找到合适的化合物来开始发现过程。但归根结底,药物发现需要设计一个足够有效、选择性和安全的候选药物,但关键还必须获得知识产权保护,以确保研究和投资成本的回报。
药物发现过程和现有的化学库(通常有数百万种真实化合物样品)不太可能符合这些新靶标和药物发现范式的要求。
虚拟筛选越来越多地作为一种赋能技术与实际湿筛选的补充而被部署使用,它可以访问更大的化学库空间(数千万个化合物),并且在满足生物靶标的条件时通常是适用的。然而,对于使用3D分子信息进行筛选,典型的局限性是由数据存储和处理所带来。任何超过1亿个化合物的筛选都会带来重大问题。
这张海报描述了一种全新的方法,使用一种全新的基于场的方法——IgniteTM技术——从可访问的合成化学库空间(如Enamine REAL Space)中解决数十亿化合物的3D虚拟筛选问题。我们认为,理想情况下,打开这些更大的化学空间应该可以为寻找新药发现所急需的新化学起点提供更多的机会。
方法
Ignite技术使用3个关键的专有处理步骤,可以在不需要完全枚举的情况下在3D中查询超大组合化学空间。这是通过(1)将组合空间分解为其组份合成子和(2)在枚举产物空间之前计算查询结构与反应物空间的3D场相似性来实现的。只有相似性高于设定阈值的反应物才能被(3)转化为产物空间,并分析3D场和形状相似性。
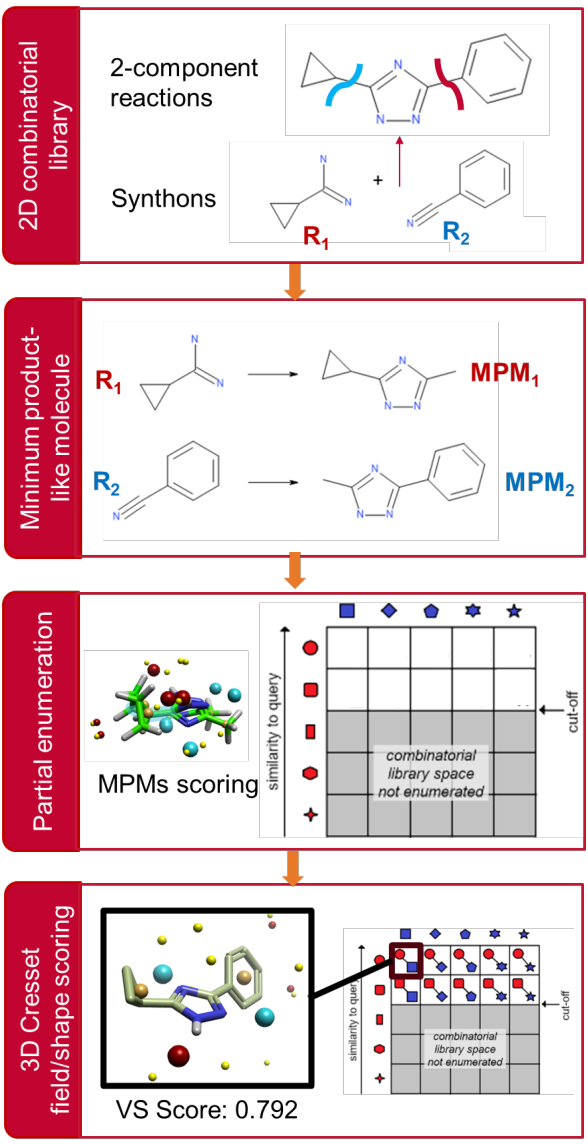
图1. Ignite原理流程图
技术细节
官能团和反应组分的识别是从Peter Ertl1描述的算法得到启发。“仅”双组分反应适用于该技术。每个反应用两个R基团来描述,它们在化学附着点被标记,RLF是反应物样片段(eactant like fragments)。出现在产物中的另一个组分包含的任何官能团也被用同样的方式定义并被标记,MPM是最小的产品样分子(minimum product like molecule)。
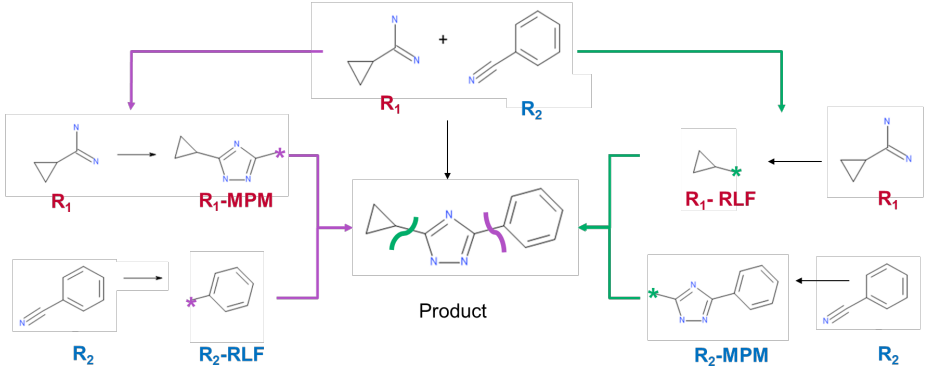
图2. 合成子的识别,RLF与MPM的定义
MPM是在3D场和形状空间中定义的,并形成一个数据库——编码较大的组合空间——可以根据3D分子查询进行匹配性检查。
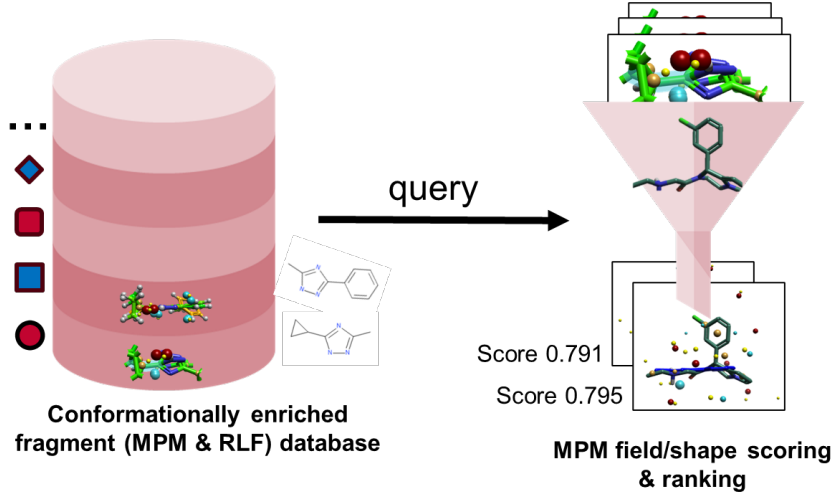
图3. MPM数据库的生成与查询
该过程定义并选择相对于3D查询哪些MPM组分值得枚举到产品。相似性阈值为每个反应定义了最佳RLF与MPM组合以形成富集的产物空间。反过来,使用相同的3D打分指标来从“产品空间”中确定最佳匹配。
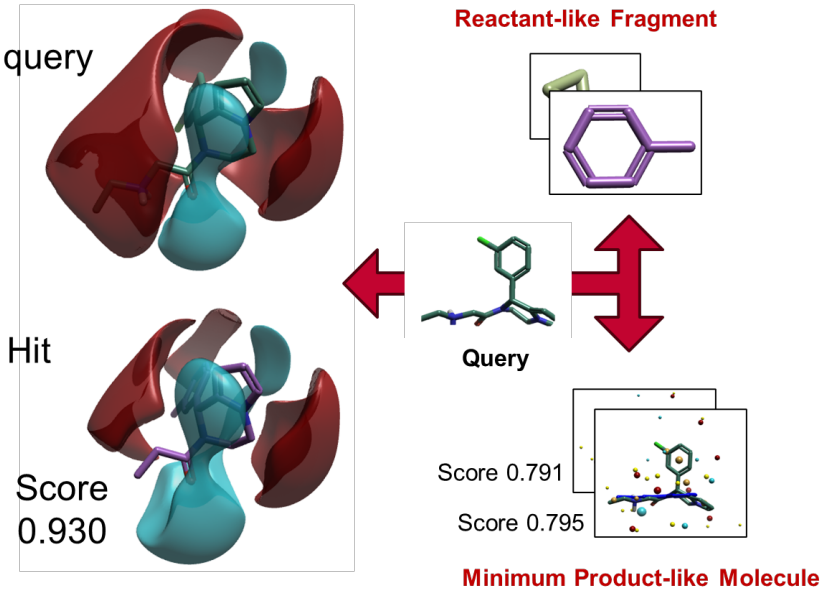
图4. MPM的选择和产物的匹配是通过相对于3D查询的场和形状相似性打分进行的。
Cresset拥有使用高级的静电和形状算法提供独特、强大的技术来描述和比较3D分子的记录。这些方法已经被充分用于从片段和产物空间中寻找药理学相关的分子匹配2,3。
结果
最初,这项技术是与拜耳公司合作开发的。这个想法是为Cresset的科学开发团队提供一个工具,可以高效地探索拜耳的内部化合物库空间,用于苗头化合物的发现和先导化合物的优化。“BaySpace3D”由易于获得的专有化学空间组成,其中包含约2亿个虚拟化合物。合作团队成功地开发并测试了这项技术,最后进行了联合案例研究:3904374 Bayspace3D searching Large 3D spaces with Cresset’s molecular fields,于2023年秋季ACS上发布4。
为了进一步验证该方法,这次使用Enamine的Real Space,以阐明如何为最近感兴趣的蛋白质靶标获得潜在有用的化学结果。
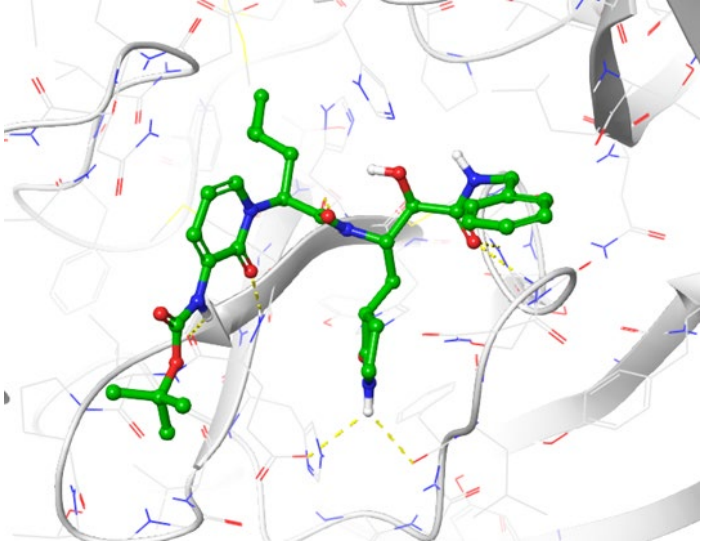
图5. SARS-Cov2主蛋白酶的晶体结构(Zhang et. al., Science, 2020, 368, 409-412.),3D分子模板输入用于IgniteTM筛选
来自X-衍射共晶结构的α-酮酰胺用于搜索REAL Space,提供了如下所示的示例匹配(图6)。
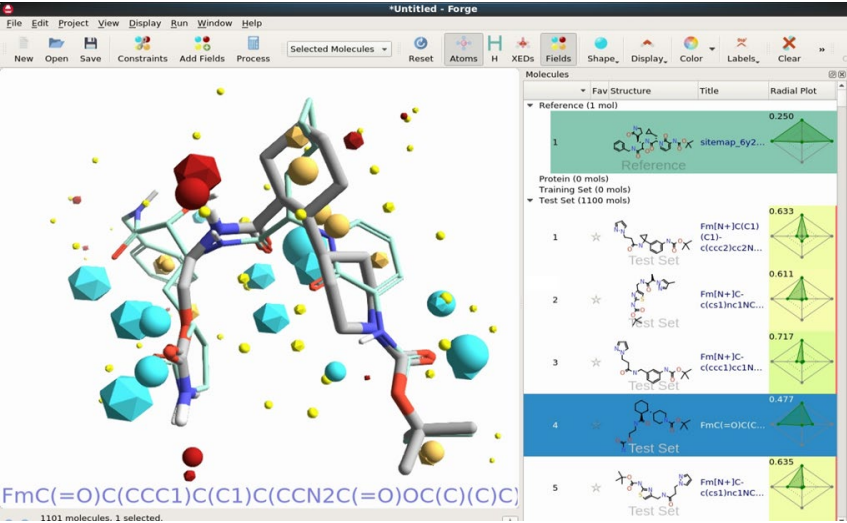
图6. Ignite筛选到的苗头化合物与模板的3D结合,用Cresset的分子模拟软件包Flare进行可视化分析
结论
IgniteTM代表了一种新颖、高效的方法,用于增强分子发现——高效地访问非常大的组合化学空间,一种独特地——并受益于真正的3D搜索方法。
将Ignite用于您的项目
目前Ignite作为一种专有技术服务通过外包应用于您的项目。Cresset发现服务拥有一支计算化学家专家团队,随时准备帮助您加速商业资产的创建。联系我们进行免费的保密讨论,以进一步探索合作方案。
服务流程
- 联系我们,进一步认识我们的业务
- 与Cresset签署三方保密协议
- 业务讨论、明确需求
- 报价
- 定制合同
- 实施
- 交付
文献
- Ertl, P. An algorithm to identify functional groups in organic molecules. J Cheminform 9, 36 (2017). https://doi.org/10.1186/s13321-017-0225-z
- Spark , Cresset®, Litlington, Cambridgeshire, UK; http://www.cresset-group.com/spark/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665-676
- Blaze , Cresset®, Litlington, Cambridgeshire, UK; http://www.cresset-group.com/blaze/; Cheeseright, T.J.; Mackey, M.D., Melville, J.L.; Vinter, J.G. FieldScreen: Virtual Screening Using Molecular Fields. Application to the DUD Data Set. J. Chem. Inf. Model. 2008, 48 (11), 2108-2117
- https://acs.digitellinc.com/sessions/573991/view



