摘要:通过一个热门的疟疾靶标,阐述了独特的3D Cresset场技术比常用2D指纹相似性方法的优势,并举例说明了BLAZE虚拟筛可降低对巨型化学空间探索并从中富集化学结构多样性的苗头化合物能力。
原文:Oliver Hills & Nathan Kidley. Avoiding chemotype bias in virtual screening
编译:肖高铿/2024-04-03
亮点
通过一个热门的疟疾靶标,阐述了独特的3D Cresset场技术比常用2D指纹相似性方法的优势;并举例说明了BLAZE虚拟筛可降低对巨型化学空间探索并从中富集化学结构多样性的苗头化合物能力。
背景
疟疾是一种通过蚊子传播的热带寄生虫感染性疾病,全球约2.19亿人感染,每年造成约435000人死亡。疟疾的致病寄生虫是恶性疟原虫,通过雌性蚊子传播给人类1。
抑制恶性疟原虫巨噬细胞迁移抑制因子蛋白(pfMIF,图1)的互变异构酶的活性一直是设计新型抗疟疾药物的核心药物设计方法2。

图1. 沿3-折叠轴向下视角的pfMIF功能性三聚体PDB 4P7M,放大部分举例说明了与稳定三聚体结构有关的氨基酸残基。
虚拟筛选的参比分子
先前的实验高通量筛选鉴定出两个具有nM Ki级别的有效pfMIF抑制剂3:
- 3-[(2-methyl-6-phenylpyridin-4-yl)oxy]phenol(化合物1)
- 4-(3-methoxy-5-methylphenoxy)-2-(4-methoxyphenyl)-6-methylpyridine (化合物2)
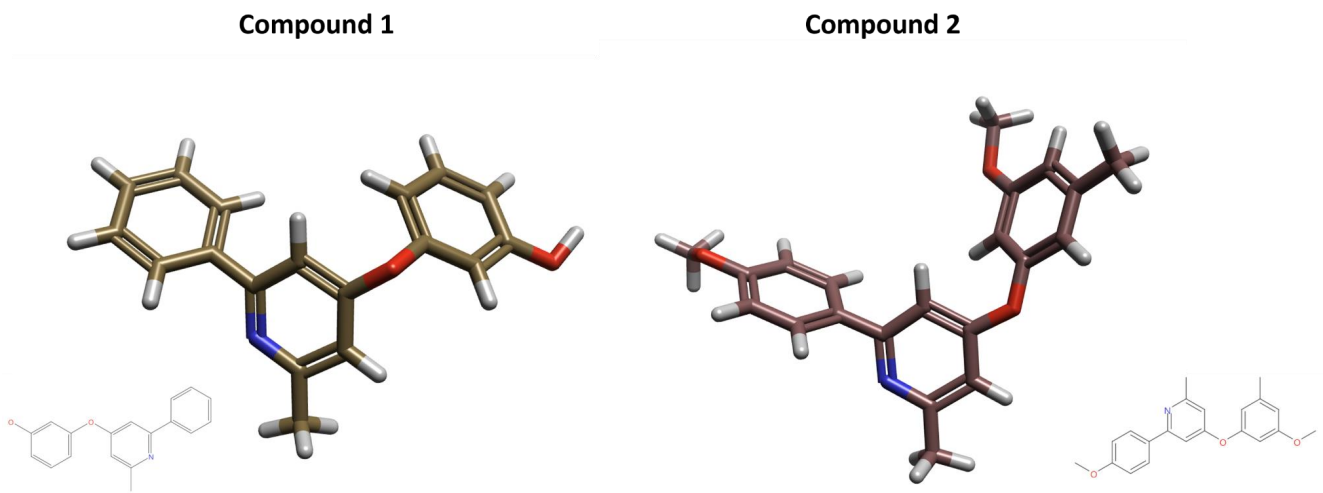
图2. pfMIF抑制剂化合物1、2及其生物活性构象
化合物1、2的生物活性构象通过公共3D场点(图 3)搜寻得到,并用作虚拟筛选的查询分子(query)。
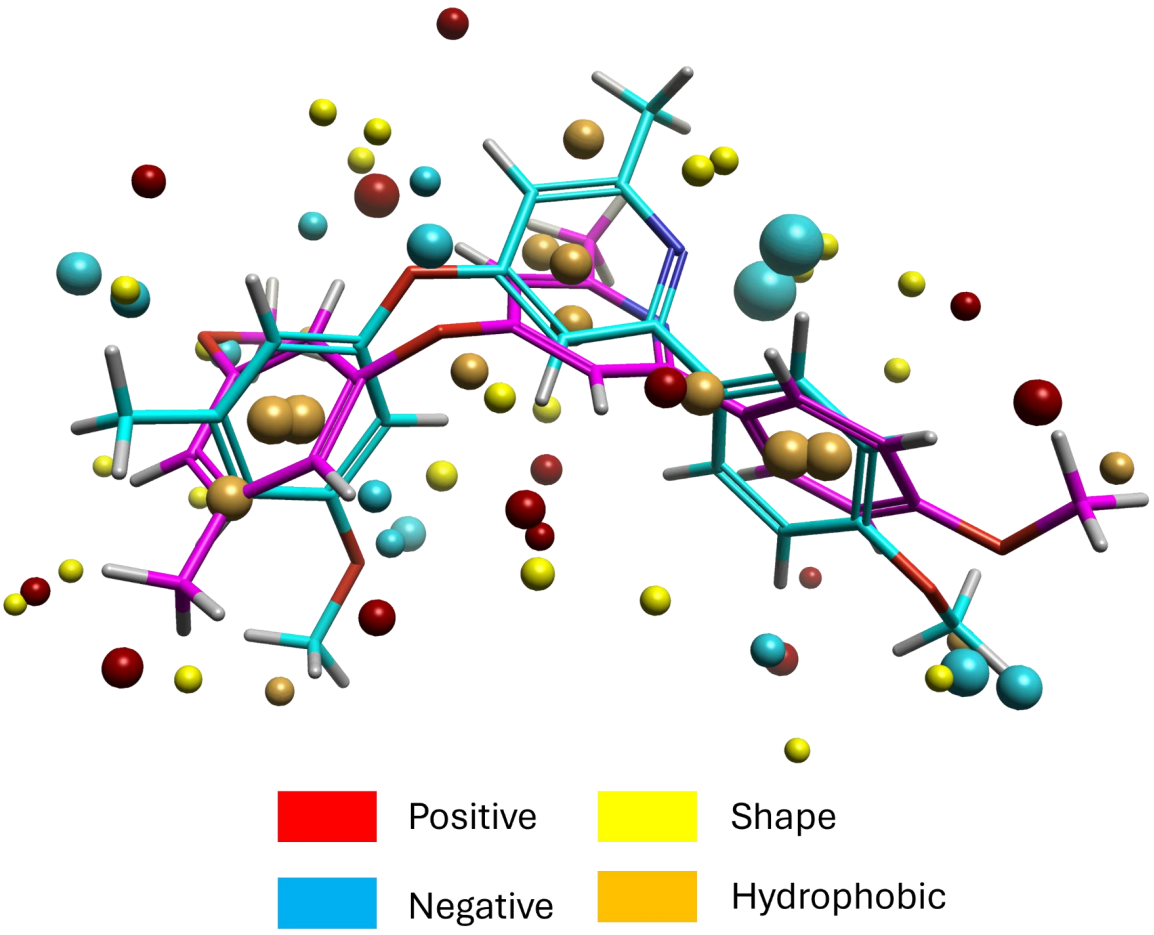
图3. pfMIF抑制剂化合物1、2生物活性构象及其叠合之后的场模式
BLAZETM虚拟筛选
BLAZE是Cresset先进的基于配体的虚拟筛选平台,它使用我们独特的3D场和形状相似性技术4。对于查询分子的3D生物活性构象,Blaze虚拟筛选工作流程如下:
- Blaze使用3D静电和形状相似性对商业现货化合物的构象数据库执行多层搜索
- 返回化学结构多样的类先导化苗头化合物的排序列表,每个苗头化合物都有一个3D结合模式
用化合物1、2作为查询种子,Blaze在全部的商业化合物数据库(3000多万化合物)里在全分子量范围内进行了多层搜索。具有高静电和形状相似性的化合物(图 4)作为虚拟筛选的苗头化合物,预计它们因此而“复制”了查询分子的生物活性。
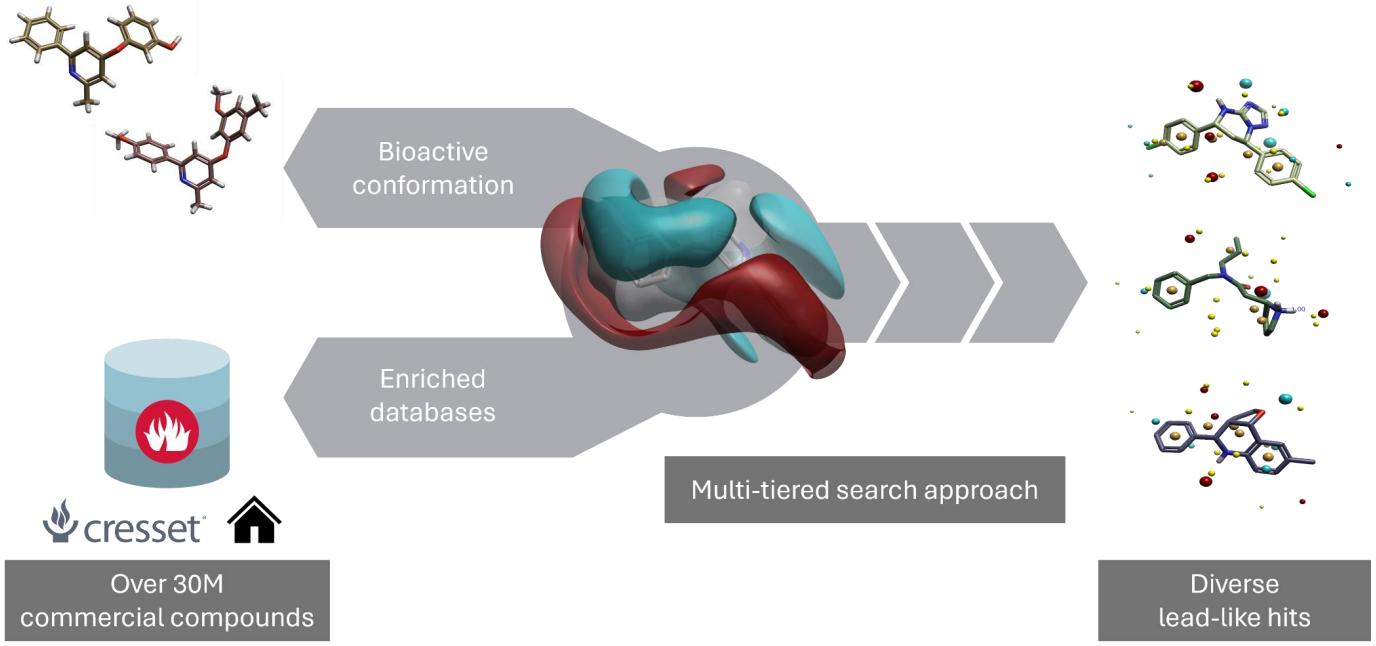
图4. BLAZE虚拟筛选工作流
2D VS 3D相似性
用2D RDK5指纹相似性分析BLAZE苗头化合物,可以清晰地表明:BLAZE识别的先导化合物具有广阔的化学多样性(图5)。
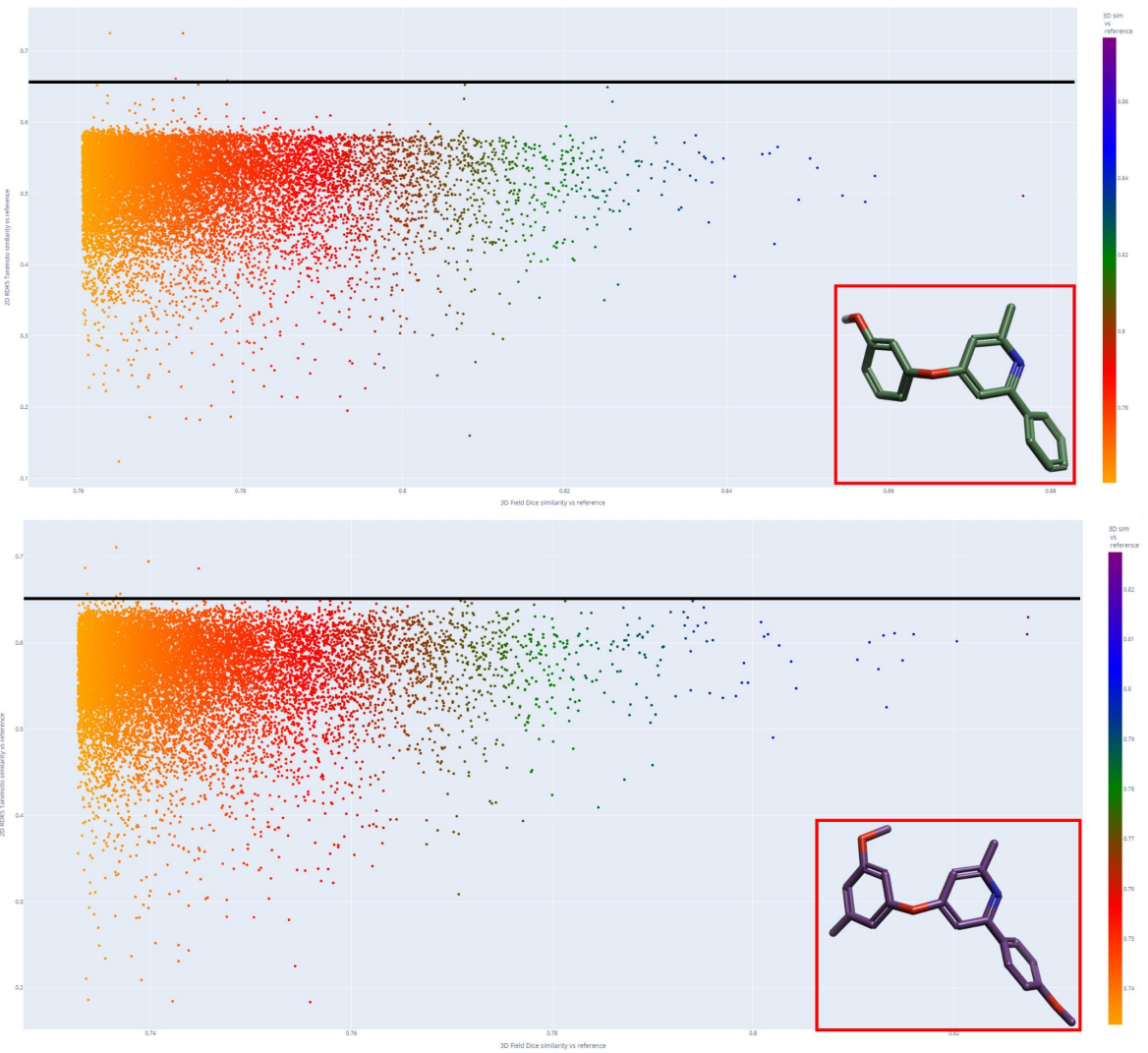
图5. 化合物1(上)和2(下)的2D RDK5指纹相似性与3D场相似性的散点图。数据点按与参比化合物的3D场相似性着色。化合物1和2显示为插图,2D相似性阈值0.65显示为黑线。
两张散点图都包含3D场相似性高但2D相似性低的配体:
- Blaze可以富集到化学多样性
- Blaze识别出的新分子与已知化合物的2D相似性远低于广泛使用的阈值0.655。
- 从3D场描述符空间里可以筛选到2D不相似化学空间的广阔区域
化学空间可视化
使用2D RDK5指纹相似性的t-分布随机邻域嵌入(t-SNE)投影来可视化所探索的化学空间的范围(图 6)。

图6. 化合物1(上)和2(下)BLAZE虚拟筛选苗头化合物的t-SNE图。外围显示的配体是来自绿色高亮显示的边缘化学聚类的示例配体。参比分子以红色高亮显示在聚类位置的旁边。
BLAZE识别出远离查询分子2D-化学空间的化合物:
- 在已探索化学空间“边缘”的化合物属于非常不同、且非显而易见的化学系列
- BLAZE苗头化合物并非直观设计的产物
- 尽管2D相似性较低,但3D静电场和形状场是保守的
- 尽管化学结构多样,但BLAZE的苗头化合物可与靶标蛋白形成相似的相互作用
避免化学结构类型上的偏好
对BLAZE的苗头化合物进行的Butina聚类分析表明,BLAZE具有克服子结构与化学类型偏好的能力,如图7所示。
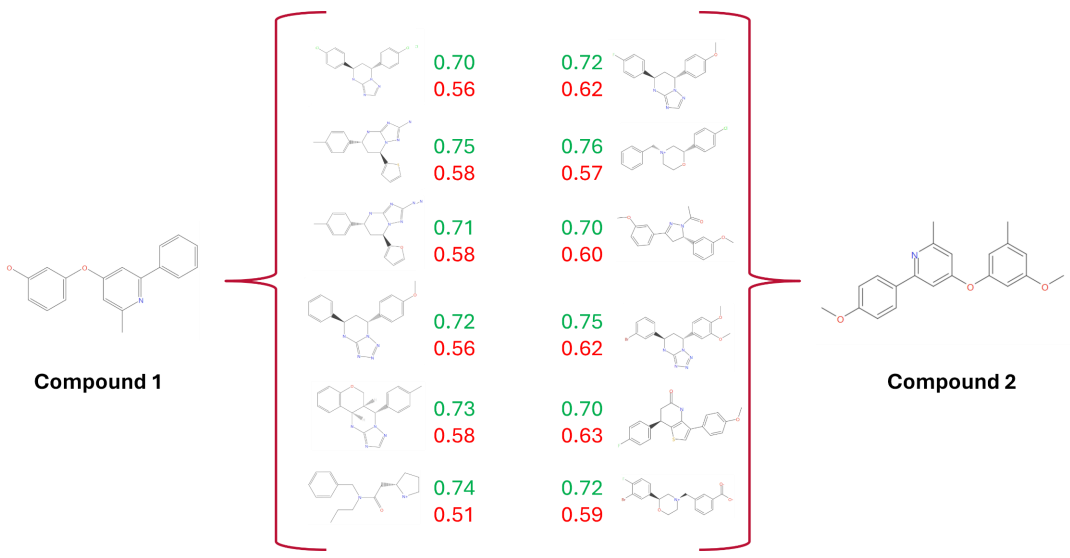
图7. 化合物1(左)和2(右)作为搜索种子,从Blaze虚拟筛选苗头化合物的前12个Butina聚类中获得聚类示例。3D静电和形状相似性打分以绿色显示,而2D相似性打分以红色显示。
参考文献
- Cordery, D.V., Kishore, U., Kyes, S., Shafi, M.J., Watkins, K.R., Williams, T.N., Marsh, K., Urban, B.C., 2007. Characterisation of Plasmodium falciparum macrophage migration inhibitory factor homologue. J Infect Dis 195, 905–912. https://doi.org/10.1086/511309
- Dahlgren, M.K., Baeza Garcia, A., Hare, A.A., Tirado-Rives, J., Leng, L., Bucala, R., Jorgensen, W.L., 2012. Virtual Screening and Optimization Yield Low-Nanomolar Inhibitors of the Tautomerase Activity of
Plasmodium falciparum Macrophage Migration Inhibitory Factor. J. Med. Chem. 55, 10148–10159. https://doi.org/10.1021/jm301269s - Pantouris, G., Rajasekaran, D., Baeza Garcia, A., Ruiz, V.G., Leng, L., Jorgensen, W.L., Bucala, R., Lolis, E.J., 2014. Crystallographic and Receptor Binding Characterization of Plasmodium falciparum
Macrophage Migration Inhibitory Factor Complexed to Two Potent Inhibitors. J. Med. Chem. 57, 8652–8656. https://doi.org/10.1021/jm501168q - Cheeseright, T., Mackey PhD, M., Rose PhD, S., Vinter PhD, A., 2007. Molecular field technology applied to virtual screening and finding the bioactive conformation. Expert Opin. Drug Discov. 2, 131–144.
https://doi.org/10.1517/17460441.2.1.131 - Maggiora, G., Vogt, M., Stumpfe, D., Bajorath, J., 2014. Molecular similarity in medicinal chemistry. J Med Chem 57, 3186–3204. https://doi.org/10.1021/jm401411z
使用BLAZE以及商务合作,请联系我们
Blaze可以通过三种方式使用:1)在我们管理的AWS云平台上计算:开箱即用;2)将软件安装在您自己本地GPU服务器或自己管理的AWS云平台上计算:需要部署、准备数据库等;3)委托我们计算(FTE服务)。



