摘要:本文介绍了辉瑞公司研究人员使用BLAZE虚拟筛选平台发现新型CCR6别构拮抗剂的研究。CCR6是与炎症性疾病如银屑病和炎症性肠病相关的趋化因子受体,抑制其信号传导为治疗提供了一种策略。研究者从已知的CCR6抑制剂方酰胺(SQA)衍生物出发,通过Cresset公司的BLAZE软件进行了基于场的3D相似性搜索,找到了化学结构不同的氧代吗啉(OXM)衍生物作为潜在的CCR6别构拮抗剂。该方法利用了XED力场计算分子静电特性,允许对原子周围的电荷分布进行准确描述,并通过四个分子场(正静电、负静电、表面及疏水性)的局部极值来表征分子。最终,从辉瑞内部数据库中发现了OXM1,其相似性得分为0.74。随后,对OXM系列化合物进行了构效关系(SAR)研究,其中OXM1和进一步优化得到的OXM2在抑制CCL20介导的CCR6+人类T细胞趋化性方面表现出良好的活性,pIC50值分别为6.8 ± 0.2 和 6.78 ± 0.11。这证明了BLAZE平台在寻找新化学类型先导化合物方面的潜力,也为CCR6相关疾病的药物开发提供了新的方向。
前言
CC趋化因子受体6(CCR6)表达于多种淋巴细胞中,包括T细胞,特别是T辅助17(Th17)细胞、B细胞和树突状细胞。CCR6由其特异性配体CCL20激活,刺激CCR6+淋巴细胞向炎症部位迁移。在慢性炎性疾病的患者上皮组织中,如银屑病和炎症性肠病(IBD)的表皮组织中,CCL20和CCR6的表达均上调。因此,抑制CCL20介导的CCR6信号传导是一种有前景的抗炎药物发现策略1,相应地,小分子CCR6拮抗剂可能作为相关疾病的口服治疗药。
最近,辉瑞公司的Ranasinghe等人2报道了可调控CCR6的新型别构抑制剂的发现2。具体来说,以已知的CCR6抑制剂方酰胺(squaramide,SQA)衍生物SQA1(图1)为起点,通过Cresset的BRAZE3软件虚拟筛选发现了新型的氧代吗啉(oxomorpholine )衍生物OXM1(图1),通过SAR研究还发现了OXM2(图2)。
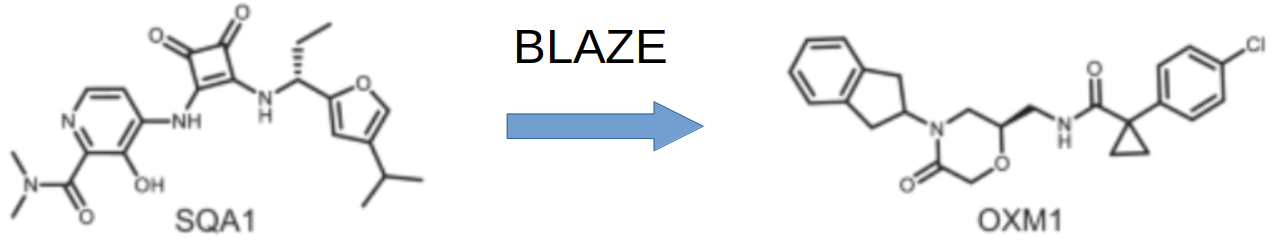
图1. 以方酰胺(SQA)为参比分子通过BLAZE虚拟筛选发现氧代吗啉(oxomorpholine )衍生物OXM1
本文的主要目的是简要介绍Ranasinghe等人2的虚拟筛选过程,以展示BLAZE在寻找新化学类型先导化合物方面的潜力。
用BLAZE进行虚拟筛选
BLAZE(以前也称为FiledScreen)是Cresset先进的基于配体的虚拟筛选平台,它使用Cresset独特的3D场和形状相似性技术从数据库种寻找与查询分子具有相似分子场点的化合物4-6。对于查询分子的3D构象,Blaze虚拟筛选工作流程如下:
- Blaze使用3D静电和形状相似性对构象数据库执行多层搜索
- 返回化学结构多样的苗头化合物的排序列表,每个苗头化合物都有一个3D结合模式
在Ranasinghe等人2的研究中,将小分子X-单晶衍射构象7作为查询分子(参比结构),使用Blaze(version 2.0)4-6进行基于场的3D-相似性搜索。简而言之,该方法利用查询配体的静电和形状来快速搜索具有类似性质的大型化学库。静电场计算的基础是可极化的XED力场,它允许对原子周围电荷分布进行准确描述。每个分子都用四种分子场的局部极大值表示的场点(Field Point)来描述:正静电、负静电、表面(范德华相互作用)和疏水性场点4。与传统的静电场方法不同,后者在原子中心放置单点偏电荷(partial charges),而XED力场这种方法则重新定义了向原子周围的多极电子分布的电荷。从这种表示中开发出的分子场被浓缩成它们的局部场点,并用于快速搜索并对具有类似静电特性的骨架进行打分,在辉瑞的内部数据库中找到OXM类似物OXM1,其相似性打分为0.74。
结果
除了虚拟筛选苗头化合物OXM1之外,还对OXM系列进行了SAR研究,结果发现,在抑制CCL20介导的CCR6 +人T细胞趋化性的活性测试上,SQA1、OXM1与OXM2都表现出相当好的抑制剂活性,如图2所示,它们的pIC50分别为: 6.7 ± 0.8,6.8 ± 0.2与6.78 ± 0.11。
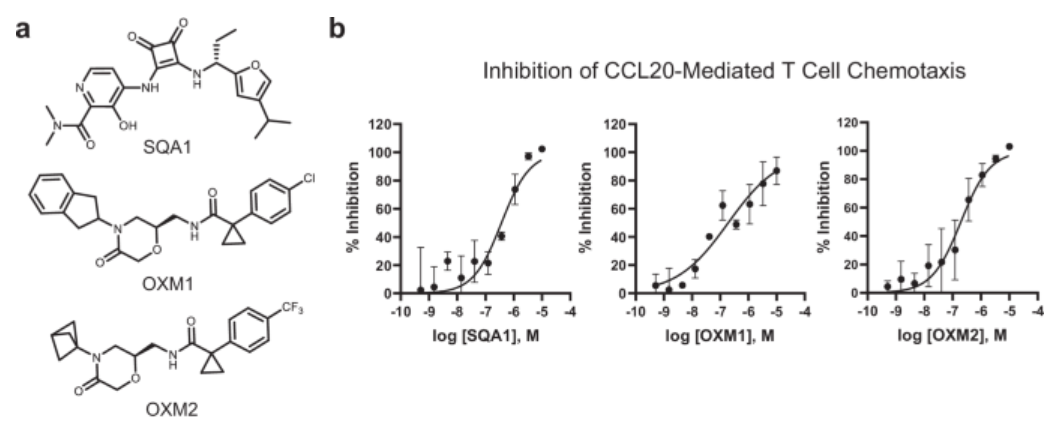
图2. a) SQA1、OXM1与OXM2的化学结构。 b)SQA1、OXM1与OXM2对CCL20-介导的CCR6+人类T-细胞趋化性抑制活性,pIC50分别为: 6.7 ± 0.8,6.8 ± 0.2与6.78 ± 0.11。数据呈现为独立3次实验的均值±标准偏差。
结论
总的来说,SQA与OXM在2D化学结构上没有任何相似的地方,但通过BLAZE独特的XED场点技术发现了它们在3D水平的相似性,实现了从一个化学系列向另一个化学系列跃迁并同时保存生物活性不变,BLAZE显示出了强大的寻找新化学类型先导化合物方面的潜力。
文献
- Ranasinghe, R. and Eri, R. (2018) “Modulation of the CCR6-CCl20 axis: A potential therapeutic target in inflammation and cancer,” Medicina (Lithuania). Available at: https://doi.org/10.3390/medicina54050088.
- Wasilko, D.J., Gerstenberger, B.S., Farley, K.A. et al. Structural basis for CCR6 modulation by allosteric antagonists. Nat Commun 15, 7574 (2024). https://doi.org/10.1038/s41467-024-52045-7
- Blaze (version 2.0). Cresset, Litlington, Cambridgeshire, UK. https://www.cresset-group.com/software/blaze
- Cheeseright, T. et al. (2006) “Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation,” Journal of Chemical Information and Modeling, 46(2), pp. 665–676. Available at: https://doi.org/10.1021/ci050357s.
- Cheeseright, T. et al. (2007) “Molecular field technology applied to virtual screening and finding the bioactive conformation,” Expert Opinion on Drug Discovery, 2(1), pp. 131–144. Available at: https://doi.org/10.1517/17460441.2.1.131.
- Cheeseright, T.J. et al. (2008) “FieldScreen: Virtual Screening Using Molecular Fields. Application to the DUD Data Set,” Journal of Chemical Information and Modeling, 48(11), pp. 2108–2117. Available at: https://doi.org/10.1021/ci800110p.
- Groom, Colin R et al. (2016) “The Cambridge Structural Database.” Acta crystallographica Section B, Structural science, crystal engineering and materials vol. 72,Pt 2: 171-9. Available at: https://doi.org/10.1107/S2052520616003954.
联系我们
Blaze是Cresset先进的基于配体的虚拟筛选工具,采用GPU加速计算,可以通过多种方式来使用:
- 本地安装
- 在自己管理的云设施上安装
- 在Cresset管理的云设施上安装:简单、快速
- 在cresset管理的云上使用:按项目计费,简单、快速
- 以服务的方式使用Blaze:委托计算服务
Blaze计算原理与Flare的Align一样,只是Flare是CPU版本。如果想测试,有两种方式:
- 按项目计费的方式,在我们管理的云上使用Blaze:需要付费、直接对2300万有商品可供采购的化合物虚拟筛选、快速且方便
- 小规模计算可通过Flare Align在本地CPU集群计算:需要自己准备数据库,计算速度慢
请现在就联系我们安排试用。我们的专业团队随时准备通过安装和设置为您提供支持,而我们全面的教程库——涵盖从常见工作流程到高级方法和功能的所有内容将帮助您开始使用。我们在这里帮助您更快地实现目标,让您设计出重要的分子。
- 电邮:info@molcalx.com
- 电话:020-38261356



