摘要:本文演示了如何连用Blaze与Flare对虚拟筛选苗头化合物进行优先级排序来发现靶向TYK2 JH1假激酶结构域的抑制剂。具体地讲,先采用 Blaze™虚拟筛选平台的对数据库进行虚拟筛选,发现靶向TYK2的虚拟筛选苗头化合物;再用Flare的构象分析工具过滤掉含有CSD低频分布两面角的分子;接着用Flare进行Lead Finder分子对接打分、静电互性打分与相似性生成多参数函数(径向图函数,RPF);将RPF打分靠前的1%化合物进一步进行人工视觉分析与过滤等后处理,最后得到化合物采购列表,推荐化合物进行活性测试。
作者:Morgan Morris, Jessica Plescia, Sílvia Tuset, Andy Smith, Stuart Firth-Clark
编译:肖高铿
前言
非受体酪氨酸蛋白激酶TYK2是一种参与IFN-α通路和各种白细胞介素通路调节炎症和免疫的酶。目前,人们正在研究其用于类风湿关节炎和牛皮癣等疾病的治疗1。
人们非常关注选择性靶向TYK2抑制剂的开发,同时避免保留结构相似的酪氨酸激酶JAK 1、2和3的功能。图1展示了一种采用 Blaze™2虚拟筛选平台的工作流用于发现针对TYK2的虚拟筛选苗头化合物。
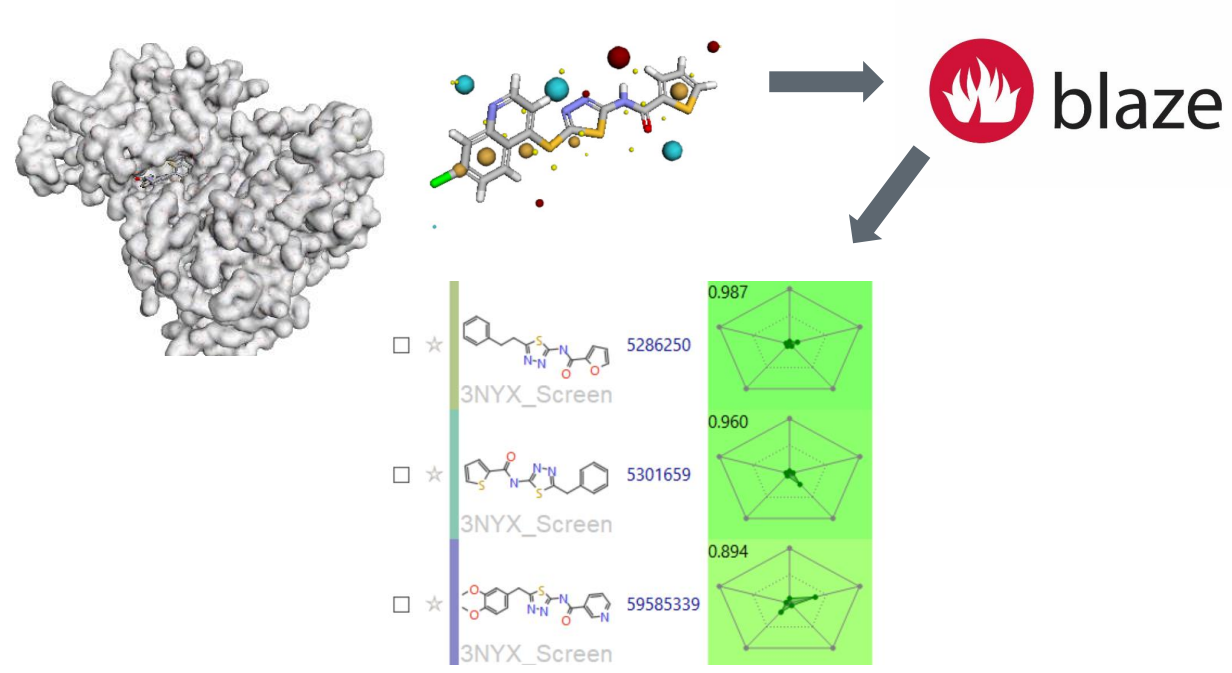
图1. 工作流用TYK2共晶结构 (PDB = 3NYX)的配体作为Blaze的输入进行虚拟筛选,然后在Flare™3 中对数据进行进行分级审查
方法
对47个TYK2共晶结构进行分析,并对其配体进行聚类分析。然后用下面的标准进行选择:
- 手性中心小于4个
- 分辨率不超过2.5Å
- 配体系列的活性重合小于1
初步过滤之后保留了22个PDB复合物结构,其共晶配体用XED力场描述的静电与形状进行聚类。每一类用生物活性最高的那个配体作为代表,并将其生物活性构象作为参比分子用于Blaze虚拟筛选,如图2所示。
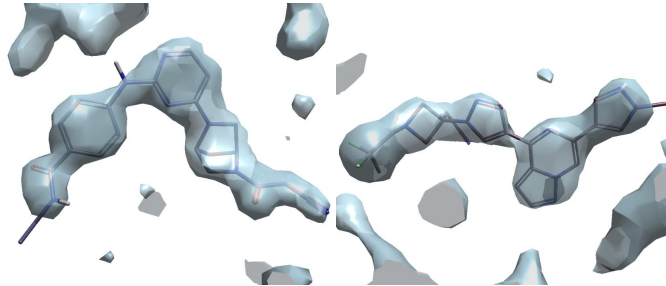
图2. 在使用XED力场选择5个结构多样的参比化合物之前根据电子密度图检查配体的晶体结构构象。左下面板=化合物2;右下面板=化合物4。
Blaze虚拟筛选
表1. 本研究用到的5个共晶结构配体
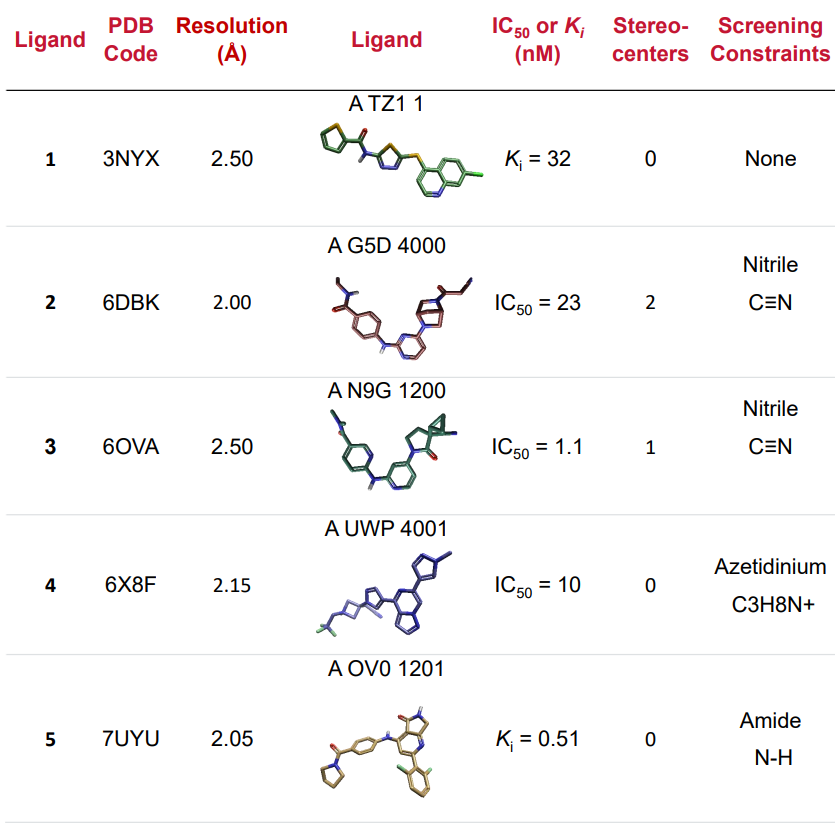
以TYK2 JH1假激酶结构域4结合的5个结构多样的代表性配体(表1)作为参比化合物,与配体共晶的蛋白作为排除体积,使用Blaze对一个大约包含2200万市售化合物的数据库进行虚拟筛选。对筛选的化合物进行构象采样,并与每个的参比化合物进行叠合,保留打分最高前 50%(clique refinemnet),然后进行单纯型优化(simplex refinement)保留打分最高最高的前10%。与参比化合物的相似性中,基于形状与场5打分的权重分别为 50% 与50%(图 3)。
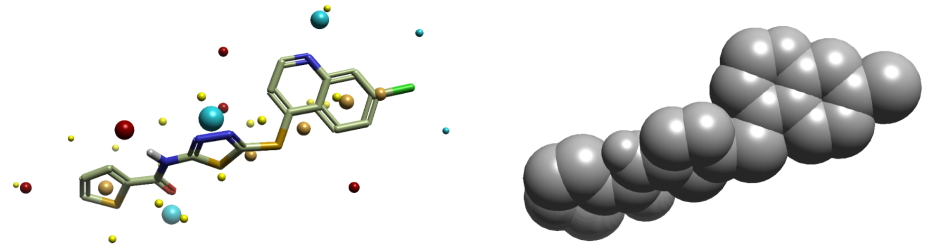
图3. 使用配体1作为参比分子在Blaze中进行相似性评分的概述。50%来自场描述,50%来自形状。红色=正场点; 青色=负场点;橙色=疏水场点; 黄色 = 高度可及的范德华表面。
为了在虚拟筛选过程中促进形成必要的相互作用,Blaze在关键官能团上添加了药效团约束(表1)。对五个虚拟筛选的各自排名前1000化合物进行一步用结构过滤器进行分析,并去除有问题的化合物。
在Flare进行数据分级审查
在子结构过滤之后,如果分子有两面角处于CSD低频分布,则过滤掉该分子6。然后,将剩余的虚拟筛选命中化合物转移到分解审查阶段。
在Flare中对过滤之后的分子用下面的正交方法进行重新打分:(a)在干的蛋白结合口袋里用LeadFinder™分子对接7打分函数重新打分,以及(b)计算配体和蛋白之间的静电互补性™(EC)8。
然后使用对接打分、EC打分、相似性打分生成多参数函数(在Flare中称为径向图函数[RPF])。
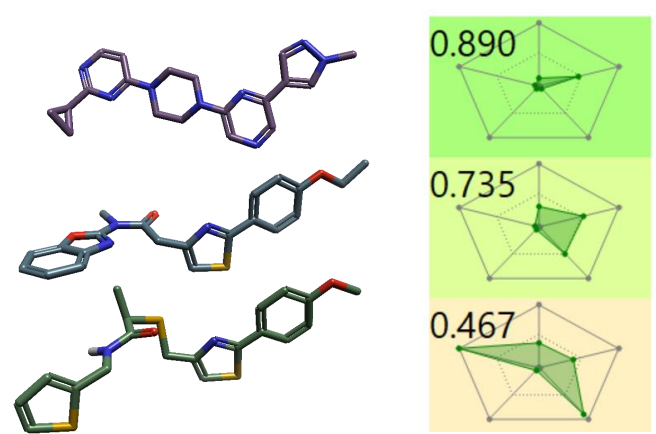
图4. 在Blaze中用配体4进行虚拟筛选获得的3个虚拟苗头化合物的RPF分析。Cresset的Flare允许用户使用自己的参数进行自定义径向图设计。
ROC分析
为了测试Flare RPF的有效性,将9个与化合物5密切相关的已知活性化合物纳入了测试集,其包含1400个具有相似物理和结构性质的化合物。对测试集进行虚拟筛选,生成ROC曲线(图5)。
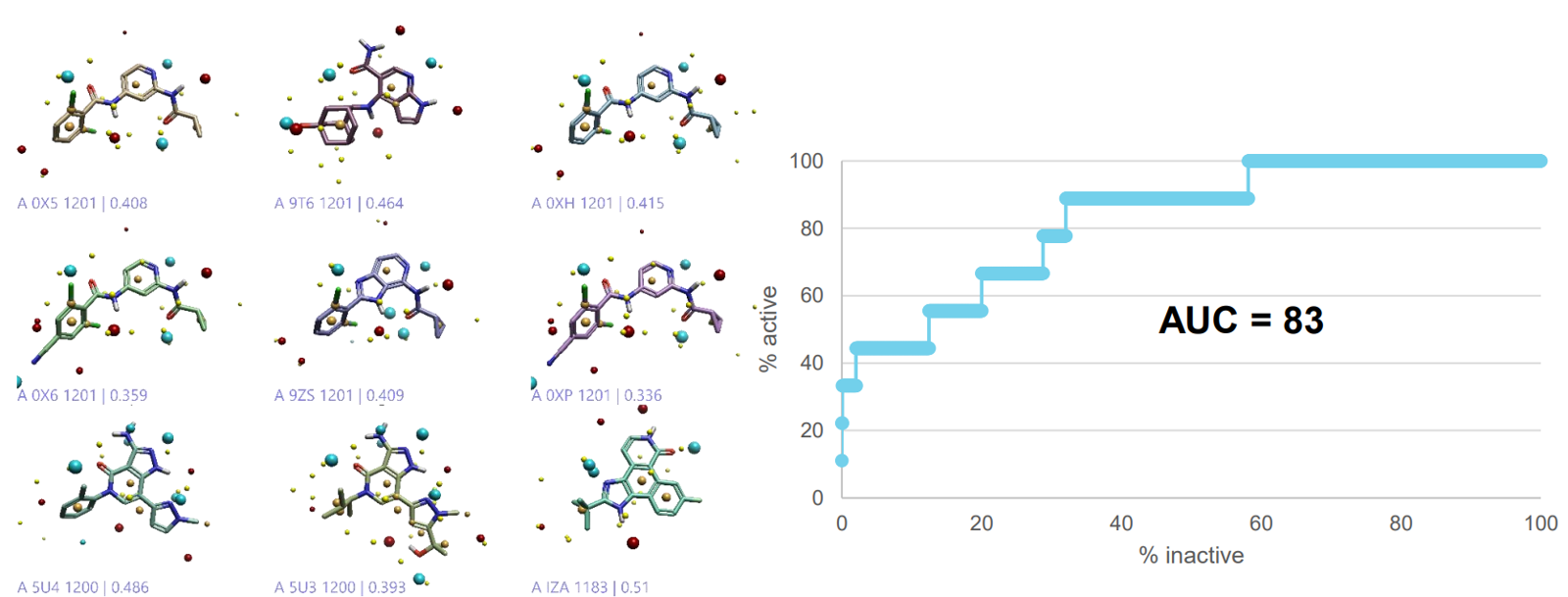
图5. 测试集命中的3D结构用来测试多参数打分函数。通过ROC曲线下面积(AUC)可外推数据的富集能力。
对于所选的RPF(对接打分、EC打分和相似性打分的组合),作为虚拟筛选苗头化合物分级分析的一部分,用前1%(大于等于0.46)的阈值作为截止打分。
蛋白配体相互作用分析
在RPF打分评估之后,使用两种方法对虚拟筛选苗头化合物进行进一步的人工视觉检查:
- 仅用配体信息:用Watson Bruns Eli Lilly规则过滤结构9。
- 用蛋白-配体信息:如果苗头化合物没有与关键的铰链结合残基Val981发生至少一种接触,则将其过滤掉。
使用FCFP6对所选化合物进行最终的聚类分析以生成结构多样的有商品可供购买虚拟筛选苗头化合物列表。
结果
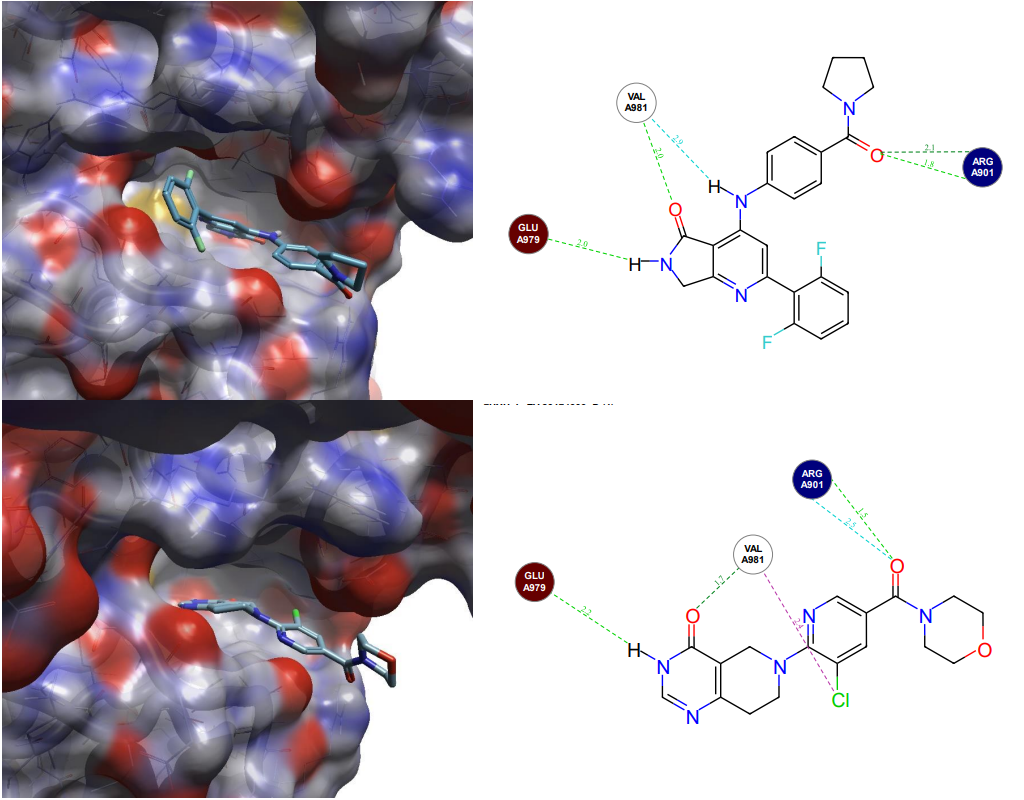
图6. 上:配体5与TYK2铰链区和富含甘氨酸的loop相互作用。下:发现了一个虚拟筛选苗头化合物,它通过与Val981的卤键与铰链区结合。蛋白-配体相互作用:浅/深绿色=强氢键;浅蓝色=弱氢键;浅紫色=卤键。

图7. Flare的EC表面8突显了与天然配体3(左图)相比,含溴呋喃的虚拟筛选苗头化合物(右图)的相互作用得到了改善。红色=与蛋白质冲突;绿色=蛋白质-配体互补性;灰色=中性。
结论
本文对TYK2 JH1假激酶结构域使用Blaze-Flare工作流虚拟筛选得到化学结构多样且具有新颖相互作用模式的苗头化合物。真正验证虚拟筛选的唯一方法是获取虚拟筛选苗头化合物并进行生物学测试,Cresset的客户已经成功地验证了许多筛选筛选的结果。
文献
- Leit S, et al.Potent and selective TYK2-JH1 inhibitors highly efficacious in rodent model of psoriasis. Bioorg Med Chem Lett. 2022 Oct 1;73:128891. doi: 10.1016/j.bmcl.2022.128891.
- Blaze™, version 10.8, Cresset®, Litlington, Cambridgeshire, UK; http://www.cresset-group.com/blaze
- Flare™, version 7, Cresset®, Litlington, Cambridgeshire, UK; http://www.cresset-group.com/flare
- Tsui V, Gibbons P, Ultsch M, et al. A new regulatory switch in a JAK protein kinase. Proteins Struct Funct Bioinforma. 2011;79(2):393-401. doi:10.1002/prot.22889
- Cheeseright T, Mackey M, Rose S, Vinter A. Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J Chem Inf Model. 2006;46(2):665-676. doi:10.1021/ci050357s
- Penner P, Guba W, Schmidt R, Meyder A, Stahl M, Rarey M. The Torsion Library: Semiautomated Improvement of Torsion Rules with SMARTScompare. J Chem Inf Model. 2022;62(7):1644-1653. doi:10.1021/acs.jcim.2c00043
- Stroganov O V, Novikov FN, Stroylov VS, Kulkov V, Chilov GG. Lead Finder: An Approach To Improve Accuracy of Protein−Ligand Docking, Binding Energy Estimation, and Virtual Screening. J Chem Inf Model. 2008;48(12):2371-2385. doi:10.1021/ci800166p
- Bauer MR, Mackey MD. Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes. J Med Chem. 2019;62(6):3036-3050. doi:10.1021/acs.jmedchem.8b01925
- Bruns RF, Watson IA. Rules for Identifying Potentially Reactive or Promiscuous Compounds. J Med Chem. 2012;55(22):9763-9772. doi:10.1021/jm301008n
获取软件试用与技术合作,请联系我们
想在自己的项目上使用Flare进行多参数优先级排序或进行项目合作机会,请联系我们。



