基于结构的过滤提高分子对接虚拟筛选的富集性能
摘要:本文以DUD-E的NRAM为子集为例,演示了如何用Flare的相互作用分析对Lead Finder分子对接虚拟筛选结果进行基于结构的过滤来实现富集性能的提升。具体而言,首先用Flare的3D-RISM分析、PIP分析、结合文献确定相互作用的关键残基;接着用Flare对虚拟筛选结果进行相互作用分析,过滤掉不能与关键残基发生相互作用的化合物;接着对保留下来的化合物进行二次打分,优先级排序与性能统计。结果表明,基于结构的过滤使得虚拟筛选早期富集性能指标EF1%从LF VS-score的1.0提高到LF_CNN_VS与CNN_VS的49.0;而另一个早期富集性能指标BEDROCα=80.5从LF VS-score的0.02提高到LF_CNN_VS的0.76与CNN_VS的0.80。总的来说,基于结构的过滤提高了分子对接虚拟筛选的早期富集性能。
肖高铿/2023-10-12
前言
Fishcer等人1的调研表明,人们基本达成共识:成功的分子对接虚拟筛选需要一定的人工后处理。Novikov与Stroylov2提出的基于结构的过滤(Structural filtration)是一种创新的分子对接结果后处理方法,基于结构的过滤器由一组蛋白特异性相互作用定义,这些相互作用不仅在特定蛋白与其结合配体的结构上保守,而且还被视为在蛋白-配体结合中起关键作用。他们为此开发了相应的过滤器,并将其应用于Lead Finder分子对接软件3-5获得的虚拟筛选结果上。在一组10种不同的蛋白靶标上进行了评估,结果表明,基于结构的过滤显著地改善了富集因子(Enrichment factor,EF),根据蛋白质靶标的不同,富集因子改善从几倍到几百倍不等。最近,Gushchina等人6开发了一种基于结构的对接结果后处理应用工具vsFilt,显著地提高了Lead Finder分子对接虚拟筛选的富集性能。
Flare7内置的蛋白-配体相互分析工具Contact analysis可以帮助我们进行此类基于结构的过滤后处理,在之前的博客中也分享过如何对Vina与Uni-Dock(一种Vina的GPU实现版本)的结果进行过滤以提高虚拟筛选的效率以便在保持性能的前提下节省大量昂贵的GPU计算成本8,9。此外,Flare的Contact analysis还可以与基于配体的方法Blaze联用,作为分级过滤的一个环节来对最有前景的虚拟筛选苗头化合物进行优先级排序,最终发现靶向TYK2 JH1假激酶结构域的抑制剂10。
本文的目的是以DUDE NRAM(neuraminidase,神经氨糖酸苷酶)子集11为例,采用图1的虚拟筛选流程,来演示基于结构的过滤如何增强Lead Finder的虚拟筛选富集性能,最终实现早期富集性能指标EF1%从1.0提高到49.0的48倍提升。
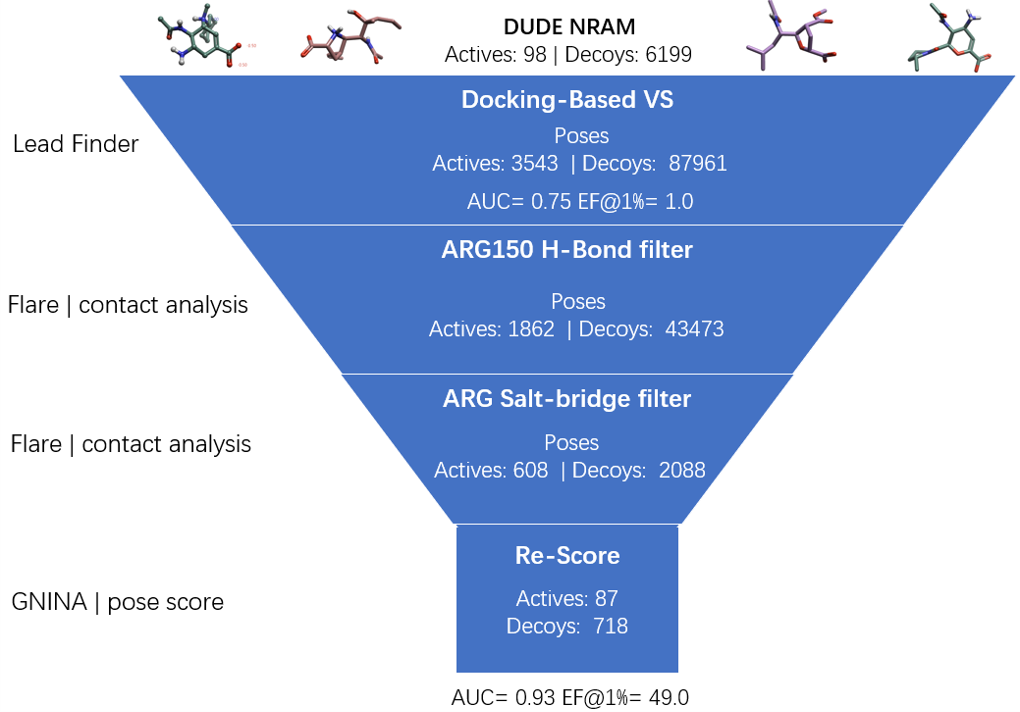
图1. 虚拟筛选流程图,包括:分子对接虚拟筛选、基于结构的过滤与重新打分
材料与方法
数据库分子的准备
DUD-E NRAM子集11里预先准备好的SDF格式actives与decoys结构直接用于虚拟筛选,不进行额外的准备。
蛋白结构的准备
用Flare下载共晶结构PDB 1B9V,并用Protein Prep工具进行蛋白结构准备,仔细检查结构以确保配体结构正确、相互作用合理、最大化配体与蛋白的氢键相互作用网络,删除全部的水,仅保留A链的蛋白部分与配体,然后分别导出蛋白结构(1b9v_prot.pdb)与配体结构(1b9v_ligand.sdf)备用。
Lead Finder分子对接虚拟筛选
Flare Docking以Lead Finder为计算引擎,典型的计算流程包括4个步骤:1)蛋白模型构建;2)能量格点计算;3)在能量格点里对接分子;4)打分。分子对接虚拟筛选使用pyflare的docking脚本,在命令行下计算能量格点并执行虚拟筛选。
Flare Docking有三种分子对接模式:标准、穷尽与虚拟筛选。在本文中采用虚拟筛选模式。Lead Finder的打分函数是一种半经验的力场方法,并显式地考虑了不同类型的分子间相互作用。对每个打分成分用一个经验系数进行缩放,得到三种不同目的的打分函数:
- dG-score:预测结合亲和力
- VS-score:在虚拟筛选中对不同的分子排序
- Rank-Score:对结合模式进行排序
采用上一步准备好的蛋白结构,以共晶配体为参比分子,进行能量格点计算:
1 2 3 4 5 6 7 8 | pyflare docking.py -p 1b9v_prot.pdb -j pdb --protein-chain-types p \ --reference 1b9v_ligand.sdf -i sdf \ --write-grid 1b9v_prot.grid \ --csv redock.csv \ --max-poses 20 \ --batch-size 120 \ --quality virtual-screening \ 1b9v_ligand.sdf -i sdf >> redock.sdf |
计算完毕,生成了一个新的文件1b9v_prot.grid即为所需的能量格点文件。
actives虚拟筛选:
1 2 3 4 5 6 7 | pyflare docking.py -p 1b9v_prot.pdb -j pdb --protein-chain-types p \ --grid-from-file 1b9v_prot.grid \ --csv actives_dock.csv \ --max-poses 20 \ --batch-size 120 \ --quality virtual-screening \ actives_final.sdf -i sdf >> actives_dock.sdf |
计算完毕得到的actives_dock.sdf文件即为actives数据集对接结果文件。
decoys虚拟筛选:
1 2 3 4 5 6 7 | pyflare docking.py -p 1b9v_prot.pdb -j pdb --protein-chain-types p \ --grid-from-file 1b9v_prot.grid \ --csv decoys_dock.csv \ --max-poses 20 \ --batch-size 120 \ --quality virtual-screening \ decoys_final.sdf -i sdf >> decoys_dock.sdf |
计算完毕得到的decoys_dock.sdf文件即为decoys数据集对接结果文件。
用GNINA进行重新打分
GNINA是一款采用深度学习CNN模型的分子对接软件12,13,在本文中用来对Flare Docking结果再打分。用GNINA对actives对接结果进行重新打分:
1 | gnina --config dock.conf -l actives_dock.sdf -o actives.sdf --score_only |
用GNINA对decoys对结果结果进行重新打分:
1 | gnina --config dock.conf -l decoys_dock.sdf -o decoys.sdf --score_only |
其中dock.conf为gnina对接参数文件,内容如下:
1 2 3 4 5 6 7 | receptor = 1b9v_prot.pdbqt center_x = 31.5685 center_y = -9.4545 center_z = 63.8425 size_x = 30 size_y = 30 size_z = 30 |
GNINA打分之后,在sdf文件里包含了3个新的打分值:
- CNNscore: GNINA的CNN模型pose打分值
- CNNaffinity:GNINA的CNN模型结合亲和力预测值
- CNN_VS:CNNscore x CNNaffinity
数据融合
Flare docking对接计算产生LF_VSscore,LF_dG与LF_Rank_Score等三个打分值。用GNINA重新打分之后,得到三个新的打分值CNNscore,CNNaffinity与CNN_VS。其中CNN_VS是CNNscore与CNNaffinity的乘积,即:
CNN_VS = CNNaffinity x CNNscore
在本文中,将CNNscore与LF_VSscore进行相乘:
LF_CNN_VS = LF_VSscore x CNNscore
CNN_VS与LF_CNN_VS是一种一致性打分策略,可以提高虚拟筛选富集能力15。
3D-RISM计算
用Flare中的3D-RISM模块针对准备好蛋白结构进行分析,使用了如下条件:
- XED force field and charge method
- 0.4Å grid spacing
- 14Å grid external border width
- Convergence tolerance: 10-8
- Maximum number of iterations: 10,000
- Total formal charge handling: neutralize with counterions.
蛋白相互作用势分析(PIP)
蛋白相互作用势(Protein Interaction Potential, PIP)是Cresset分子相互作用势对蛋白质的延伸,使用XED力场计算的。该方法在原理上类似于配体场的计算:蛋白质的活性位点被探针原子充满,并且计算每个格点上的相互作用势。该方法利用了Mehler等人14的距离依赖的介电函数来更好地处理蛋白质结构中的大量带电基团。在本文中,仅计算和显示活性位点的蛋白质相互作用电势。
Flare基于结构的过滤
Flare除了提供可视化分析之外,还提供了Python API通过cresset.flare.contacts来分析各种相互作用,以帮你从分子对接结果中过滤出满足条件的分子或pose。这些相互作用包括:
- aromatic_aromatic
- cation_pi
- h_bonds
- halogen_bonds
- steric_clashes
- sulfur_lone_pair
- salt_bridge
将上述的对接结果SDF文件导入到Flare之后,点击Flare | Python | Python Interpreter,打开Python解释器(图2),可以通过Python对Docking的结果相互作用分析。
图2. Flare的Python解释器
数据处理与虚拟筛选性能评估
鉴于从DUD-E下载的actives与decoys数据集进行了互变异构体与质子化状态枚举处理,导致一个化合物会以不同的形式多次出现、并可能被虚拟筛选多次命中,因此对虚拟筛选结果按化合物名称进行去重,同名化合物仅以其中打分最高那个来代表,所有的性能评估指标都在去重的基础上进行计算。
在虚拟筛选过程中,有可能有的化合物处理失败而没有出现在结果文件里,为了保证性能评估的结果与其它文献报道的具有可比性,将缺失的化合物人为地分配一个打分值0然后再进行性能评估。
用AUC、logAUC、三种α(321.9,80.5与20.0)值的BEDROC、富集因子(top 0.5%,1%,5%与10%)等指标来评估虚拟筛选的性能,采用ODDT的metrics模块来计算16。
结果与讨论
数据处理
对接结果用python pandas聚合函数groupby对化合物分值进行处理:同一个化合物的互变异构体、不同质子化状态仅保留打分最优的那个作为该化合物的打分值。actives与decoys数据的处理结果如表1所示。其中20个decoys化合物没有出现在了结果里面,这些处理失败的分子给予一个打分值0之后参与统计,以便不同方法得到的性能指标具有可比性。
表1. DUD-E NRAM数据集化合物统计
| NRAM数据集 | 化合物数 | 异构体数 | Pose数(化合物数) |
|---|---|---|---|
| actives | 98 | 222 | 3543(98) |
| decoys | 6199 | 6227 | 87961(6178) |
其中括号里的数为不重复的化合物数
3D-RISM分析
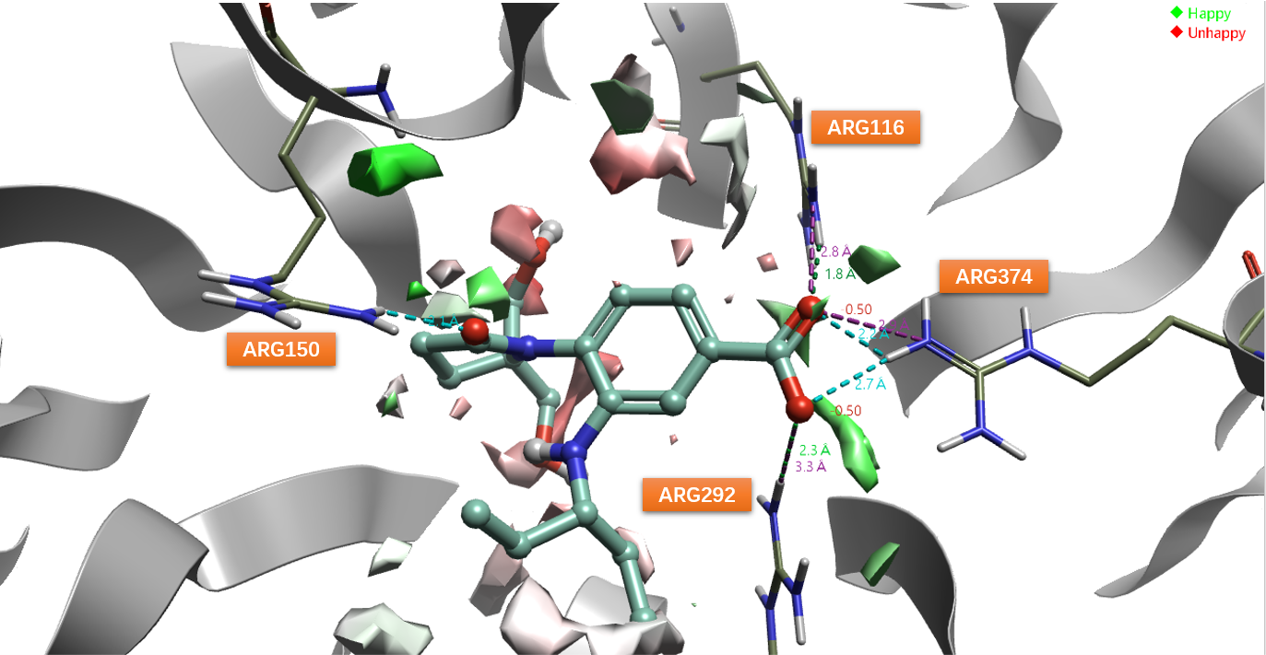
图2. 3D-RISM分析: 呈现了ρO大于5的等值面并根据ΔG着色。
一般来说,高密度水占据的位置是药效团位点,杂原子出现在高密度水位置上是pose正确的一个信号17。对蛋白apo结构结合位点的3D-RISM分析结果表明,如图2所示,在共晶配体羧基与内酰胺环的碳基氧所在位置被高密度(ρO大于5)的水覆盖,这说明该羧基片段与内酰胺环羰基参与的相互作用可能很重要。
PIP分析

图3. PIP分析。蓝色:负静电势等值面(ve=-0.5);红色:正静电势等值面(ve=+0.5)
对蛋白apo结构结合位点的PIP分析结果表明,如图3所示,在共晶配体羧基与内酰胺环的碳基氧所在位置被红色正静电势覆盖(+ev大于0.5),蛋白的PIP与配体的的化学性质互补。
相互作用分析与基于结构的过滤

图4. 基于结构的过滤策略
根据3D-RISM水的分析、PIP场分析以及文献报道的关键相互作用残基,最终确定的相互分析与基于结构的过滤步骤如下:
- 以ARG150作为关键残基(图4),进行相互作用分析,过滤出与之发生氢键相互作用的配体
- 以ARG374作为关键残基(图4),进行相互作用分析,过滤出与之发生氢键相互作用的配体
- 以ARG116,ARG292,ARG374作为关键残基(图4),进行相互作用分析,过滤出与之任意一个发生盐桥相互作用的配体
在分子对接、过滤等操作过程中,pose数量与化合物数量的变化汇总如表2所示。最终,过滤之后共有87个(608个pose)actives化合物与718个(2088个pose)decoys化合物满足全部过滤条件,如表2所示。
表2. 过滤对pose数量与化合物数量的影响
| Items | Actives | Decoys |
|---|---|---|
| unique compound | 98 | 6199 |
| ligand preparation | 222 | 6227 |
| Poses generated by docking | 3543(98) | 87961(6178) |
| Poses after ARG150 filtration | 1862 | 43473 |
| Poses after ARG374 filtration | 662 | 7190 |
| Poses after Salt-bridge filtration | 608(87) | 2088(718) |
其中括号里的数为不重复的化合物数
重新打分、数据融合与性能指标
将上一步过滤得到的actives(87个化合物,608个pose)与decoys(718个化合物,2088个pose)用Flare docking在score-fixed模式下进行打分,并用GNINA在score_only模式下打分,然后数据融合,得到LF VS-score以及数据融合打分LF_CNN_VS、CNN_VS等三种打分用于虚拟筛选性能统计分析。过滤前后的主要性能指标如表3所示。
表3. 基于结构的过滤对虚拟筛选性能的影响
| Metric | LF VSscore | LF_CNN_VS | CNN_VS | |||
|---|---|---|---|---|---|---|
| unfiltered | filtered | unfiltered | filtered | unfiltered | filtered | |
| AUC | 0.749 | 0.917 | 0.924 | 0.932 | 0.939 | 0.933 |
| adj-logAUCλ=0.1% | 9.2 | 41.4 | 35.5 | 62.2 | 42.3 | 64.2 |
| BEDROCα=321.9 | 0.008 | 0.224 | 0.314 | 0.833 | 0.494 | 0.913 |
| BEDROCα=80.5 | 0.017 | 0.399 | 0.324 | 0.760 | 0.456 | 0.800 |
| BEDROCα=20.0 | 0.090 | 0.632 | 0.506 | 0.823 | 0.588 | 0.838 |
| EF0.5% | 0 | 18.1 | 18.1 | 56.2 | 32.1 | 62.2 |
| EF1% | 1.0 | 21.4 | 18.4 | 49.0 | 28.0 | 49.0 |
| EF5% | 0.8 | 14.1 | 10.6 | 17.5 | 12.0 | 17.3 |
| EF10% | 1.9 | 8.8 | 7.2 | 8.9 | 7.5 | 8.9 |
过滤提高了LF VS-score的综合性能与早期富集性能
从LF VS-score打分的ROC曲线(图5)可以直观地看出,过滤后的曲线(洋红色)在未过滤曲线(深蓝色)的左上方,且显著地靠近左上角,因此过滤提高了LF VS-score的综合性能(overall performance)。从表1的AUC值可以定量看出,LF VS-score的AUC提高了22.4%,从过滤前的0.749提高到过滤后的0.917;而强调早期富集性能的logAUC则提高了350%,由9.2提高到41.4。
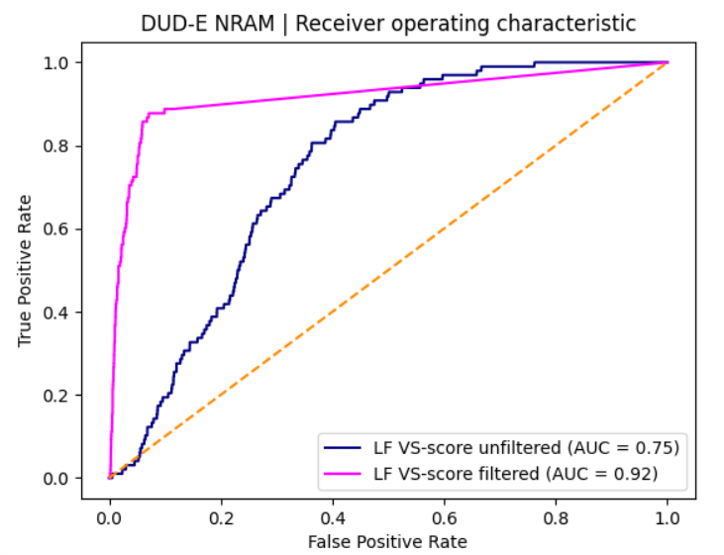
图5. LF VS-score打分的ROC曲线
从图5的ROC曲线还可以发现,在大约FPR小于0.5%的早期阶段,未过滤的ROC曲线位于对角线(随机曲线)的下方,这说明未经过滤的LF VS-score不仅没有早期富集性能,甚至富集假阳性化合物;在大约FPR小于0.5%阶段,经过过滤的LF VS-score曲线显著地靠近纵坐标,这是具有良好早期富集性能的标志。从早期富集性能指标BEDROC(α=80.5)来看,过滤使得LF VS-score的富集性能提高了22.5倍,从过滤前的0.018提高到0.399;从另一个早期富集性能指标EF1%来看,过滤使得LF VS-score的EF1%值从1.0提高到21.4,提高了20倍左右。
数据融合提高了虚拟筛选的综合性能与富集性能
CNN_VS与LF_CNN_VS是GNINA Pose打分(CNN score)分别与GNINA亲和力打分(CNN affinity)以及LF VS-score打分的数据融合打分,是一种一致性策略打分。
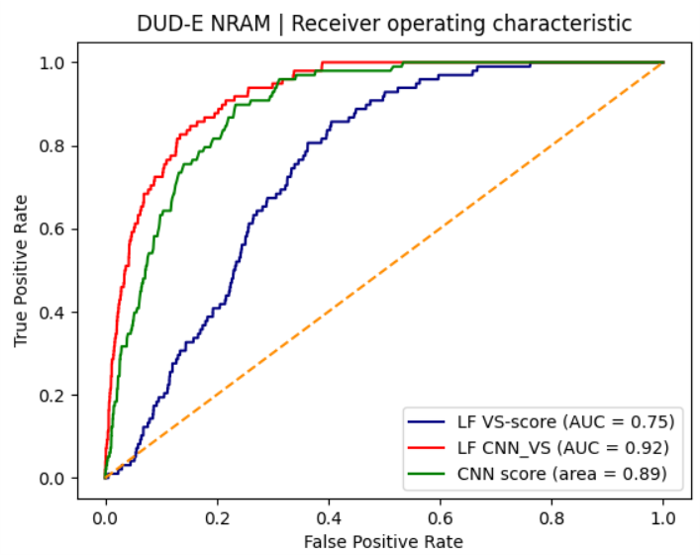
图6. LF VS-score、CNN score与LF_CNN_VS打分的ROC曲线
图6呈现了在未经过滤时LF VS-score、CNN score与LF_CNN_VS三种打分的ROC曲线。可以看出,三条曲线区分明显,而且LF_CNN_VS最靠近左上角、CNN score居中、LF VS-score在最下面,这反映了三者综合性能从高到低的顺序,与三者AUC值的排序是一致的。也就是说,LF VS-score与CNN score的数据融合策略提高了虚拟筛选综合性能。
从图6的ROC曲线还可以清晰地看出,在FPR小于0.1阶段,数据融合打分LF_CNN_VS的曲线还处于最左上方,接着是CNN score,最下方的是LF VS-score。这说明三种打分的富集性能从高到低的排序为:LF_CNN_VS,CNN score、LF VS-score。数据融合策略打分LF_CNN_VS的富集性能优于独立打分CNN score与LF VS-score。
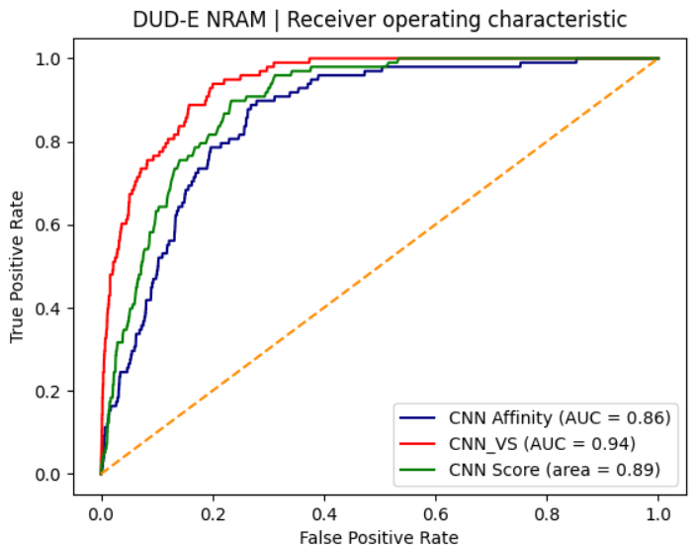
图7. CNN affinity、CNN score与CNN_VS打分的ROC曲线
图7呈现了在未经过滤时GNINA亲和力打分CNN affinity、结合模式打分CNN score以及两者的数据融合打分CNN_VS等三种打分的ROC曲线。可以看出,三条ROC曲线区分明显,而且数据融合打分CNN_VS最靠近左上角、CNN score次之、CNN affinity在最下方,这反应了三者打分综合性能从高到低的排序,也与三者AUC值的排序是一致的。再一次,数据融合打分在综合性能上优于两个独立打分,数据融合策略提高了虚拟筛选综合性能。
从图7的ROC曲线还可以清晰地看出,在FPR小于0.1的早期阶段,数据融合打分CNN_VS的曲线还是处于最左上方,接着是CNN score,最下方的是LF VS-score。这说明三种打分的富集性能从高到低的排序为:CNN_VS,CNN score、CNN affinity。数据融合策略打分CNN_VS的富集性能优于独立打分CNN score与CNN affnity。
总的来说,数据融合策略提高了flare默认打分LF VS-score与GNINA默认打分CNN Affinity的虚拟筛选综合与富集性能。
LF_CNN_VS与CNN_VS两种数据融合打分的比较
比较两种数据融合打分LF_CNN_VS与CNN_VS的ROC曲线,如图8所示,可以发现红色的CNN_VS与深蓝色LF_CNN_VS曲线非常贴近,说明两者综合性能相差不大;但是CNN_VS曲线总在LF_CNN_VS的左上方,说明CNN_VS比LF_CNN_VS具有更好的富集性能。
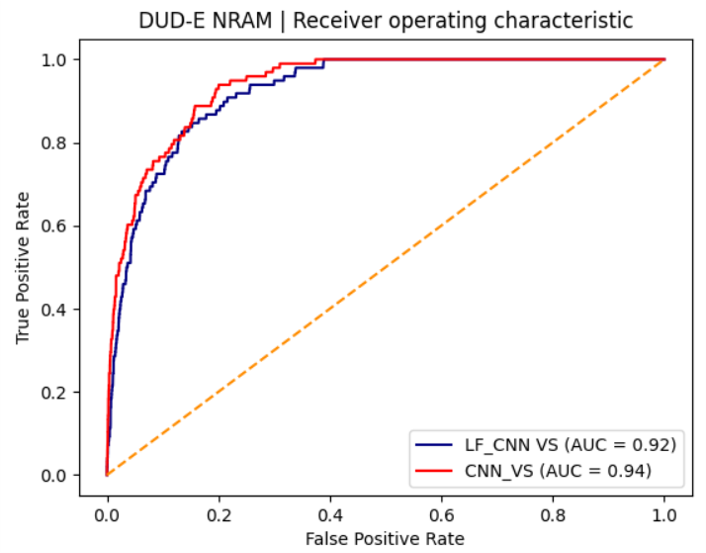
图8. 两种数据融合策略打分LF_CNN_VS与CNN_VS的ROC曲线
一方面,从早期富集性能指标BEDROCα=80.5上看,CNN_VS优于LF_CNN_VS,分别为0.456与0.326。另一方面,从早期富集性能指标EF1%上看,CNN_VS还是优于LF_CNN_VS,分别为28.0与18.4。
基于结构的过滤提高了LF_CNN_VS与CNN_VS两种数据融合打分的早期富集性能
从早期富集性能指标BEDROCα=80.5看,如图9所示,基于结构的过滤提高了LF_CNN_VS与CNN_VS的早期富集性能,BEDROC分别从0.32、0.46提高到0.76、0.80。这个性能优于Shen等人18报道的Glide SP打分性能,其BEDROCα=80.5=0.724。
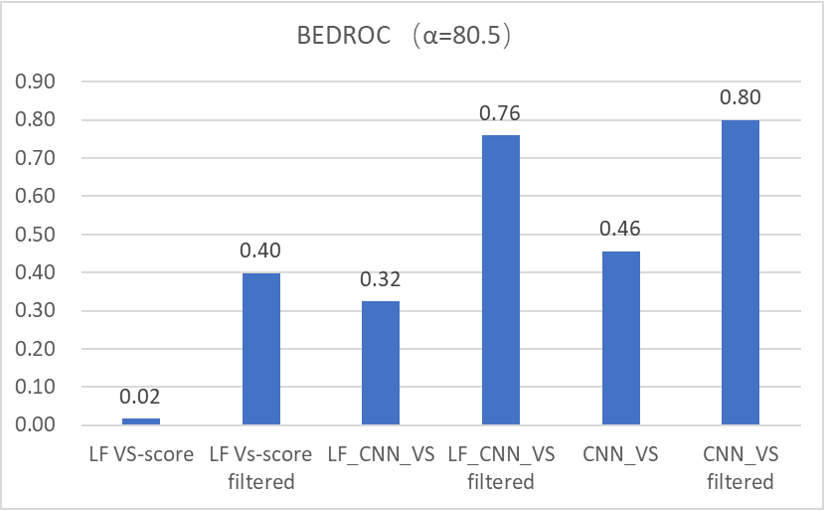
图9. 基于结构过滤前后的BEDROCα=80.5比较
从早期富集性能指标EF1%看,如图10所示,基于结构的过滤提高了LF_CNN_VS与CNN_VS的早期富集性能,EF值分别从18.4、28.0提高到49.0、49.0,与Shen等人18报道的Glide SP打分性能一样。
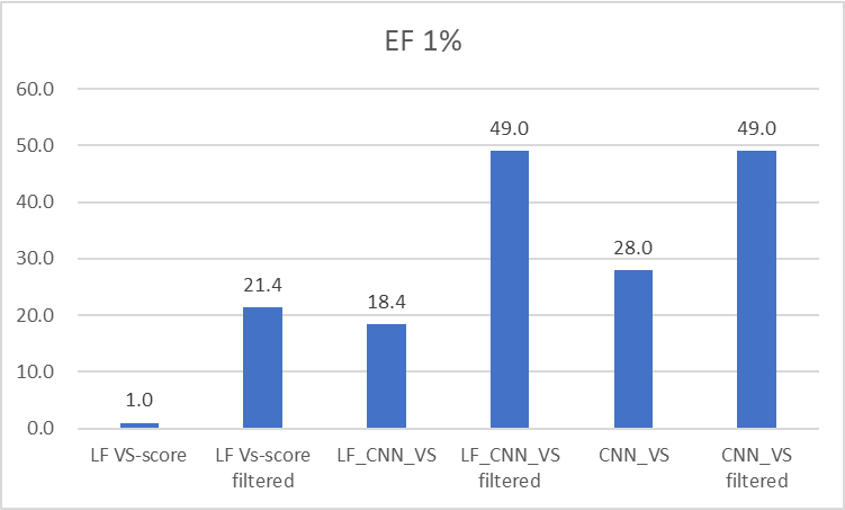
图10. 基于结构过滤前后的EF1%比较
基于结构的蛋白配体相互作用指纹(structural protein-ligand interaction fingerprint,SPLIF)是一种常用的分子对接后处理方法19。用ODDT中实现的SPLIF方法16以PDB 1B9V共晶配体为参比,对虚拟筛选结果进行Tanimoto相似性打分,虽然具ROC AUC(0.86)很高,但是不具有早期富集性能,因为在早期阶段ROC曲线位于随机曲线的下方(如图11所示),且BEDROCα=80.5=0.017,EF1%=0。从这个算例上看,基于结构的过滤优于以SPLIF代表的相互作用指纹相似性分析。
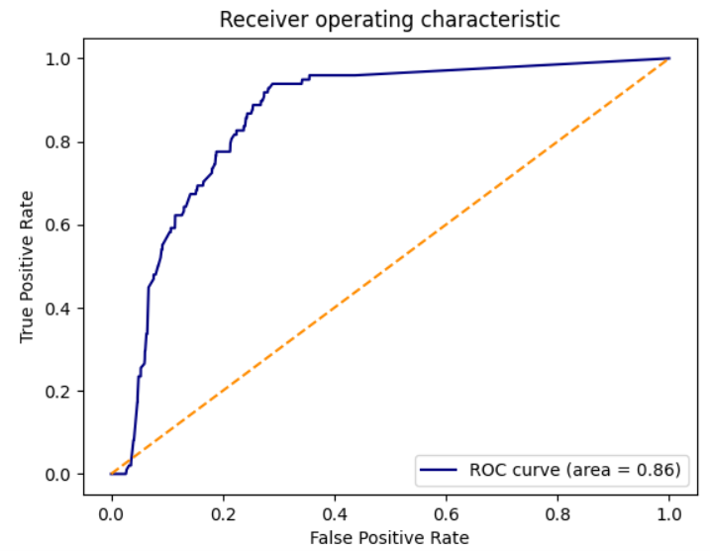
图11. SPLIT相互作用指纹相似性ROC曲线
SPLIF相似性除了作为一个独立的打分函数之外,也可以作为过滤器。以0.6作为Tanimoto系数的截断值,考察LF VS-score、LF_CNN_VS与CNN_VS的虚拟筛选性能,主要指标如表4所示、
表4. 基于SPLIF相似性指数的过滤对虚拟筛选性能的影响
| Metric | LF VSscore | LF_CNN_VS | CNN_VS | |||
|---|---|---|---|---|---|---|
| unfiltered | filtered | unfiltered | filtered | unfiltered | filtered | |
| AUC | 0.749 | 0.871 | 0.924 | 0.905 | 0.939 | 0.911 |
| adj-logAUCλ=0.1% | 9.2 | 25.0 | 35.5 | 43.0 | 42.3 | 49.2 |
| BEDROCα=321.9 | 0.008 | 0.053 | 0.314 | 0.502 | 0.494 | 0.662 |
| BEDROCα=80.5 | 0.017 | 0.129 | 0.324 | 0.468 | 0.456 | 0.585 |
| BEDROCα=20.0 | 0.090 | 0.342 | 0.506 | 0.615 | 0.588 | 0.679 |
| EF0.5% | 0 | 4.0 | 18.1 | 38.2 | 32.1 | 46.2 |
| EF1% | 1.0 | 8.2 | 18.4 | 28.6 | 28.0 | 36.7 |
| EF5% | 0.8 | 7.1 | 10.6 | 13.5 | 12.0 | 13.9 |
| EF10% | 1.9 | 6.1 | 7.2 | 8.1 | 7.5 | 8.2 |
SPLIF相似性过滤可提高虚拟筛选的早期富集性能。如表4所示,将SPLIF Tanimoto相似性指数低于0.6的pose过滤之后,LF VS-score、LF_CNN_VS与CNN_VS打分相应的BEDROCα=80.5与EF1%均得到提高。
表5. 基于结构的过滤与基于SPLIF相似性指数过滤(Tanimoto index cutoff=0.6)的富集性能比较
| Metric | LF VSscore | LF_CNN_VS | CNN_VS | |||
|---|---|---|---|---|---|---|
| Struct. | SPLIF | Struct. | SPLIF | Struct. | SPLIF | |
| BEDROCα=80.5 | 0.40 | 0.13 | 0.76 | 0.47 | 0.80 | 0.59 |
| EF1% | 21.4 | 8.2 | 49.0 | 28.6 | 49.0 | 36.7 |
Struct.: Structural filtration
与SPLIF相似性过滤(Tanimoto index cutoff=0.6)相比,如表5所示,基于结构的过滤在三种打分函数的两个早期富集性能指标BEDROCα=80.5与EF1%上均具有大幅度的优势。
总的来说,基于结构的过滤提高了LF_CNN_VS与CNN_VS两种数据融合打分的早期富集性能,并且在早期富集性能上优于以SPLIF代表的相互作用指纹方法。
结论
本文以DUD-E的NRAM为子集为例,演示了如何用Flare的相互作用分析对Lead Finder分子对接虚拟筛选结果进行基于结构的过滤来实现富集性能的提升。具体而言,首先用Flare的3D-RISM分析、PIP分析、结合文献确定相互作用的关键残基;接着用Flare对虚拟筛选结果进行相互作用分析,过滤掉不能与关键残基发生相互作用的化合物;接着对保留下来的化合物进行二次打分,进行优先级排序与性能统计。结果表明,基于结构的过滤使得虚拟筛选早期富集性能指标EF1%从LF VS-score的1.0提高到LF_CNN_VS与CNN_VS的49.0;而另一个早期富集性能指标BEDROCα=80.5从LF VS-score的0.02提高到LF_CNN_VS的0.76与CNN_VS的0.80。总的来说,基于结构的过滤提高了分子对接虚拟筛选的早期富集性能。
附件
链接:https://pan.baidu.com/e/1g2u_88myF3EIUAndMIlenQ
提取码: nram
附件内容:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 | nram ├── 1b9v_ligand.sdf ├── 1b9v_prot.grid ├── 1b9v_prot.pdb ├── dock.conf ├── 1_dock │ ├── actives_dock.sdf │ ├── all_score.csv │ ├── all_score_grouped.csv │ └── decoys_dock.sdf ├── 2_ARG150 │ ├── actives_dock.sdf │ └── decoys_dock.sdf ├── 3_ARG374 │ ├── actives_dock.sdf │ └── decoys_dock.sdf ├── 4_salt-bridge │ ├── actives_dock.sdf │ └── decoys_dock.sdf ├── 5_lf_rescore │ ├── actives_dock.sdf │ └── decoys_dock.sdf └── 6_gnina_rescore ├── actives.sdf ├── all_score.csv ├── all_score_grouped.csv └── decoys.sdf |
文献
- a)Fischer, A.; Smieško, M.; Sellner, M.; A. Lill, M. Decision Making in Structure-Based Drug Discovery: Visual Inspection of Docking Results. J. Med. Chem. 2021, 64 (5), 2489–2500. https://doi.org/10.1021/acs.jmedchem.0c02227. b)基于结构药物发现中的决策:对接结果的手工检查.墨灵格的博客. http://blog.molcalx.com.cn/2021/05/22/visual-inspection-of-docking-results.html
- Novikov, F. N.; Stroylov, V. S. Improving Performance of Docking-Based Virtual Screening by Structural Filtration. J. Mol. Model. 2010, 16 (7), 1223–1230. https://doi.org/10.1007/s00894-009-0633-8.
- Lead Finder. https://www.cresset-group.com/software/leadfinder
- Novikov, F. N.; Stroylov, V. S.; Zeifman, A. A.; Stroganov, O. V.; Kulkov, V.; Chilov, G. G. Lead Finder Docking and Virtual Screening Evaluation with Astex and DUD Test Sets. J Comput Aided Mol Des 2012, 26 (6), 725–735. https://doi.org/10.1007/s10822-012-9549-y.
- Stroganov, O. V; Novikov, F. N.; Stroylov, V. S.; Kulkov, V.; Chilov, G. G. Lead Finder: An Approach To Improve Accuracy of Protein−Ligand Docking, Binding Energy Estimation, and Virtual Screening. J Chem Inf Model 2008, 48 (12), 2371–2385. https://doi.org/10.1021/ci800166p.
- Gushchina, I. V.; Polenova, A. M.; Suplatov, D. A.; Švedas, V. K.; Nilov, D. K. VsFilt: A Tool to Improve Virtual Screening by Structural Filtration of Docking Poses. J. Chem. Inf. Model. 2020, 60 (8), 3692–3696. https://doi.org/10.1021/acs.jcim.0c00303.
- Flare V7. https://www.cresset-group.com/software/flare
- 肖高铿. 分子对接后处理——与指定残基发生特定相互作用配体的过滤. 墨灵格的博客. 2021-11-02. http://blog.molcalx.com.cn/2021/11/02/docking-post-filtering.html
- 肖高铿. 用基于结构的过滤提高分子对接虚拟筛选的效率. 墨灵格的博客. 2023-09-23. http://blog.molcalx.com.cn/2023/09/24/post-filtering-for-docking-vs.html
- Morgan Morris, Jessica Plescia, Sílvia Tuset, Andy Smith, Stuart Firth-Clark. 对最有前景的虚拟筛选苗头化合物进行优先级排序. 墨灵格的博客. 2023-10-07. http://blog.molcalx.com.cn/2023/10/07/prioritizing-the-virtual-screening-hits.html
- DUD-E NRAM subset. https://dude.docking.org/targets/nram
- Sunseri, J., & Koes, D. R. (2021). Virtual Screening with Gnina 1.0. Molecules, 26(23), 7369. https://doi.org/10.3390/molecules26237369
- GNINA 1.0, https://github.com/gnina
- E. L. Mehler, The Lorentz-Debye-Sack theory and dielectric screening of electrostatic effects in proteins and nucleic acids, in Molecular Electrostatic Potentials: Concepts and Applications, Theoretical and Computational Chemistry Vol. 3, 1996
- 肖高铿. 用数据融合增强分子对接的虚拟筛选性能. 墨灵格的博客. 2023-07-15. http://blog.molcalx.com.cn/2023/08/31/boosting-virtual-screening-enrichments-with-data-fusion.html
- a)Wójcikowski, M., Zielenkiewicz, P., & Siedlecki, P. Open Drug Discovery Toolkit (ODDT): a new open-source player in the drug discovery field. Journal of Cheminformatics, 2015, 7(1), 26. https://doi.org/10.1186/s13321-015-0078-2. b)https://github.com/oddt
- Yoshidome, T., Ikeguchi, M., & Ohta, M. Comprehensive 3D-RISM analysis of the hydration of small molecule binding sites in ligand-free protein structures. Journal of Computational Chemistry, 2020, 41(28), 2406–2419. https://doi.org/10.1002/jcc.26406
- Shen, C.; Hu, Y.; Wang, Z.; Zhang, X.; Pang, J.; Wang, G.; Zhong, H.; Xu, L.; Cao, D.; Hou, T. Beware of the Generic Machine Learning-Based Scoring Functions in Structure-Based Virtual Screening. 2020, 00 (April), 1–22. https://doi.org/10.1093/bib/bbaa070.
- Da, C., & Kireev, D. Structural Protein–Ligand Interaction Fingerprints (SPLIF) for Structure-Based Virtual Screening: Method and Benchmark Study. Journal of Chemical Information and Modeling, 2014, 54(9), 2555–2561. https://doi.org/10.1021/ci500319f
获取软件试用与技术合作,请联系我们
想在自己的项目上使用基于结构的过滤或进行项目合作机会,请联系我们。



