用基于结构的过滤提高分子对接虚拟筛选的效率
摘要:本文以DUD-E PDE5A子集为例,演示了如何用Flare的相互作用分析来对Uni-Dock结果进行基于结构的过滤,在不损失富集性能的前提下,在GNINA的CNN模型再打分阶段节省不止90%的GPU计算资源,提高了虚拟筛选的整体速度与效率。
肖高铿/2023-09-23
前言
GPU-Vina 2.01与Uni-Dock2等GPU加速的AutoDock Vina 1.23分子对接衍生软件在保留虚拟筛选性能的前提下使得计算速度得以极大提升。比之单核CPU计算的AutoDock Vina,使用一张现在主流GPU计算卡的Uni-Dock速度提升了1000多倍2。为了提高AutoDock Vina的虚拟筛选性能,Koes课题组4-6开发的另一个AutoDock Vina变体GNINA,它内置了深度学习CNN模型的结合亲和力与结合模式打分函数。在DUD-E数据集上,GNINA采用默认的CNN模型affinity打分函数,富集性能(Median EF1%=15.5)比Vina打分(Median EF1%=7.1)提高了大约1倍。虽然GNINA的富集性能高,但其计算速度无法与Uni-Dock相比,因此用GNINA基于CNN模型的打分函数对Uni-Dock的对接结果再打分以兼具速度与富集性能是一个自然而然的想法。我们7前期针对DUD-E激酶子集8(26个靶标)的虚拟筛选测试证明这种组合使用是可行的,用GNINA的默认CNN亲和力打分函数对Uni-Dock的fast模式虚拟筛选结果再打分,结果发现采用GNINA默认CNN模型亲和力打分的富集性能(Median EF1%=18.8)比Uni-Dock Vina打分(Median EF1%=9.3)的高了约1倍,而相较GNINA对接(Median EF1%=20.5,根据文献6统计)来说富集性能损失非常低,如图1所示。
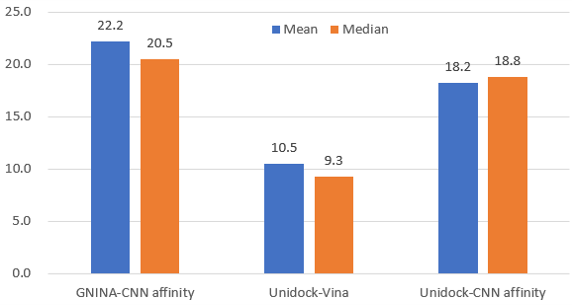
图1. 几种方法在DUD-E Kinase子集上的富集性能(EF1%)。左:GNINA采用默认CNN affinity打分函数;中:Uni-Dock采用Fast模式与Vina打分函数;右:Uni-Dock采用Fast模式用GNINA的CNN affinity打分函数再打分
然而,采用深度学习CNN模型的打分函数也是需要GPU计算资源,分子对接的再打分后处理与GPU加速分子对接计算对GPU计算资源消耗几乎一样,这使得高精度的CNN模型打分函数并不高效。这主要有两个原因:1)化合物结构准备过程使得实际处理数量扩增,在分子对接虚拟筛选的前期配体结构准备中,通常需要进行互变异构体枚举、质子化状态枚举、不明确的立体化学中心枚举等操作,这使得需要实际进行对接计算化合物数量急剧增加;2)Uni-Dock的fast模式对接计算为每个输入生成了9个结合模式,这使得下一步的亲和力计算需要处理大约10倍输入分子数量的结合模式。
Fishcer等人9对文献的调研以及对93位来自学术与制药工业的专家问卷调查表明,人们基本达成共识:成功的分子对接虚拟筛选需要一定的人工后处理。Novikov与Stroylov10提出的基于结构的过滤(Structural filtration)是一种创新的分子对接结果后处理方式,结构过滤器由一组蛋白特异性相互作用定义,这些相互作用(a)在特定蛋白与其结合配体的结构上保守,(b)可被视为在蛋白-配体结合中起关键作用。Novikov与Stroylov10为此开发了相应的结构过滤器,并将其应用于Lead Finder分子对接软件12-14获得的虚拟筛选结果。他们在一组10种不同的蛋白靶标上进行了评估,结果表明,结构过滤的应用使富集因子得到了显著改善,根据蛋白质靶标的不同,富集因子从几倍到几百倍不等。Gushchina等人开发的vsFilt11是一种基于结构的对接结果后处理网页应用,显著提高了Lead Finder分子对接虚拟筛选的性能。Flare15内置的蛋白-配体相互分析工具Contact analysis可以帮助我们进行此类的基于结构的后处理,在之前的博客中也分享过如何对AutoDock Vina的结果进行过滤以提高虚拟筛选的性能16。本文以DUD-E PDE5A子集17为例,采用如图2所示虚拟筛选与后处理流程,演示了如何用Flare的相互作用分析来对Uni-Dock结果进行基于结构的过滤,在不损失富集性能的前提下,实现了为GNINA的CNN模型再打分后处理节省不止90%的GPU计算资源,提高了深度学习打分函数的效率并进一步提高虚拟筛选的整体速度。
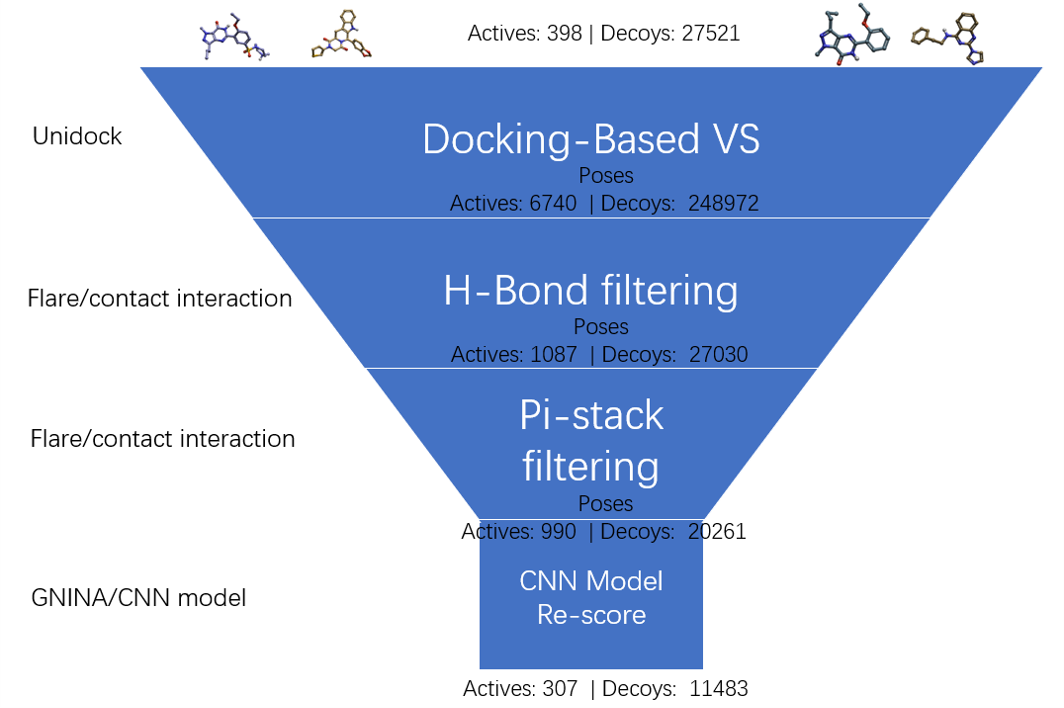
图2. 虚拟筛选流程图,包括分子对接虚拟筛选、基于结构的过滤与重新打分
材料与方法
数据库分子的准备
DUD-E PDE5A子集17里预先准备好的SDF格式actives(actives_final.sdf)与decoys(decoys_final.sdf)结构直接用于虚拟筛选,不进行额外的准备。这么做是出于与文献报道方法保持一致以便性能比较的目的。鉴于该数据集的部分化合物存在电荷与键级的错误,因此建议在实际的研究中,应该重新进行准备,比如用XEDTools对结构重新归属formal charge以便修复错误。在本文中,所有的分子用Meeko (Version 0.4)18的python脚本(mk_prepare_ligand.py,为了方便处理,略有修改)转化为PDBQT格式,并用于后续的Uni-Dock计算。
Actives分子的准备:
1 2 3 4 | mk_prepare_ligand.py -i actives_final.sdf \ --multimol_outdir actives \ --multimol_prefix actives \ --rigid_macrocycles |
Decoys分子的准备:
1 2 3 4 | mk_prepare_ligand.py -i decoys_final.sdf \ --multimol_outdir decoys \ --multimol_prefix decoys \ --rigid_macrocycles |
准备完毕,生成索引列表以便Uni-Dock进行对接计算:
Actives分子的索引列表准备:
1 | ls actives/*.pdbqt >> actives.index |
Decoys分子的索引列表准备:
1 | ls decoys/*.pdbqt >> decoys.index |
蛋白结构的准备
用Flare下载Virgra-PDE5A共晶结构(PDB 1UDT),并用Protein Prep工具进行蛋白结构准备,仔细检查结构以确保配体结构正确、相互作用合理,删除全部的水,仅保留A链的蛋白部分与配体,然后分别导出蛋白结构(1udt_prot.pdb)与配体结构(1udt_ligand.sdf)。其中蛋白结构用MGLTools (Version 1.5.7)19的prepare_receptor4.py转化为PDBQT格式(1udt_prot.pdbqt)备用。
Uni-Dock分子对接虚拟筛选
用前一步得到共晶配体的质心来定义格点计算的中心,准备对接配置文件(dock.conf)如下:
1 2 3 4 5 6 7 | receptor = 1udt_prot.pdbqt center_x = 1.548 center_y = 68.184 center_z = 83.831 size_x = 30 size_y = 30 size_z = 30 |
然后用Uni-Dock (Version 1.0)20进行分子对接虚拟筛选。Actives分子的对接计算:
1 2 3 4 | unidock --ligand_index actives.index \ --config dock.conf \ --dir actives \ --search_mode fast |
Decoys分子的对接计算:
1 2 3 4 | unidock --ligand_index decoys.index \ --config dock.conf \ --dir decoys \ --search_mode fast |
计算完毕,用Meeko18的mk_export.py脚本(为了方便处理,略有修改)导出为SDF文件,最后得到SDF格式的对接结果文件actives_dock.sdf与decoys_dock.sdf,参见附件。在SDF文件的性质部分里包含了Vina打分值,并用于后续的虚拟筛选性能指标计算。
Flare基于结构的过滤
如前文5.3小节的图6所示16,我们用Flare分析过PDE5抑制剂Viagra与Cialis的共晶结构PDB 1UDT与1UDU,我们可以看到与两个药物同时发生相互作用的残基(金黄色的CPK),其中包括PDB 1UDT中的GLN817与PHE820。如图3所示,GLN817与Viagra的芳香母核上的酰胺片段形成氢键互补,PHE820与该芳香双环母核发生Pi-Pi芳香相互作用。在本文中,以与这两个残基的相互作用作为条件进行过滤。
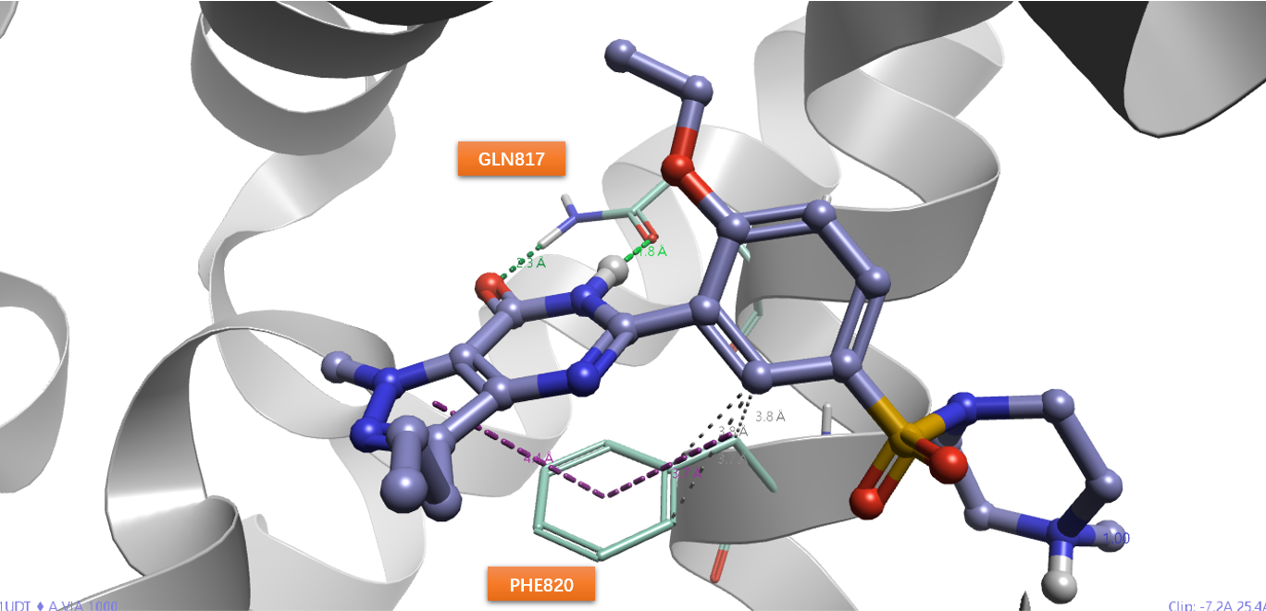
图3. Virgra-PDE5A共晶结构(PDB 1UDT)的两个关键相互作用
Flare除了提供可视化分析之外,还提供了Python API通过cresset.flare.contacts来分析各种相互作用,以帮你从分子对接结果中过滤出满足条件的分子或pose。这些相互作用包括:
- aromatic_aromatic
- cation_pi
- h_bonds
- halogen_bonds
- steric_clashes
- sulfur_lone_pair
- salt_bridge
将上述的对接结果SDF文件导入到Flare之后,点击Flare | Python | Python Interpreter,打开Python解释器,可以通过Python对Uni-Dock的结果进行过滤。
图4. Flare的Python解释器
与GLN817发生氢键相互作用分子的过滤,所用Python代码如图5所示。
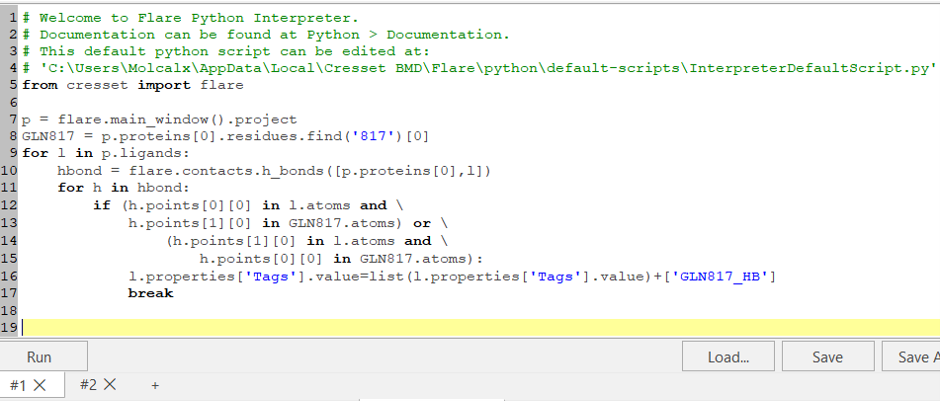
图5. 与GLN817发生氢键相互作用的过滤
与PHE820发生Pi-Pi堆积相互作用的过滤,所用Python代码如图6所示。
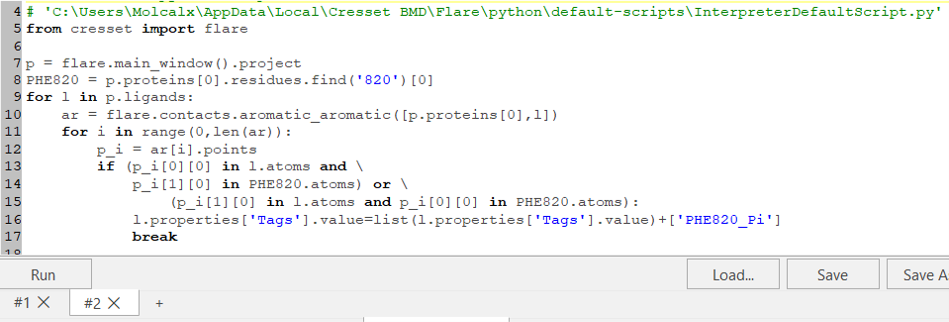
图6. 与PHE820发生Pi-Pi堆积相互作用的过滤
在Python解释器上点击Run按钮,可执行预定义的分析,并在分子表单的Tag列添加标签。在Flare工具栏点击Filters,选择对Tag进行过滤,用AND逻辑运算,仅显示与GLN817、PHE820同时发生相互作用的对接结果,如图7所示。
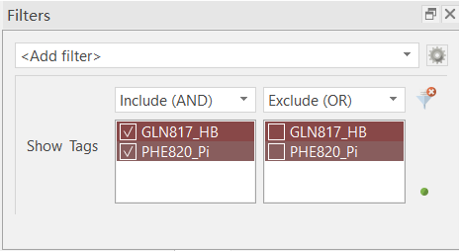
图7. 过滤器的使用
将过滤后同时满足两个相互作用条件的actives与decoys对接结果分别保存为actives_dock_filtered.sdf与decoys_dock_filtered.sdf,参见附件。
为了方便操作,你还可以用命令行来操作上述过滤步骤。Flare的pyflare可以让您在命令行独立运行python代码,修改上面代码可以得到通用的过滤脚本,并直接输出满足条件的结果。
需要注意的是:Uni-Dock与Vina一样,其输出结果里的极性氢原子是随机取向,与氢键相互作用取向无关。因此,在进行氢键相互作用过滤之前要进行极性氢优化或用与Vina一样的无方向氢键方式来评估氢键。在本文中,优化与不优化氢凑巧差异不大,因此在这里没有进一步对氢进行处理。
用GNINA重新打分
用GNINA对未经过滤的actives对接结果进行重新打分:
1 | gnina --config dock.conf -l actives_dock.sdf -o actives.sdf --score_only |
用GNINA对未经过滤的decoys对接结果进行重新打分:
1 | gnina --config dock.conf -l decoys_dock.sdf -o decoys.sdf --score_only |
用GNINA对经过滤的actives对接结果进行重新打分:
1 | gnina --config dock.conf -l actives_dock_filtered.sdf -o actives_filtered.sdf --score_only |
用GNINA对经过滤的decoys对接结果进行重新打分:
1 | gnina --config dock.conf -l decoys_dock_filtered.sdf -o decoys_filtered.sdf --score_only |
GNINA打分之后,在sdf文件里包含了3个新的打分值:
- CNNscore: GNINA的深度学习pose打分值
- CNNaffinity:GNINA的深度学习结合亲和力预测值
- CNN_VS:CNNscore x CNNaffinity
在本文中,用CNN_VS来计算虚拟筛选性能指标。
数据处理与虚拟筛选性能评估
鉴于从DUD-E下载的actives与decoys数据集进行了互变异构体与质子化状态枚举处理,导致一个化合物会以不同的形式多次出现、并可能被虚拟筛选多次命中,因此对虚拟筛选结果按化合物名称进行去重,同名化合物仅以其中打分最高那个来代表,所有的性能评估指标都在去重的基础上进行计算。
在虚拟筛选过程中,有可能有的化合物处理失败而没有出现在结果文件里,为了保证性能评估的结果与其它文献报道的具有可比性,将缺失的化合物人为地分配一个打分值0然后再进行性能评估。
用AUC、logAUC、三种α(321.9,80.5与20.0)值的BEDROC、富集因子(top 0.5%,1%,5%与10%)等指标来评估虚拟筛选的性能,具体的计算方法参见前文21。
结果
数据处理与主要性能指标
在本次计算中,仅有一个decoys分子因为键级与电荷的原因而对接失败没有结果,鉴于1个分子的差异对结果统计没有影响,因此之后的统计过程中忽略这个分子。对接之后的结果用python的pandas软件包聚合函数groupby对化合物分值进行处理:同一个化合物的互变异构体、不同质子化状态仅保留打分最优的那个作为该化合物的打分值。由于氢键与芳香相互作用过滤导致部分actives与decoys分子没出现在最终结果文件里,这部分被过滤掉的化合物各项打分值用0表示。
表1、DUD-E PDE5A数据集的化合物统计
| PDE5A数据集 | 化合物数 | 异构体数 | 生成的Pose数 | 过滤后的Pose数 |
|---|---|---|---|---|
| actives | 398 | 706 | 6740 | 990 |
| decoys | 27521 | 27826 | 248972 | 20261 |
虚拟筛选,过滤与GNINA重新打分等各个阶段的化合物(Pose)数量变化情况汇总于表1与图2,主要虚拟筛选性能指标见表2。
表2. 三种方法的主要虚拟筛选性能指标
| 指标 | Unidock-Vina unfiltered | CNN_VS unfiltered | CNN_VS filtered |
|---|---|---|---|
| AUC | 0.671 | 0.823 | 0.767 |
| logAUCλ=0.1% | 29.8 | 43.3 | 41.6 |
| adjusted-logAUCλ=0.1% | 15.3 | 28.9 | 27.2 |
| BEDROCα=321.9 | 0.227 | 0.406 | 0.439 |
| BEDROCα=80.5 | 0.199 | 0.331 | 0.329 |
| BEDROCα=20.0 | 0.258 | 0.398 | 0.358 |
| EF0.5% | 16.0 | 31.6 | 31.1 |
| EF1% | 12.0 | 22.5 | 21.8 |
| EF5% | 5.3 | 7.6 | 7.4 |
| EF10% | 3.3 | 5.0 | 5.0 |
CNN_VS再打分提高了Uni-Dock的综合性能与早期富集能力
比较未经过滤的对接结果,如表2所示,使用CNN_VS作为打分函数之后,ROC AUC从用Vina的0.671提高到CNN_VS的0.823,这说明CNN_VS打分提高了Uni-Dock的综合性能(Overall performance)。综合性能的提高还可从图8的ROC曲线上直观地看到:深蓝色的CNN_VS ROC曲线位于红色Vina ROC曲线的左上方。
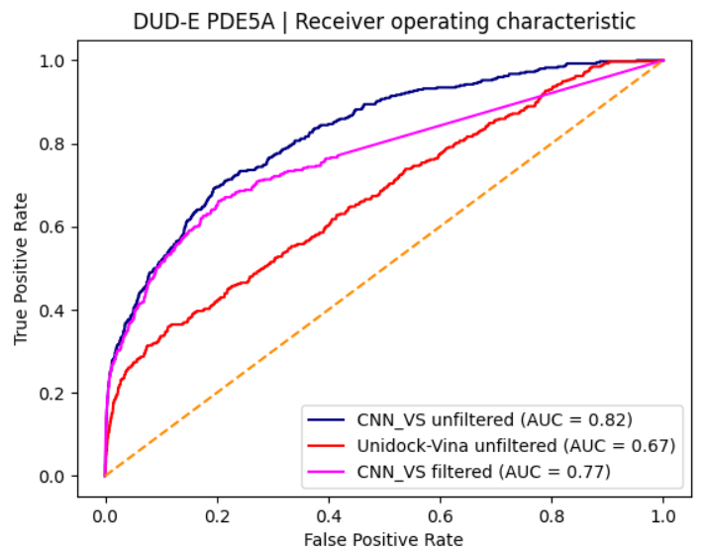
图8. 不同方法的ROC曲线
然而,我们对早期富集能力更感兴趣,文献里常用BEDROCα=80.5与EF1%来表示早期富集性能。如表2所示,CNN_VS比Vina打分的BEDROCα=80.5提高了66.3%,分别为0.331与0.199;与此同时,CNN_VS比Vina打分的EF1%提高了87.5%,分别为22.5与12.0。总的来说,在BEDROCα=80.5与EF1%表示的早期富集能力上,CNN_VS全面优于Vina,CNN_VS再打分提高了Uni-Dock的早期富集能力。
基于结构的过滤虽然降低了CNN_VS的综合性能,但不影响早期富集能力
比较过滤前后的ROC AUC,如表2所示,可以发现CNN_VS的AUC从过滤之前的0.823降低到过滤之后的0.767,这说明虚拟筛选的综合性能降低。这是因为92个actives化合物的pose不能与ASN817的末端酰胺发生氢键相互作用或没有与PHE820发生Pi-Pi堆积相互作用,导致这些actives化合物被过滤,如图2所示,从起始的398个开始,到最后得到307个。另外,有58.2%(16038个)的decoys化合物没有满足氢键与Pi-Pi堆积相互作用而被过滤。被过滤的分子打分值设为常数0,体现在图8的ROC曲线上,在大约FPR从0.4开始,过滤的CNN_VS(品红色)ROC曲线变直。
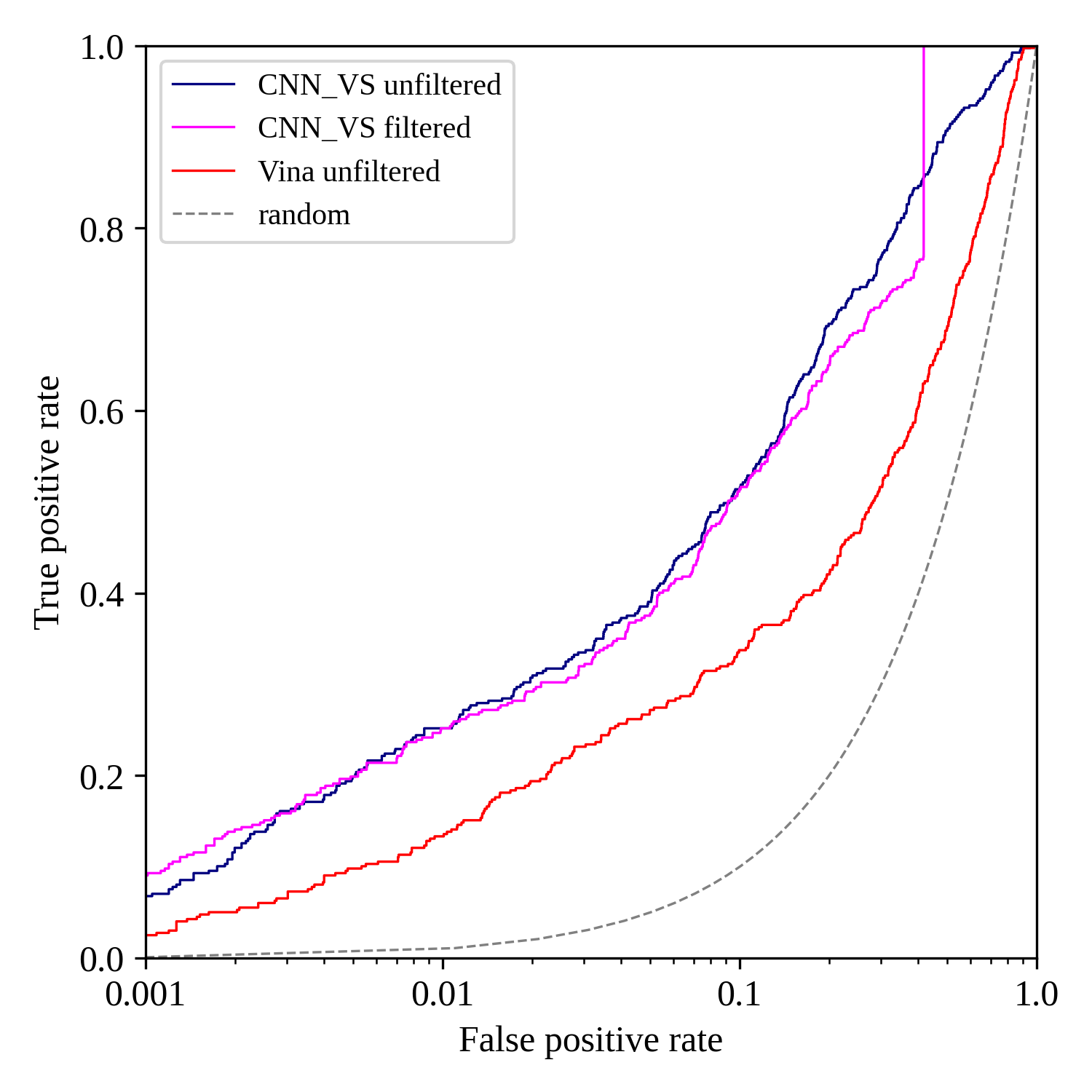
图9. 不同方法的半对数ROC曲线
比较表2的过滤前后CNN_VS的早期富集性能指标BEDROCα=80.5,可以发现性能基本没变,分别为0.331与0.329;同时,过滤前后CNN_VS的早期富集性能的另一个指标EF1%降低非常小,分别为22.5与21.8。这种难以区分的早期富集性能也反应在ROC曲线上,如图8所示,未过滤的CNN_VS(深蓝色)ROC曲线与过滤过的CNN_VS(品红色)ROC曲线早期阶段几乎重合在一起。半对数ROC曲线可以对早期性能加以区分,如图9所示,在FPR=[0.001,0.004]的区间内,过滤后的CNN_VS(品红色)曲线明显位于未过滤的CNN_VS(深蓝色)曲线上方,这说明,在这个非常早期的区间,过滤提高了CNN_VS的富集性能。
在真实的应用中,仅将打分靠前的少数化合物进一步用于实验活性测试,因此比较从不同方法打分靠前X化合物里命中的活性化合物数是另一个直观的性能比较方法。如表3所示,CNN_VS打分比Vina在打分靠前的50个化合物明显命中更多的阳性化合物,而过滤之后的CNN_VS比过滤之前的CNN_VS略有优势,但是相差不大。
表3. 三种方法打分靠前列表里命中actives化合物的数量
| Top X | Unidock-Vina unfiltered | CNN_VS unfiltered | CNN_VS filtered |
|---|---|---|---|
| 10 | 2 | 6 | 8 |
| 20 | 6 | 10 | 11 |
| 30 | 9 | 15 | 16 |
| 40 | 10 | 18 | 23 |
| 50 | 15 | 23 | 29 |
总的来说,在DUD-E PDE5A这个算例上,虽然基于结构的过滤降低了CNN_VS的综合性能,ROC AUC从过滤之前的0.823降低到过滤之后的0.767;但是基于结构的过滤对BEDROCα=80.5与EF1%表示的早期富集性能几乎没有影响,甚至还提高了更早期的富集性能。
基于结构的过滤:在不降低富集性能的前提下,更高效的GPU资源利用,更低的计算成本
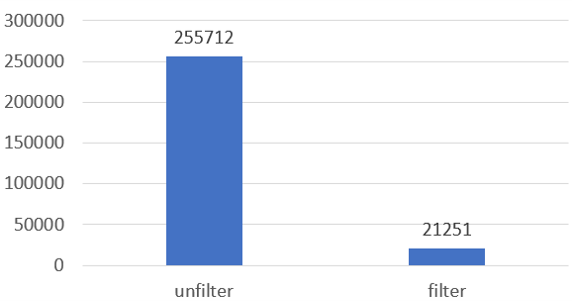
图10. 过滤前后Pose数量的比较
如图2与表1所示,DUDE PDE5A数据集包含27919个化合物(398 actives,27521 decoys);经过结构准备与分子对接,共生成255712个pose(6740 actives,248972 decoys);经过两步的基于结构过滤,大约91.7%的pose被过滤掉,仅剩余8.3%(21251个)的pose需要用GNINA调用GPU资源进行打分,图10。也就是说,基于结构的过滤节省了GNINA重新打分的92% GPU计算资源,而保持早期富集性能不变,这意味着更高的效率、更低的计算成本。
基于结构过滤的优缺点
过滤的计算量非常小,非常高效、快速,几乎没有成本。过滤的前提是需要知道过滤什么,也就是你需要一个hypothesis,这也成为该方法的缺陷,因为这不够“傻瓜式智能”。
结论
本文以DUD-E PDE5A子集为例,演示了如何用Flare的相互作用分析来对Uni-Dock结果进行基于结构的过滤,在不损失富集性能的前提下,在GNINA的CNN模型再打分阶段节省不止90%的GPU计算资源,提高了虚拟筛选的整体速度与效率。
附件
附件内容:
1 2 3 4 5 6 7 8 9 10 11 | unidock_post_filtering/ ├── 1udt_ligand.sdf ├── 1udt_prot.pdb ├── 1udt_prot.pdbqt ├── actives_dock.sdf ├── actives_filtered.sdf ├── actives.sdf ├── decoys_dock.sdf ├── decoys_filtered.sdf ├── decoys.sdf └── dock.conf |
链接: unidock_post_filtering.tar.gz
提取码: it4m
文献
- Ding, J., Tang, S., Mei, Z., Wang, L., Huang, Q., Hu, H., Ling, M., & Wu, J. (2022). Vina-GPU 2.0: Further Accelerating AutoDock Vina and Its Derivatives with Graphics Processing Units. Journal of Chemical Information and Modeling. https://doi.org/10.1021/acs.jcim.2c01504
- Yu, Y., Cai, C., Wang, J., Bo, Z., Zhu, Z., & Zheng, H. (2023). Uni-Dock: GPU-Accelerated Docking Enables Ultralarge Virtual Screening. Journal of Chemical Theory and Computation, 19(11), 3336–3345. https://doi.org/10.1021/acs.jctc.2c01145
- Eberhardt, J., Santos-Martins, D., Tillack, A. F., & Forli, S. (2021). AutoDock Vina 1.2.0: New Docking Methods, Expanded Force Field, and Python Bindings. Journal of Chemical Information and Modeling, 61(8), 3891–3898. https://doi.org/10.1021/acs.jcim.1c00203
- Ragoza, M.; Hochuli, J.; Idrobo, E.; Sunseri, J.; Koes, D. R. Protein–Ligand Scoring with Convolutional Neural Networks. J Chem Inf Model 2017, 57 (4), 942–957. https://doi.org/10.1021/acs.jcim.6b00740.
- McNutt, A. T.; Francoeur, P.; Aggarwal, R.; Masuda, T.; Meli, R.; Ragoza, M.; Sunseri, J.; Koes, D. R. GNINA 1.0: Molecular Docking with Deep Learning. J Cheminform 2021, 13 (1), 43. https://doi.org/10.1186/s13321-021-00522-2.
- Sunseri, J.; Koes, D. R. Virtual Screening with Gnina 1.0. Molecules 2021, 26 (23), 7369. https://doi.org/10.3390/molecules26237369.
- 肖高铿,徐锡明. 未公开数据
- DUD-E Kinase subset. https://dude.docking.org/subsets/kinase
- a)Fischer, A.; Smieško, M.; Sellner, M.; A. Lill, M. Decision Making in Structure-Based Drug Discovery: Visual Inspection of Docking Results. J. Med. Chem. 2021, 64 (5), 2489–2500. https://doi.org/10.1021/acs.jmedchem.0c02227. b)基于结构药物发现中的决策:对接结果的手工检查.墨灵格的博客. http://blog.molcalx.com.cn/2021/05/22/visual-inspection-of-docking-results.html
- Novikov, F. N.; Stroylov, V. S. Improving Performance of Docking-Based Virtual Screening by Structural Filtration. J. Mol. Model. 2010, 16 (7), 1223–1230. https://doi.org/10.1007/s00894-009-0633-8.
- Gushchina, I. V.; Polenova, A. M.; Suplatov, D. A.; Švedas, V. K.; Nilov, D. K. VsFilt: A Tool to Improve Virtual Screening by Structural Filtration of Docking Poses. J. Chem. Inf. Model. 2020, 60 (8), 3692–3696. https://doi.org/10.1021/acs.jcim.0c00303.
- Lead Finder. https://www.cresset-group.com/software/leadfinder
- Novikov, F. N.; Stroylov, V. S.; Zeifman, A. A.; Stroganov, O. V.; Kulkov, V.; Chilov, G. G. Lead Finder Docking and Virtual Screening Evaluation with Astex and DUD Test Sets. J Comput Aided Mol Des 2012, 26 (6), 725–735. https://doi.org/10.1007/s10822-012-9549-y.
- Stroganov, O. V; Novikov, F. N.; Stroylov, V. S.; Kulkov, V.; Chilov, G. G. Lead Finder: An Approach To Improve Accuracy of Protein−Ligand Docking, Binding Energy Estimation, and Virtual Screening. J Chem Inf Model 2008, 48 (12), 2371–2385. https://doi.org/10.1021/ci800166p.
- Flare V7. https://www.cresset-group.com/software/flare
- 肖高铿. 分子对接后处理——与指定残基发生特定相互作用配体的过滤. 墨灵格的博客. 2021-11-02 http://blog.molcalx.com.cn/2021/11/02/docking-post-filtering.html
- DUD-E PDE5A subset. https://dude.docking.org/targets/pde5a
- Meeko (Version 0.4). https://github.com/forlilab/Meeko
- MGLTOOLS (Version 1.5.7). https://ccsb.scripps.edu/mgltools
- Uni-Dock (Version 1.0). 2023-06-21. https://github.com/dptech-corp/Uni-Dock
- 肖高铿. 如何进行虚拟筛选的方法学验证.墨灵格的博客. 2016-09-22. http://blog.molcalx.com.cn/2016/09/22/virtual-screening-methodology-validation.html
获取软件试用与技术合作,请联系我们
想在自己的项目上使用基于结构的过滤或进行项目合作机会,请联系我们。



