如何进行虚拟筛选的方法学验证
摘要:本文主要讨论了如何评价虚拟筛选方法的性能,如何绘制ROC曲线、计算命中率、富集因子(EF)、ROC曲线下面积(ROC AUC)、logAUC与BEDROC等等。还讨论了为什么要选择合理的数据集进行方法学验证,并汇总了文献报道的流行软件的虚拟筛选性能指标。
肖高铿/2016-09-22
肖高铿/2022-03-04 汇总文献报道的流行软件性能指标
1. 背景
虚拟筛选的终极目的是将活性化合物从海量的数据库中尽可能地富集出来。在大规模虚拟筛选前需要考察一个虚拟筛选方法的性能:只有显著有效的方法我们才敢将之进一步用于大规模虚拟筛选。评估虚拟筛选方法性能常用回溯性验证:收集一个靶点的活性化合物与decoy化合物,进行虚拟筛选,然后用多种指标评估不同方法的性能。比较常用的指标有:ROC曲线(Receiver Operating Characteristic Curve, ROC Curve)、半对数ROC曲线、ROC曲线下面积(ROC AUC)、logAUC、BEDROC、命中率(hit rate)、富集因子(Enrichment Factor,EF)等等。本文的目的是为了方便使用,将些指标的计算方法进行汇总。
2. 常用的虚拟筛选性能评价指标
2.1 ROC曲线与ROC AUC
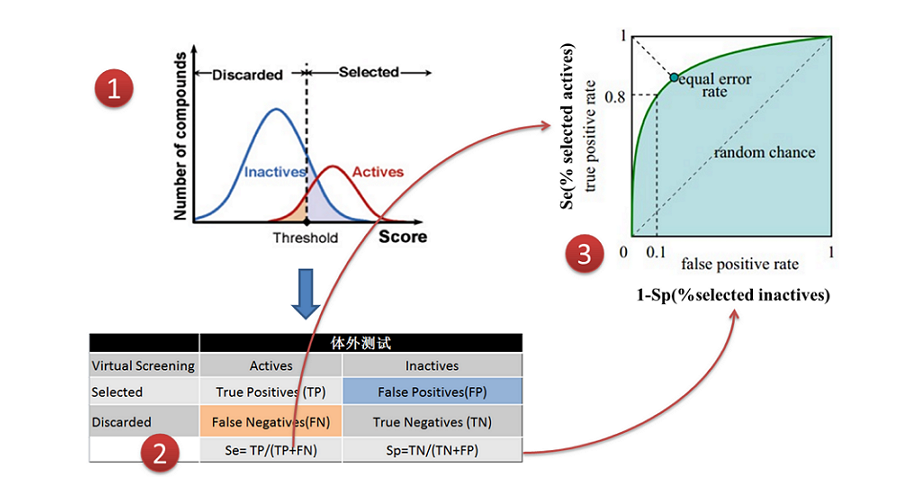
与高效液相分离性质相近的两个组份——目标化合物与杂质——类似,虚拟筛选要分离的两个组份是:活性(actives)与非活性(inactives,decoys)化合物(如图1所示)。分子对接、药效团筛、分子形状与静电技术以及2D指纹图谱等等之类的虚拟筛选技术就像是柱子、虚拟筛选的参数就像是液相分离的条件,我们的目标是要使用合适的条件将活性化合物与非活性化合物这两个组份充分分离。无论是那种虚拟筛选技术,都是对化合物进行打分,打分值就像保留时间,通过对打分值的统计,我们可以评估一个虚拟筛选方法是否可以有效的将活性化合物从海量的数据库中分离、富集起来。
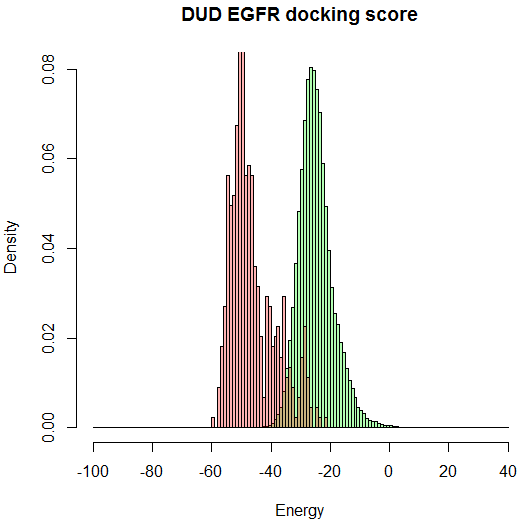
图1.虚拟筛选与高效液相分离性质相近两个组份–目标化合物与杂质–类似,只不过虚拟筛选要分离的两个组份是:活性(actives,红色)与非活性(inactives/decoys,绿色)化合物。
ROC曲线绘制原理如图2所示。我们根据虚拟筛选的打分值来设定截断值,将优于截断值的化合物选出用于进一步活性测试,而将比截断值差的化合物抛弃掉;在这过程中有的阳性化合物被虚拟筛选判为阴性化合物,此时产生假阴性;阳性化合物被正确识别为阳性,称为真阳性;对阴性化合物而言,同理也有假阳性与真阴性。绘制不同截断值下的假阳性率与真阳性率图,即得到ROC曲线(绿色曲线)。
图2. ROC曲线绘制的原理
好的虚拟筛选方法,其ROC曲线应该尽可能靠近左上角,完美方法的曲线下面积ROC AUC=1;靠运气、随机命中方法的ROC曲线靠近对角线(虚线),此时ROC AUC=0.5。如果一个虚拟筛选方法接近对角线,说明该方法与靠运气的随机筛选无显著区别;如果一个虚拟筛选方法的ROC曲线位于对角线的下方,说明这个方法虽然有富集能力,但是富集的是非活性化合物。
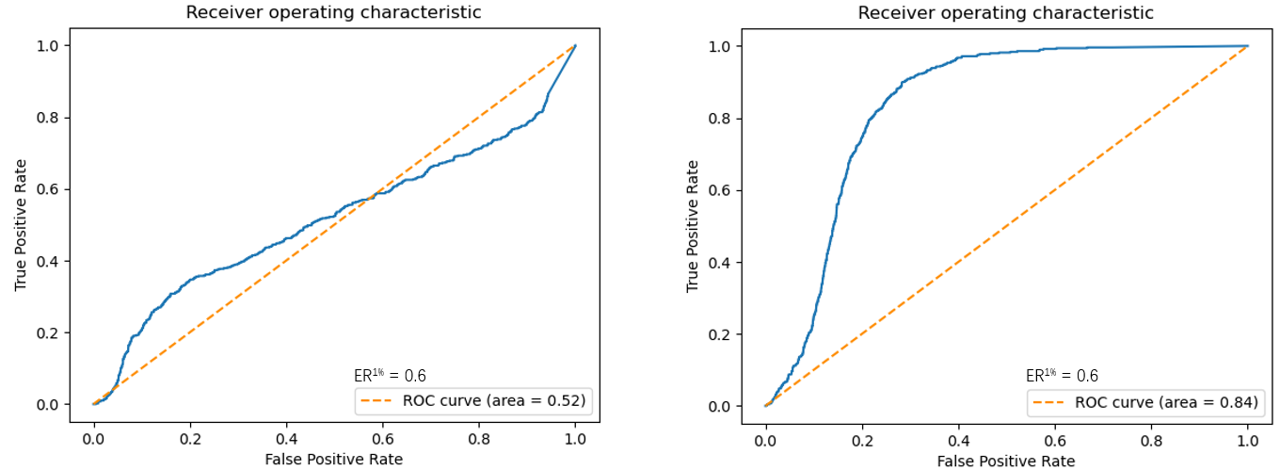
图3. 左:AUC低、早期富集能力差;右:AUC高、早期富集能力差
有意思的是,文献不太喜欢用信息丰富的ROC曲线,而更喜欢定量的ROC曲线下面积(ROC AUC)。AUC仅仅体现了虚拟筛选方法的总体性能。虽然AUC越大的方法,其总体性能越好,但是AUC的大小并不能反映一个虚拟筛选是否真正有用。如图3所示,两种方法的AUC面积差异巨大,左边的一个AUC=0.52,右边的那个AUC=0.84,但是两者在打分靠前的1%化合物,ROC曲线(蓝色)都位于对角线的下方,也就是打分靠前的1%化合物里找不到活性化合物。换句话说,如果对一个100万的化合物库进行虚拟筛选,需要在排名10000之后的化合物里面才能找到活性化合物,这说明AUC=0.52与AUC=0.84的两种方法是同样的不好。
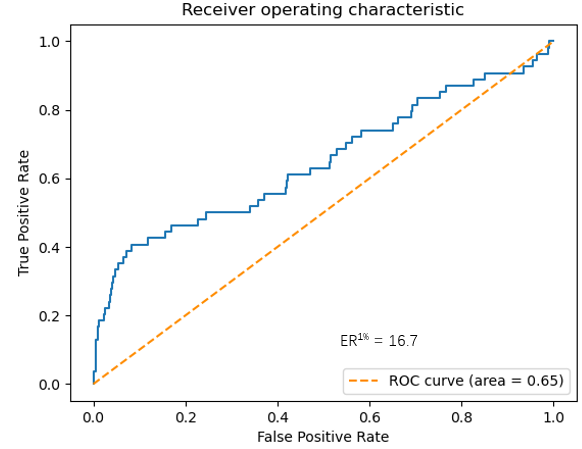
图4. AUC低、早期富集能力好的算例
AUC小于0.7通常被认为性能不好的虚拟筛选方法,这是非常有偏见而且经常会得出错误判断。如果图4所示的方法,其AUC=0.65,但是在打分靠前的部分,ROC曲线靠近纵坐标,这是富集能力好的表现:以假阳性率1%代价,获得真阳性率百分比16.7。
总的来说,使用单一的AUC值来比较性能差异存在诸多缺陷,AUC不能代替曲线图来评估虚拟筛选的性能,下面的评价早期富集能力的指标是非常必要的。更多的关于ROC曲线与ROC AUC的计算与讨论,请参阅Truchon等人的文献[1]。
2.2 命中率(Hit rate)
$$Hit\ rate = 100\% \times {Hits^{sampled} \over N^{sampled} }$$
其中N sampled是打分靠前的N个化合物;Hitssampled是打分靠前的N个化合物中真正为活性化合物的数量。
2.3 富集因子(Enrichment Factor, EF)与EFX%
$$EF = {{Hits^{sampled} \over N^{sampled}} \div {Hits^{total} \over N^{total}}}$$
其中Hitstotal为数据库中真正为活性化合物的总数;Ntotal为整个数据库中化合物的总数。
一般认为当EF值大于1时,这个方法具有显著地富集活性化合物的能力。
EF数学形式可以变换为:
$$EF = {{Hits^{sampled} \over N^{sampled}} \times {N^{total} \over Hits^{total}}}$$
其中:
$$EF^{Max} = {N^{total} \over Hits^{total}}$$
因此有:
$$EF = Hit\ rate \times EF^{Max}$$
对于给定的数据集,actives与decoys化合物数量一定,Ntotal/Hitstotal是常数,称为EFMax。不同的测试集其EFMax值不同,其EF值也不同,因此计算的EF值与化合物数量相关,可比性很差。
由于受到测试能力的限制,对于大化合物库的虚拟筛选只能对其中少部分化合物进行生物活性测试,尤其是打分最佳的那些化合物,这使得我们只关心虚拟筛选的早期富集能力。打分最高前的0.5%, 1%, 2%的EF就显得特别重要,此时的EF通常写为EFX%的形式:EF0.5%,EF1%,EF2%。
为了解决上述提到的EF值与active与decoy化合物数量比依赖问题,人们还提出了ROCe(ROC enrichment)这个指标,是指当收集到特定百分比的decoy化合物时,活性化合物的比例除以假阳性化合物的比例。ROCe代表了测试区分两类化合物的能力:活性化合物和非活性化合物。此指标需要说明所用的百分比信息,Jain和Nicholls25建议用0.5%、1.0、2%和5%等百分比值来报告“早期富集”值。
更多的富集因子讨论,请参阅Truchon等人的文献[1]。
2.4 BEDROC
Truchon等人[1]给出的BEDROC定义如下:
$$BEDROC = {{\sum_{i=1}^{n}{e^{-\alpha r_i/N}} \over {R_a({1-e^{-\alpha})} \over {e^{\alpha/N}-1}}} \times {R_asinh(\alpha/2) \over {cosh(\alpha/2)-cosh(\alpha/2-{\alpha R_a})}}+{1 \over {1-e^{\alpha(1-R_a)}}}}$$
其中n为actives化合物数;N为总的化合物数;Rα=n/N,即活性化合物数占总化合物数的比例;ri是根据打分排序,第i个活性物的排序位置。
BEDROC的计算相当复杂,理解起来不像EF那么直观。对于一个特定的靶标虚拟筛选结果,不像EF那样限于考察靠前特定百分数的化合物,BEDROC考虑了全部的化合物。然而,BEDEOC可以通过调整参数α来达到类似于考虑靠前特定百分数的效果。根据Chaput等人[2]对DUDE102个靶标研究,当α=321.9、80.5与20.0时BEDROC的评估效果分别与打分靠前0.5%、2%以及8%富集因子(EF)的评估效果相当。
2.5 ER1%
ER1%由Jain课题组提出用于评估早期富集能力[3],被定义为假阳性率为1%时的真阳性率百分数。
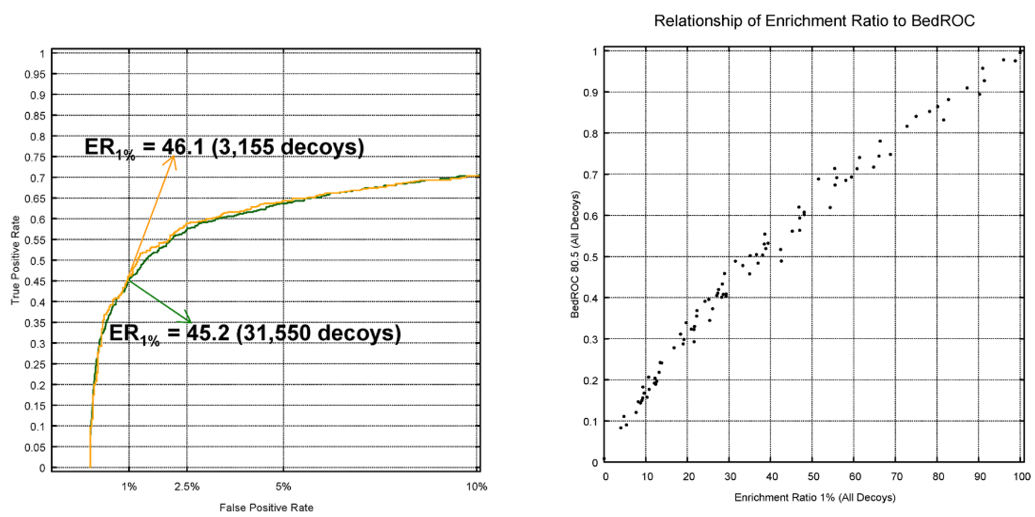
图5. 左:ER1%示意图;右:在DUDE 102个靶标上ER1%与BEDROCα=80.5的比较
图5(左)呈现了DUDE AA2AR靶标用两种虚拟筛选方法的ROC曲线,当FP rate=1%时对应TP Rate的百分数即为ER1%。根据Jain课题组的报道[3],在DUDE数据集的102个靶标上,ER1%与BEDROCα=80.5具有很好的相关性,虽然不是严格的线性,但是单调上升,如图5(右)所示。
2.6 半对数ROC曲线与logAUCλ=0.001
如2.1所述,ROC曲线通将真阳性率量化为假阳性率的函数来评估虚拟筛选方法从decoys中富集活性化合物的性能。ROC曲线下面积 (AUC) 是一个公认的性能度量标准,它通过一个数字来监控虚拟筛选的性能,图6a。对ROC曲线图横坐标的假阳性率进行对数转换具有放大真阳性率表示的早期富集能力的效果[4],这时得到半对数ROC曲线,如图6b所示。
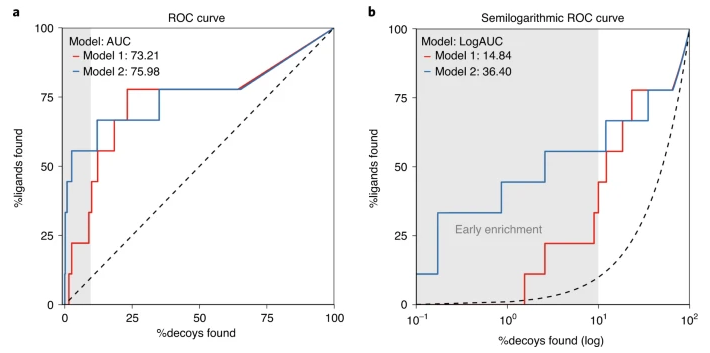
图6. 左:ROC曲线示意图;右:半对数ROC曲线示意图。本图片来自Bender等人的文章[5]。
提高早期富集性能的评估能力这一点很重要,因为在对数亿个分子的对接虚拟筛选中,通常只有排名靠前的0.1%化合物才会受到重视而下一步的实验评估。如果在回溯性对接计算中显示已知活性化合物仅在10%左右开始被识别出来,则预期在虚拟筛选中可能会遗漏新的活性化合物。半对数ROC曲线下面积用logAUC表示(如图6b所示),较高的LogAUC值意味着能更好地区分活性和非活性化合物,为对接参数以及方法识别活性化合物的能力提供了很好的指标。如何在虚拟筛选中使用logAUC评估对接方法的参数,推荐阅读Bender等人[5]与Stein等人[21]最近的文章,后者还提供了python代码[22]。
3. ROC曲线的绘制
3.1 用rocplotter绘制ROC曲线
药物设计平台ligandscout[6]提供了irocplotter来绘制ROC曲线,仅需要提供输入文件阳性与阴性化合物数量,以及命中化合物的sdf文件即可。
需要注意的是,在DUDE数据集中,阳性与阴性化合物被进行了互变异构体与质子化状态枚举,因此命中的化合物具有重复的可能,因此在绘制ROC曲线与计算AUC之前,要对命中的化合物进行去重等。以下的计算都是去重后进行的。输入阳性化合物的数量、阴性化合物的数量以及命中化合物sdf文件,即可绘制ROC曲线、计算EF与AUC。命令如下:
1 2 3 4 5 | (rdkit2020) [gkxiao@master pde5a]$ /public/gkxiao/software/ligandscout4/irocplotter \ -a 398\ -d 27521\ -i hits_nodup.sdf \ -o pde5a_roc.png |
其中-a参数为actives数据集的化合物数;其中-d参数为decoy数据集的化合物数;-i参数为虚拟筛选命中的化合物,由actives与decoys命中化合物合并、去重而来;-o参数为输出文件名称,默认生成png格式图片。键入命令后出现如下提示:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 | _ _ _
_ __ ___ ___ _ __ | | ___ | |_ | |_ ___ _ __
| '__| / _ \ / __| _____ | '_ \ | | / _ \ | __|| __| / _ \| '__|
| | | (_) || (__ |_____|| |_) || || (_) || |_ | |_ | __/| |
|_| \___/ \___| | .__/ |_| \___/ \__| \__| \___||_|
|_|
roc-plotter - LigandScout V4.4.8
Licensed to: Gaokeng Xiao, Guangzhou Molcalx Ltd (until 2023-01-01).
Actives: 398
Decoys: 27521
Hitlist: hits_nodup.sdf
Output: multi_vs_roc.png
Image-size: 600x600
Reading data (2/4) [=========================================] 100.000%
Reading data (4/4) [=========================================] 100.000%
Creating ROC-curve [=========================================] 100.000%
roc-plotter finished successfully. |
irocplotter默认保存的图片分辨率为600×600,结果如图7所示。

图7. irocplotter绘制的PDE5A系综虚拟筛选ROC曲线
3.2 用vROCS绘制ROC曲线
vROCS是OpenEye基于分子形状虚拟筛选ROCS的图形用户界面,使用户能够直接上手使用ROCS,vROCS包括一组统计工具来评估不同query的虚拟筛选性能。

图8. vROCS的的perform a ROCS validation
在vROCS点击perform a ROCS validation,见图8,导入actives与decoys化合物,完成虚拟筛选即可并绘制ROC曲线、计算EF值用于分析不同参数、方法对actives与decoys的区分能力,并给出不同方法之间具有统计学意义的显著差异,如图9所示。
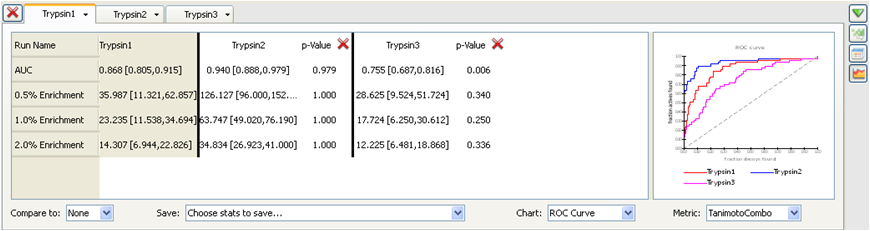
图9. 验证完毕vROCS报告EF值与ROC曲线
3.3 用R软件包绘制ROC曲线
R软件包提供了多种ROC曲线绘制工具,比如enrichvs、ROCR、pROC等。以enrichvs为例,说明如何绘制ROC曲线。
- 数据整理
- 数据导入
- 绘制ROC曲线
- 计算pROC AUC
- 计算ROC AUC
从DUDE上下载PDE5A数据集[8,9],用FRED将active与decoy的分别进行对接计算,具体方法见2022-03-02的博客文章[10]。将打分结果合并生成一个文本文件,至少包含两列: 一列为打分值(越低越好),一列为标签(1表示为活性化合物ligand的打分,0表示对应这decoy的打分):
1 2 3 4 5 6 7 8 | Title score label ZINC04199924 -16.976294 1 ZINC04199924 -16.976294 1 ZINC04199929 -16.28743 1 ZINC04199923 -16.262144 1 ZINC03792330 -14.876609 1 ZINC03786304 -14.802482 0 ... |
假设上面的内容保存在pde5_score.txt的文件里。
在R里键入如下命令:
1 | pde5=read.table("d:/fred/dud/pde5_score.txt",header=T) |
在R里键入如下命令:
1 | plot_enrichment_curve(pde5$score, pde5$label,decreasing=FALSE, col="blue") |
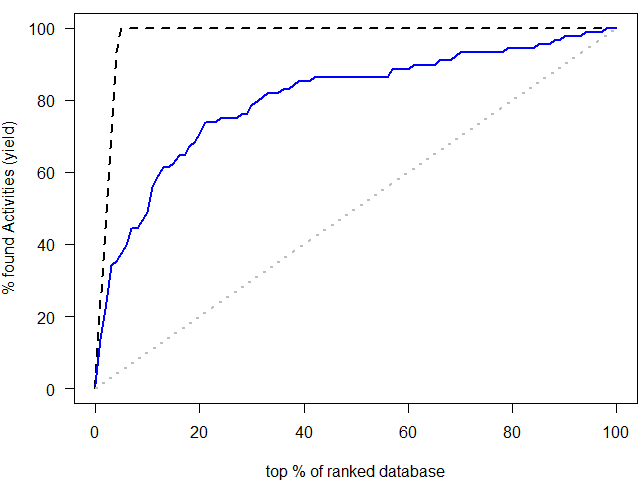
结果如图10所示。
图10. FRED对接计算的打分值绘制ROC曲线图(DUD PDE5A数据集)
计算top 1%的ROC AUC:
1 2 | auac(score$score,score$label,decreasing=FALSE,top=0.01) [1] 0.08435961 |
1 2 | auc(score$score,score$label,decreasing=FALSE) [1] 0.7996552 |
3.4 用python的sklearn软件包来绘制ROC曲线
Python的sklearn库提供了强大的模块可以用来对虚拟筛选方法进行性能评估[11],包括ROC曲线的绘制,AUC的计算等等。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 | #!/usr/bin/env python # coding: utf-8 import pandas as pd import numpy as np from sklearn import metrics import os # 读入docking后的打分文件 df = pd.read_csv('pde5_score.csv') scores = df.score y = df.label # 计算假阳性,假阴性与阈值 fpr, tpr, thresholds = metrics.roc_curve(y, -scores, pos_label=1) # 计算ROC AUC auc = metrics.auc(fpr, tpr) print('AUC ROC:',auc) #绘图 import matplotlib.pyplot as plt plt.plot(fpr,tpr) plt.plot([0, 1], [0, 1], color="darkorange", linestyle="--",label="ROC curve (area = %0.2f)" % roc_auc) plt.ylabel('True Positive Rate') plt.xlabel('False Positive Rate') plt.title("Receiver operating characteristic") plt.legend(loc="lower right") plt.show() |
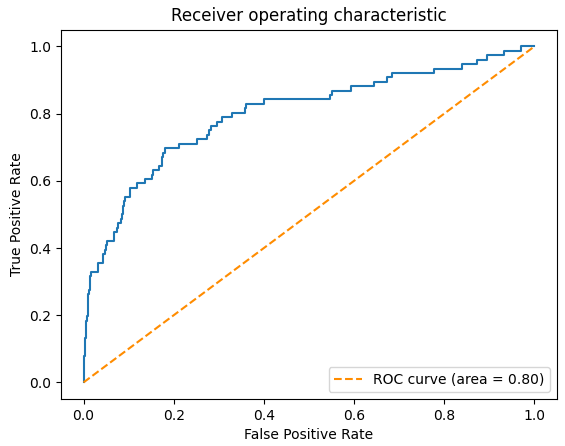
图11 用Python绘制的ROC曲线
3.5 用ODDT软件包来计算AUC,logAUC,BEDROC与Enrichment Factor
Open Drug Discovery Toolkit(ODDT)是一个综合性的化学信息学与分子模拟工具包[12],它可以一站式计算AUC,logAUC,BEDROC与EF值等。下面的Jupyter notebook演示了这些指标的详细计算过程。
可以发现,本节计算得到的AUC与3.3、3.4小节计算得到的AUC是一致的。在文献里经常还用到减扣掉随机曲线下面积的logAUC(即adjusted-logAUC):
adjusted-logAUC = logAUC – 0.14462
在文献里adjusted-logAUC经常也被称为logAUC,需要注意到底是指哪一种logAUC。
3.6 用Rocker可视化分析AUC与logAUC
Rocker是Lätti等人开发的虚拟筛选性能评估与可视化工具[19],我们就用Rocker绘制过DUDE+ PDE5A数据集FRED与HYBRID的半对数ROC曲线[20],如图12所示。
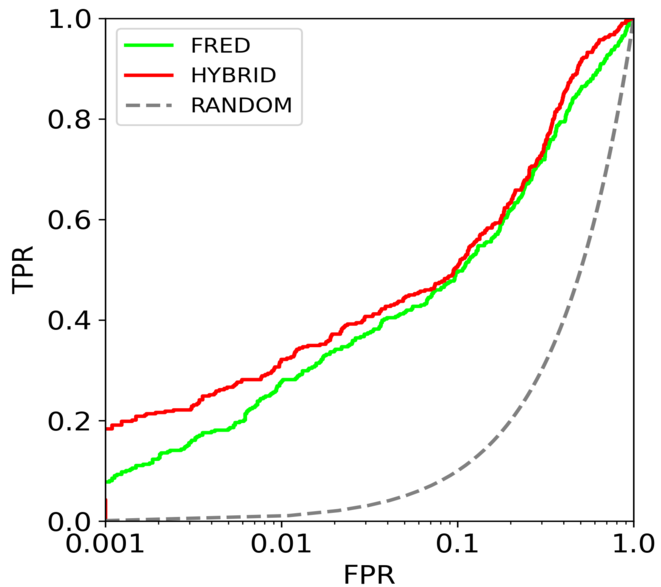
图 12. FRED与HYBRID的半对数ROC曲线,λ=0.001,图片来自文献[20]
由于Rocker极其友好,且文章对使用场景与使用方法描述的非常清楚,本文就不再叙述,如果有需要的话请阅读Lätti等人的原文[19]。
3.7 用bootstrap可视化分析AUC与logAUC
UCSF的stein等人[21,22]提供的bootstrap工具可以非常方便地对AUC与logAUC进行可视化,并附有非常详细的教程,建议将bootstrap作为logAUC可视化的首选。
简单来说,使用前需要预先准备三个文件:(1)排序过的是打分结果文件(比如D4_std.txt),包含两列:第1列是化合物名称,第2列是打分值;(2)actives名称文件(ligands.name):包含了活性化合物的名称;(3)decoys化合物名称文件(decoys.name):包含了decoys化合物的名称。
其中,D4_std.txt为对接打分结果文件,排序从优到差, 前10行如下:
1 2 3 4 5 6 7 8 9 10 | C72350273 -61.01 C21631568 -58.74 C11663356 -57.78 C39779742 -57.13 C70981220 -57.03 Nemonapride -56.66 C53017158 -56.66 C08915125 -56.49 C85039153 -56.44 C50617914 -56.27 |
D4_std.txt对接打分结果文件的后10行如下:
1 2 3 4 5 6 7 8 9 10 | C64058375 14.90 C70129778 15.13 C37857560 17.21 C87843872 17.88 C21151058 20.40 C00413647 22.57 C96013516 22.63 C55343107 22.63 C58364575 24.62 C70069523 27.35 |
其中,ligands.name文件给出了打分文件中属于actives化合物的名称,前10行如下:
1 2 3 4 5 6 7 8 9 10 | Nemonapride
L750667
Spiperone
NGD941
Benperidol
L745870
Sonepiprazole
U101958
A-381393
Haloperidol |
其中,decoys.name文件给出了打分文件中属于decoys化合物的名称,前10行如下:
1 2 3 4 5 6 7 8 9 10 | C97366660 C97408377 C97408722 C97409464 C98261867 C06668623 C53073497 C80988397 C87843461 C90817333 |
绘制半对数曲线则很简单:
1 | bootstrap_tldr.py -lig ligands.name -dec decoys.name -s1 D4_std.txt -m logAUC |
计算完毕,会生成一个名为plot_logAUC.png的文件,如图13所示。
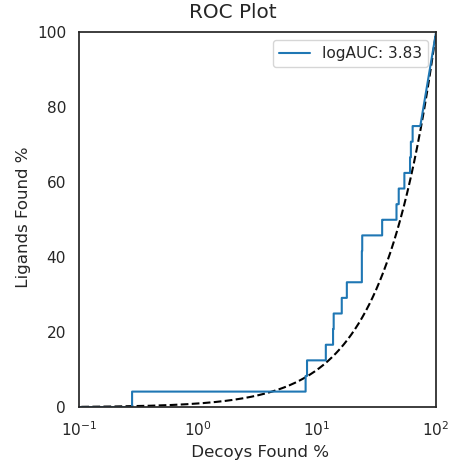
图 13. 用UCSF boostrap绘制D4算例的半对数ROC曲线
需要注意的是,bootstrap计算的logAUC已经减扣了随机曲线下面积(0.14462),这时logAUC也称为adjusted-logAUC,通常都不加区分的称为logAUC。
4. 为什么要设计decoy数据集来进行方法学验证
以分子对接为例,分子对接常用于:虚拟筛选发现苗头化合物、预测结合模式、根据打分值进行先导化合物物优化。为了考察分子对接打分能否可靠的用于先导物优化,Enyedy等人[13]考察了打分值能否区分活性化合物与非活性化合物。结果发现,就ROC AUC而言,分子量与ClogP跟GLIDE的GLIDE_XP与GLIDE_SP一样具有同样的活性与非活性化合物区分能力,见图14A、B。
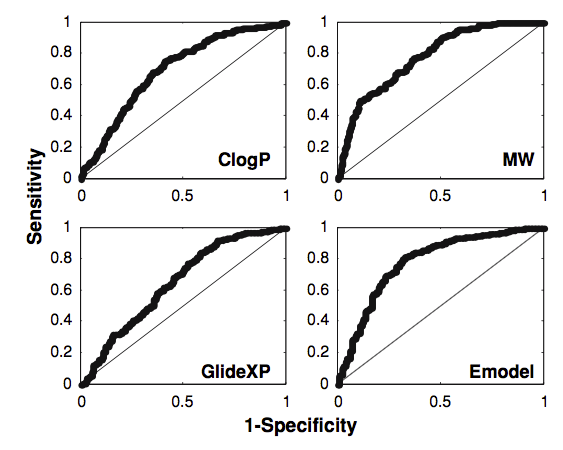
图14 A. KDR(1YWN)对接计算的ROC曲线

图14 B. KDR(1YWN)对接计算的ROC曲线
由此可见,我们不能仅根据分子对接与打分值来指导先导的结构优化,专家的解释还是非常重要的;对接软件的打分值与可能化合物的物理性质诸如分子量、极性表面积、油水分配系数、柔性键数量、氢键受体数、氢键供体数有很强的相关性。如果活性化合物与数据库化合物在这些物理性质上很大的差异,那么虚拟筛选性能的评估结果就会很受质疑:很可能是物理性质自身差异的原因导致化合物被富集出来,而不是分子对接或打分函数本身。
同时,也说明合理地选用验证用的数据集很重要:首先这个数据集应该消除化合物物理性质带来的偏差。Huang等人[14]开发的DUD基准数据集正是专为虚拟筛选方法验证而设计。DUD数据集在使用过程中也发现一些缺陷,并被不断改进。DUDE[8]不仅是DUD的曾强版,还可以帮助我们生成活性化合物的decoy数据集。还有其它的数据集可供使用,比如Bauer等人开发的DEKOIS 2.0数据集[15],以及最近Cleves等人的DUDE+数据集[3]。其中DUDE+是为评估ensemble虚拟筛选策略的而设计。
5.一些流行软件的虚拟筛选性能评估文献
文献报道的一些流行的虚拟筛选软件在公开数据集上的性能评估汇总见表1,为了方便使用,已经将结果制成csv表格,具体见virtual-screening-validation[18]。
表1、常见流行软件的虚拟筛选性能评估文献
| 文献 | 数据集 | 涉及的软件 | 性能指标 |
|---|---|---|---|
| [2] | DUDE | GLIDE,GOLD,Surflex,FLEXX | BEDROCα=80.5 |
| [3] | DUDE+ | Surflex | AUC,ER1% |
| [8] | DUDE | DOCK | AUC,logAUC,EF1% |
| [16] | DUDE | GLIDE | AUC,BEDROCα=80.5 |
| [17] | DUDE | Vina 1.2 | AUC,BEDROCα=20.0,EF1%,EF5%,EF10% |
| [23] | DUDE | AtomNet | AUC,logAUC |
| [24] | DUDE,DEKOIS2.0,dataset III | GLIDE,GOLD,LeDock | AUC,logAUC,BEDROCα=80.5,EF0.1%,EF0.5%,EF1%,EF5% |
6.附件
PDE5数据集FRED打分的结果:pde5_score.csv
7.文献
- Truchon, J. F.; Bayly, C. I. Evaluating Virtual Screening Methods: Good and Bad Metrics for the “Early Recognition” Problem. J. Chem. Inf. Model. 2007, 47 (2), 488–508. https://doi.org/10.1021/ci600426e.
- Chaput, L.; Martinez-Sanz, J.; Saettel, N.; Mouawad, L. Benchmark of Four Popular Virtual Screening Programs: Construction of the Active/Decoy Dataset Remains a Major Determinant of Measured Performance. J. Cheminform. 2016, 8 (1), 56. https://doi.org/10.1186/s13321-016-0167-x.
- Cleves, A. E.; Jain, A. N. Structure- and Ligand-Based Virtual Screening on DUD-E + : Performance Dependence on Approximations to the Binding Pocket. J. Chem. Inf. Model. 2020, 0 (0), acs.jcim.0c00115. https://doi.org/10.1021/acs.jcim.0c00115.
- Mysinger, M. M.; Shoichet, B. K. Rapid Context-Dependent Ligand Desolvation in Molecular Docking. J. Chem. Inf. Model. 2010, 50 (9), 1561–1573. https://doi.org/10.1021/ci100214a.
- Bender, B. J.; Gahbauer, S.; Luttens, A.; Lyu, J.; Webb, C. M.; Stein, R. M.; Fink, E. A.; Balius, T. E.; Carlsson, J.; Irwin, J. J.; et al. A Practical Guide to Large-Scale Docking. Nat. Protoc. https://doi.org/10.1038/s41596-021-00597-z.
- Ligandscout. http://www.inteligand.com/ligandscout
- ROCS. https://www.eyesopen.com/rocs.
- Mysinger, M. M.; Carchia, M.; Irwin, J. J.; Shoichet, B. K. Directory of Useful Decoys, Enhanced (DUD-E): Better Ligands and Decoys for Better Benchmarking. J. Med. Chem. 2012, 55 (14), 6582–6594. https://doi.org/10.1021/jm300687e.
- DUDE的PDE5a数据集. 2022-01-25访问. http://dude.docking.org/targets/pde5a
- 肖高铿.用多个蛋白信息来提高用单一蛋白结构分子对接虚拟筛选的性能. 墨灵格的博客. 2022-02-03. http://blog.molcalx.com.cn/2022/03/02/oedocking-pde5a.html
- sklearn.metrics.roc_auc_score. 2022-03-04访问. https://scikit-learn.org/stable/modules/generated/sklearn.metrics.roc_auc_score.html
- ODDT. Open Drug Discovery Toolkit. https://github.com/oddt/oddt
- Enyedy, I. J.; Egan, W. J. Can We Use Docking and Scoring for Hit-to-Lead Optimization? Journal of computer-aided molecular design 2008, 22 (3-4):161–168.
- Huang, N.; Shoichet, B. K.; Irwin, J. J. Benchmarking Sets for Molecular Docking. J. Med. Chem. 2006, 49 (23), 6789–6801. https://doi.org/10.1021/jm0608356.
- Bauer, M. R.; Ibrahim, T. M.; Vogel, S. M.; Boeckler, F. M. Evaluation and Optimization of Virtual Screening Workflows with DEKOIS 2.0 – A Public Library of Challenging Docking Benchmark Sets. J. Chem. Inf. Model. 2013, 53 (6), 1447–1462. https://doi.org/10.1021/ci400115b.
- Wang, D.; Cui, C.; Ding, X.; Xiong, Z.; Zheng, M.; Luo, X.; Jiang, H.; Chen, K. Improving the Virtual Screening Ability of Target-Specific Scoring Functions Using Deep Learning Methods. 2019, 10 (August), 1–11. https://doi.org/10.3389/fphar.2019.00924.
- Eberhardt, J.; Santos-Martins, D.; Tillack, A. F.; Forli, S. AutoDock Vina 1.2.0: New Docking Methods, Expanded Force Field, and Python Bindings. J. Chem. Inf. Model. 2021, acs.jcim.1c00203. https://doi.org/10.1021/acs.jcim.1c00203.
- virtual screening validation. https://github.com/gkxiao/virtual-screening-validation
- Lätti, S.; Niinivehmas, S.; Pentikäinen, O. T. Rocker: Open Source, Easy-to-Use Tool for AUC and Enrichment Calculations and ROC Visualization. J. Cheminform. 2016, 8 (1), 45. https://doi.org/10.1186/s13321-016-0158-y.
- 肖高铿. 用多个蛋白信息来提高用单一蛋白结构分子对接虚拟筛选的性能. 墨灵格的博客. 2022-03-01. http://blog.molcalx.com.cn/2022/03/02/oedocking-pde5a.html
- Stein, R. M.; Yang, Y.; Balius, T. E.; O’Meara, M. J.; Lyu, J.; Young, J.; Tang, K.; Shoichet, B. K.; Irwin, J. J. Property-Unmatched Decoys in Docking Benchmarks. J. Chem. Inf. Model. 2021, 61 (2), 699–714. https://doi.org/10.1021/acs.jcim.0c00598.
- bootstrap. https://dudez.docking.org
- Izhar, W.; Michael, D.; Abraham, H. AtomNet: A Deep Convolutional Neural Network for Bioactivity Prediction in Structure-Based Drug Discovery. arXiv:1510.02855 2015.
- Shen, C.; Hu, Y.; Wang, Z.; Zhang, X.; Pang, J.; Wang, G.; Zhong, H.; Xu, L.; Cao, D.; Hou, T. Beware of the Generic Machine Learning-Based Scoring Functions in Structure-Based Virtual Screening. Brief. Bioinform. 2021, 22 (3), 1–22. https://doi.org/10.1093/bib/bbaa070.
- Jain, A. N., & Nicholls, A. Recommendations for evaluation of computational methods. Journal of Computer-Aided Molecular Design, 2008, 22(3), 133–139. https://doi.org/10.1007/s10822-008-9196-5