摘要:本文以DUD-E的EGFR子集为例,在Flare Docking的虚拟筛选模式下对Actives与Decoys进行了分子对接计算,并进一步用GNINA的CNNscore与CNNaffinity进行再次打分,将Flare docking的VSscore与CNNscore进行相乘作为新的打分值,最后对不同的打分值进行了虚拟筛选性能评估,尤其是表示早期富集能力的BEDROC与EF值,并与文献报道的Glide SP进行了比较。结果表明,LF CNN VS的富集能力优于独立的LF VSscore与CNNscore打分函数;同样,CNN_VS的富集能力也优于各自独立的CNNaffinity与CNNscore。总的来说,CNNscore与Flare Docking的LF VSscore以及GNINA的CNNaffinity显示出协同增强虚拟筛选性能的能力。在DUD-E的EGFR这个子集上,LF CNN VS与CNN_VS在EBEDROC与EF等关键的虚拟筛选性能指标上与文献报道的Glide SP表现出相似的性能。
作者:肖高铿/2023-07-15
前言
分子对接方法预测蛋白-配体结合模式的问题之一是打分最佳的那个结合模式(Pose)不一定是对的。比如,Wang等人1以PBDbind数据库(2014版)的2002个蛋白-配体复合物结构作为测试用数据集,评估了10个分子对接软件的采样性能与打分性能。结果表明,在这2002个算例上,不同分子对接软件的打分最佳结合模式为正确的算例百分比为40-60%,在输出的多个结合模式中包含一个正确结合模式的算例百分比为60-80%。
打分函数的上述缺陷可能导致虚拟筛选的排序失败,表现为虚拟筛选苗头化合物包含过多的假阳性分子。这是因为在实际的虚拟筛选研究中通常将打分最佳的结合模式认为是最合理的那个,并用于优先级排序以识别苗头化合物。Lyu等人2对打分最优的化合物进行了仔细检查,发现高分的假阳性分子往往利用利用了打分函数近似处理的局限性与靶标本身的限制,从而欺骗打分函数得到了过高的分值。假设假阳性率仅为十万分之一至百万分之一之间,在对10亿级别的超大规模数据库虚拟筛选时,排名靠前的位置大部分将被假阳性分子占据。也就是说,若仅从排名靠前的分子中进行选择,除非后续有能力合成几千个化合物进行活性测试,否则所得到的化合物绝大多数都是假阳性。
对分子对接虚拟筛选结果进行后处理是降低假阳性率的必要步骤,已经有非常多的综述讨论,比如最近Fischer等人3的文章就对此做了全面的描述。对接后处理方法主要方法之一是对pose过滤,这要求用户对研究对象有深刻的了解并将之转化为药效团之类的假设才能完成。无需在先知识的后处理方法也是人们所乐见的。随着深度学习方法在分子对接领域应用的不断发展,出现了很多新的打分函数,包括专门评估结合模式的打分函数,比如实现在分子对接软件GNINA的CNNscore4-6是一个Pose打分函数,其取值范围在0-1之间,值越大,pose为正确的概率越大。将GNINA的Pose打分与亲和力打分(CNN affinity)相乘是一个非常朴素的一致性打分思路:让亲和力预测值高且Pose正确概率高的结果排在前面,亲和力预测值高而Pose正确概率低的结果排在后面。事实证明,这个数据融合策略可以显著提高虚拟筛选的性能6,这同时也提供了一种便利的、避免需要使用在先知识的分子对接后处理方法。本文以DUD-E的EGFR子集7,8为例,演示了如何将Flare docking9与GNINA CNNscore通过数据融合(Data fusion)来提高虚拟筛选的性能。
方法
分子对接虚拟筛选流程
Flare Docking以Lead Finder12为计算引擎,如下图1所示,典型的计算流程包括4个步骤:1)蛋白模型构建;2)能量格点计算;3)在能量格点里对接分子;4)打分。
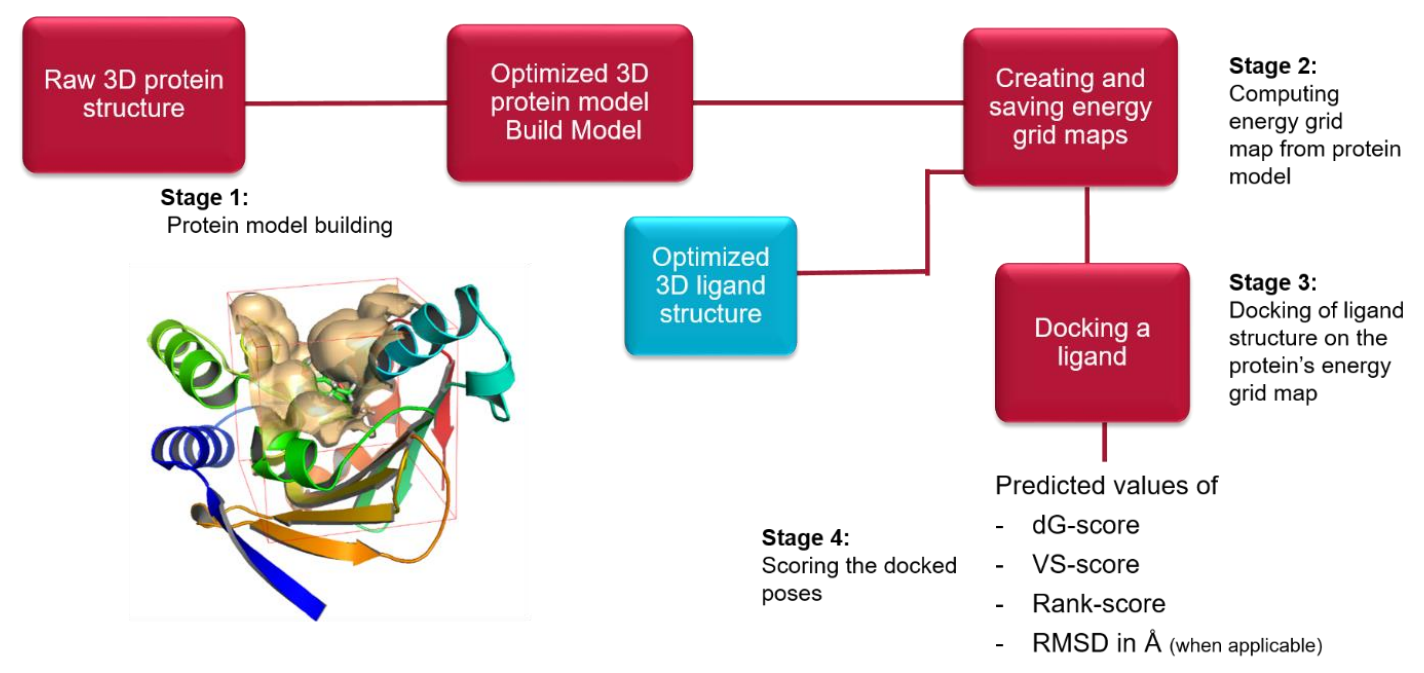
图1. Flare docking的工作流
Flare Docking有三种分子对接模式:标准、穷尽与虚拟筛选。在本文中为了兼顾速度,采用虚拟筛选模式。Lead Finder的打分函数是一种半经验的力场方法,并显式地考虑了不同类型的分子间相互作用。对每个打分成分用一个经验系数进行缩放,得到三种不同目的的打分函数:
- dG-score:预测结合亲和力
- VS-score:在虚拟筛选中对不同的分子排序
- Rank-Score:对结合模式进行排序
蛋白的准备
用Flare下载HYZ-EGFR kinase的共晶结构PDB 2RGP,并用Protein Prep工具进行结构准备,仔细检查结构以确保配体结构正确、相互作用合理,删除全部的水,仅保留A链的激酶部分与配体,分别导出为2rgp_prot.pdb与2rgp_ligand.sdf备用。
数据库分子的准备
DUD-E EGFR数据集8里预先准备好的SDF格式actives(actives_final.sdf)与decoys(decoys_final.sdf)结构直接用于虚拟筛选,不进行额外的准备。
分子对接虚拟筛选
分子对接虚拟筛选使用pyflare的docking脚本,在命令行下计算能量格点并执行虚拟筛选。
以共晶配体为参比分子,跑一个redocking,同时进行能量格点计算:
1 2 3 4 5 6 7 8 | pyflare docking.py -p 2rgp_prot.pdb -j pdb --protein-chain-types p \ --reference 2rgp_ligand.sdf -i sdf \ --write-grid 2rgp_prot.grid \ --csv redock.csv \ --max-poses 20 \ --batch-size 120 \ --quality virtual-screening \ 2rgp_ligand.sdf -i sdf >> redock.sdf |
计算完毕,生成了一个新的文件2rgp_prot.grid即为所需的能量格点文件。
actives虚拟筛选:
1 2 3 4 5 6 7 | pyflare docking.py -p 2rgp_prot.pdb -j pdb --protein-chain-types p \ --grid-from-file 2rgp_prot.grid \ --csv actives_dock.csv \ --max-poses 20 \ --batch-size 120 \ --quality virtual-screening \ actives_final.sdf -i sdf >> actives_dock.sdf |
计算完毕得到的actives_dock.sdf文件即为actives数据集对接结果文件。
decoys虚拟筛选:
1 2 3 4 5 6 7 | pyflare docking.py -p 2rgp_prot.pdb -j pdb --protein-chain-types p \ --grid-from-file 2rgp_prot.grid \ --csv actives_dock.csv \ --max-poses 20 \ --batch-size 120 \ --quality virtual-screening \ decoys_final.sdf -i sdf >> decoys_dock.sdf |
计算完毕得到的decoys_dock.sdf文件即为decoys数据集对接结果文件。
用GNINA对对接结果进行重新打分
用GNINA对actives对接结果进行重新打分:
1 | gnina --config dock.conf -l actives_dock.sdf -o actives.sdf --score_only |
用GNINA对decoys对接结果进行重新打分:
1 | gnina --config dock.conf -l decoys_dock.sdf -o decoys.sdf --score_only |
其中dock.conf为gnina对接参数文件,内容如下:
1 2 3 4 5 6 7 | receptor = 2rgp_prot.pdbqt center_x = 17.2055 center_y = 35.6595 center_z = 91.068 size_x = 30 size_y = 30 size_z = 30 |
GNINA打分之后,在sdf文件里包含了3个新的打分值:
- CNNscore: GNINA的深度学习pose打分值
- CNNaffinity:GNINA的深度学习结合亲和力预测值
- CNN_VS:CNNscore x CNNaffinity
数据融合
Flare docking对接计算产生LF VSscore,LF dG与LF Rank Score等多个打分值,其中LF VSscore用于对化合物进行打分排序。用GNINA重新打分之后,得到三个新的打分值CNNscore,CNNaffinity与CNN_VS。其中CNNscore是pose打分,CNNaffinity是亲和力打分,CNN_VS是CNNscore与CNNaffinity的乘积,即:
CNN_VS = CNNaffinity x CNNscore
在本文中,将CNNscore与LF VSscore进行相乘:
LF CNN VS = LF VSscore x CNNscore
我们关心的是LF CNN VS是否比独立LF VSscore或CNNscore具有更好的虚拟筛选富集能力;同时也验证CNN_VS是否比CNNaffinity或CNNscore具有更好的虚拟筛选富集能力。
数据处理与虚拟筛选性能评估
鉴于从DUD-E下载的actives与decoys数据集进行了互变异构体与质子化状态枚举处理,导致一个化合物会以不同的形式多次出现、并可能被虚拟筛选多次命中,因此对虚拟筛选结果按化合物名称进行去重,同名化合物仅以其中打分最高那个来代表,所有的性能评估指标都在去重的基础上进行计算。
在虚拟筛选过程中,有可能有的化合物处理失败而没有出现在结果文件里,为了保证性能评估的结果与其它文献报道的具有可比性,将缺失的化合物人为地分配一个打分值0然后再进行性能评估。
用AUC、logAUC、三种α(321.9,80.5与20.0)值的BEDROC、富集因子(top 0.5%,1%,5%与10%)等指标来评估虚拟筛选的性能,具体的计算方法参见前文10。
结果
数据处理
在本次计算中,对接之后的结果用python的pandas的聚合函数groupby对化合物分值进行处理:同一个化合物的互变异构体、不同质子化状态仅保留打分最优的那个作为该化合物的打分值。actives与decoys数据集的处理结果如表1所示:actives都成功处理,decoys有42个分子处理失败。因此,在之后的性能评估中,这些处理失败的分子给予一个0的打分值之后参与统计,以便得到的性能指标与文献报道的指标具有可比性。
表1、DUD-E EGFR数据集化合物统计
| EGFR数据集 | 输入的化合物数 | 输入的异构体数 | 输出的化合物数 |
|---|---|---|---|
| actives | 542 | 832 | 542 |
| decoys | 35020 | 35442 | 34978 |
数据融合后的打分结果见附件all_score.csv,去重后的可直接用于统计处理的打分结果文件见附件all_score_grouped.csv,主要的虚拟筛选性能统计学指标如表2所示。
表2、用PDB 2RGP的共晶结果进行分子对接虚拟筛选的性能指标
| 指标 | LF VSscore | LF CNN VS | CNNaffinity | CNNscore | CNN VS | Glide11 |
|---|---|---|---|---|---|---|
| AUC | 0.713 | 0.853 | 0.772 | 0.833 | 0.847 | 0.821 |
| logAUCλ=0.1% | 35.1 | 50.6 | 44.7 | 47.5 | 51.7 | 46.1 |
| adj-logAUCλ=0.1% | 20.6 | 36.2 | 30.2 | 33.0 | 37.2 | 31.7 |
| BEDROCα=321.9 | 0.262 | 0.607 | 0.598 | 0.498 | 0.637 | 0.641 |
| BEDROCα=80.5 | 0.248 | 0.433 | 0.406 | 0.390 | 0.462 | 0.392 |
| BEDROCα=20.0 | 0.338 | 0.501 | 0.436 | 0.474 | 0.522 | 0.427 |
| EF0.5% | 16.2 | 40.5 | 37.2 | 32.4 | 40.9 | 41.3 |
| EF1% | 14.6 | 26.9 | 26.0 | 24.5 | 28.0 | 25.6 |
| EF5% | 7.1 | 10.1 | 8.5 | 9.7 | 10.5 | 8.2 |
| EF10% | 4.4 | 6.3 | 5.1 | 5.9 | 6.4 | 5.3 |
Glide的性能指标是根据Shen等人11报道的打分值重新统计得到。
数据融合产生了协同效应
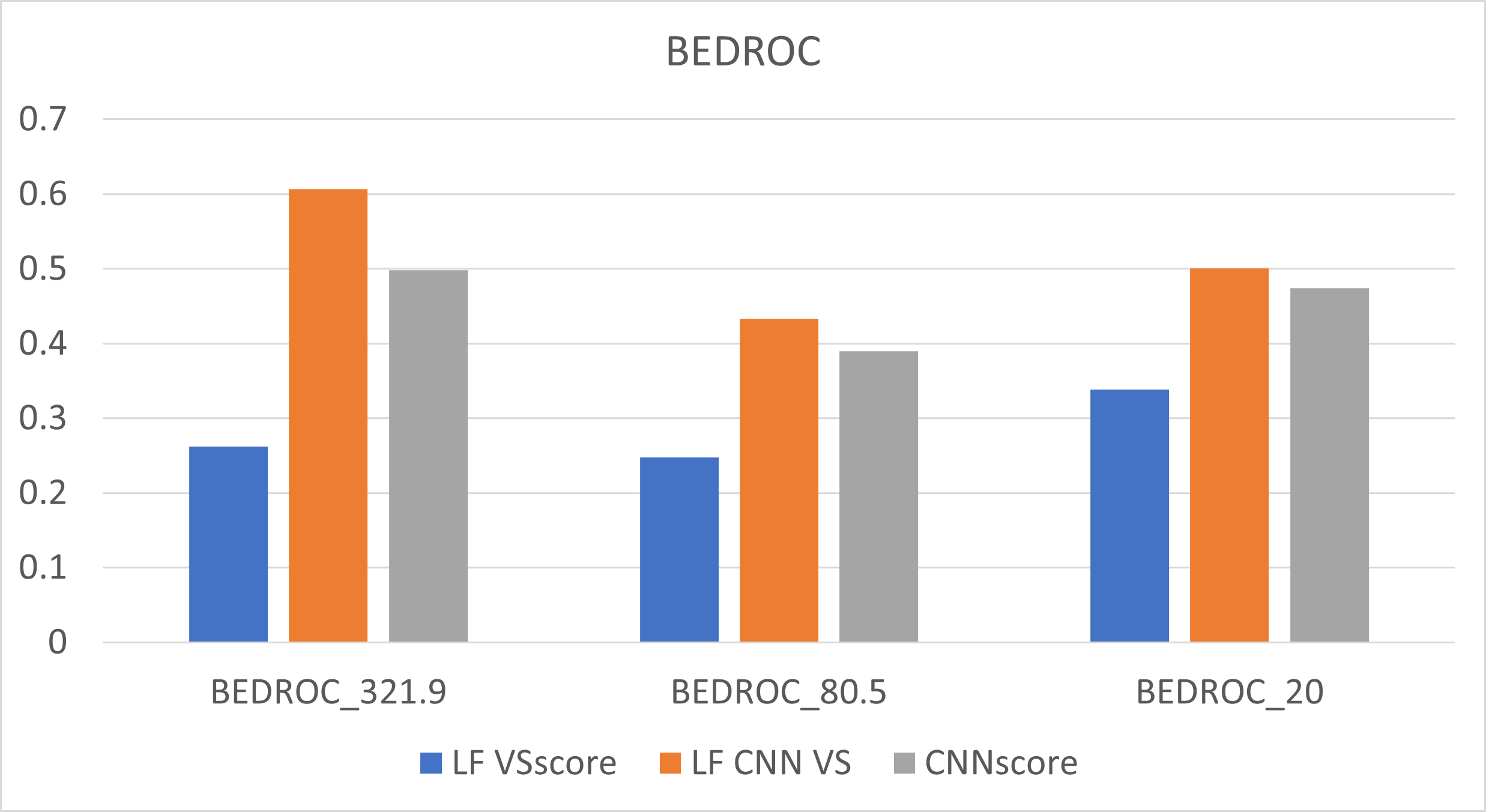
图1. LF VSscore、CNNscore、LF CNN VS的BEDROC比较
首先,以BEDROC表示的早期富集能力为例,无论α=321.9、80.5、20.0,LF VSscore与CNNscore相乘之后的LF CNN VS得到的BEDROC比相乘之前任意一个单独打分函数的BEDROC都高,如图1所示。
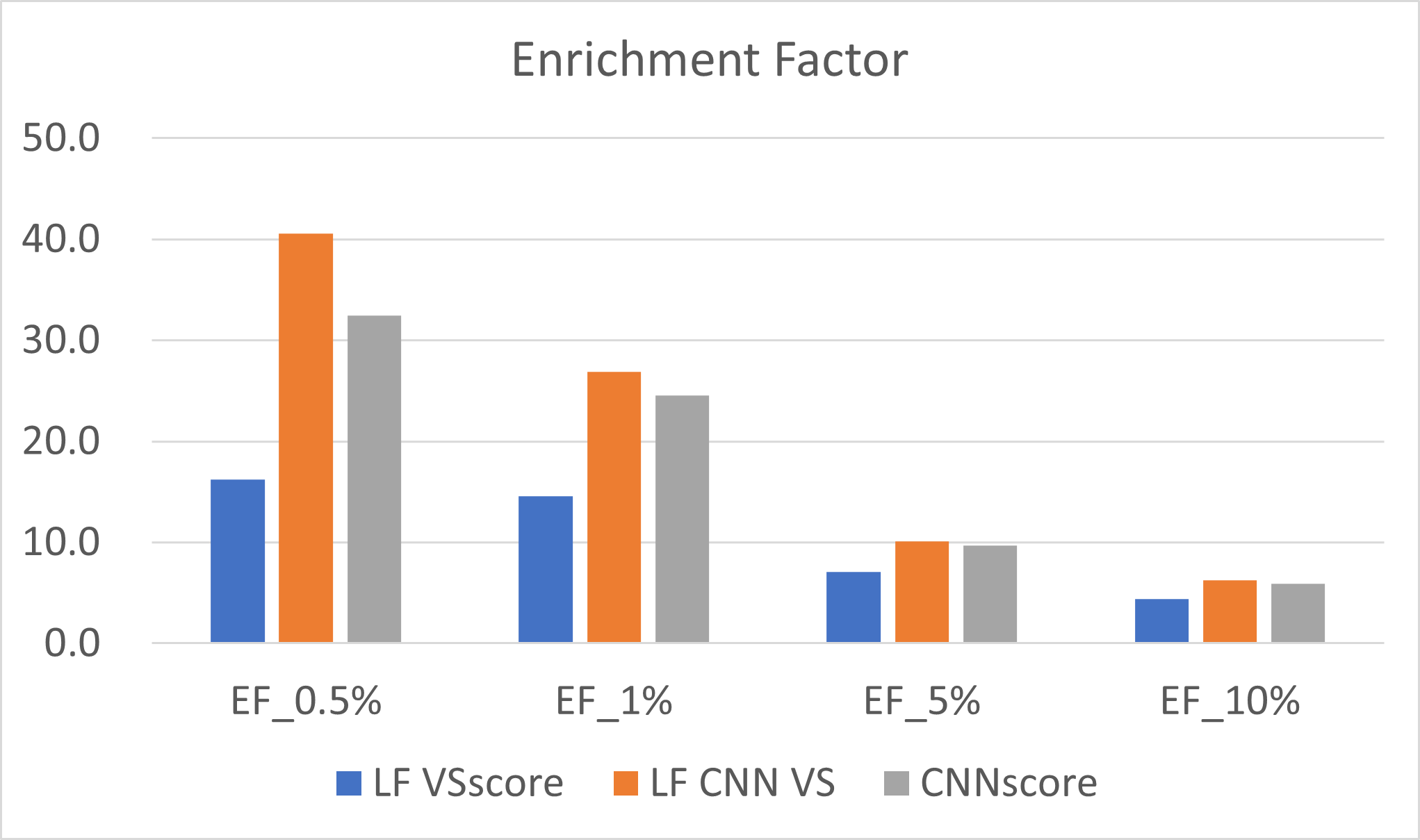
图2. LF VSscore、CNNscore、LF CNN VS的EFX%(X=0.5,1,5,10)比较
其次,数据融合显著提高了top 0.5%与1%的富集因子EF,如图2所示,LF VSscore与CNNscore相乘之后的LF CNN VS得到的EF值比相乘之前的任意一个单独打分函数的都高。
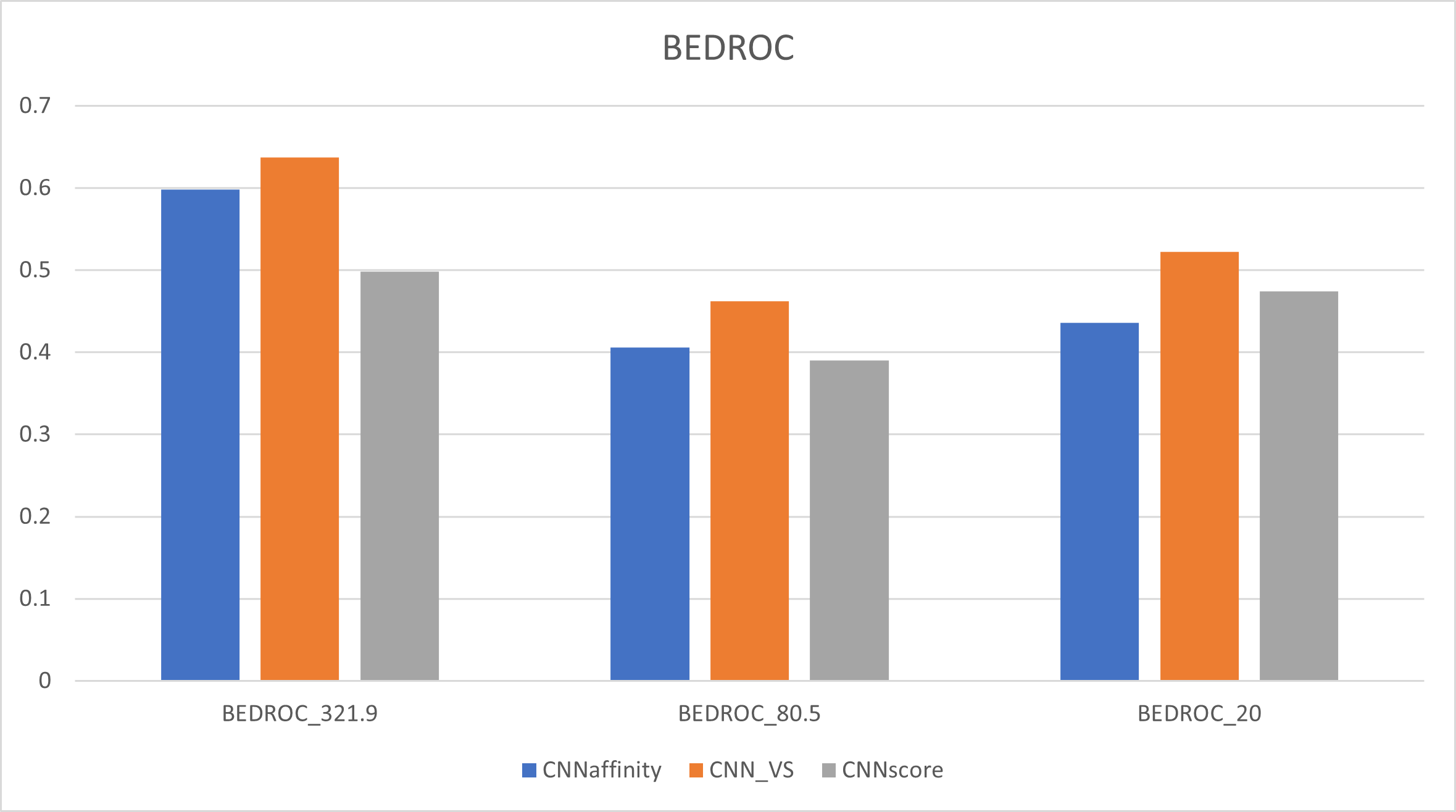
图3. CNNaffinity,CNNscore与CNN_VS重新打分的BEDROC比较
在GNINA的测试中6,CNNaffinity同时在虚拟筛选富集能力与pose预测能力上都表现出一致的良好性能并用做默认的虚拟筛选打分函数,但是CNNscore与CNNaffinity相乘得到的CNN_VS依旧表现有相似的协同效应。在本算例中,无论是BEDROD还是EF值,CNN_VS的富集能力比独立的CNNaffinity或CNNscore高。如图3所示,CNN_VS在3种α参数下的BEDROC均高于单独的CNNscore与CNNaffinity。如图4所示,当X%=0.5,1的时候,CNN_VS的EF均高于单独的CNNscore与CNNaffinity。
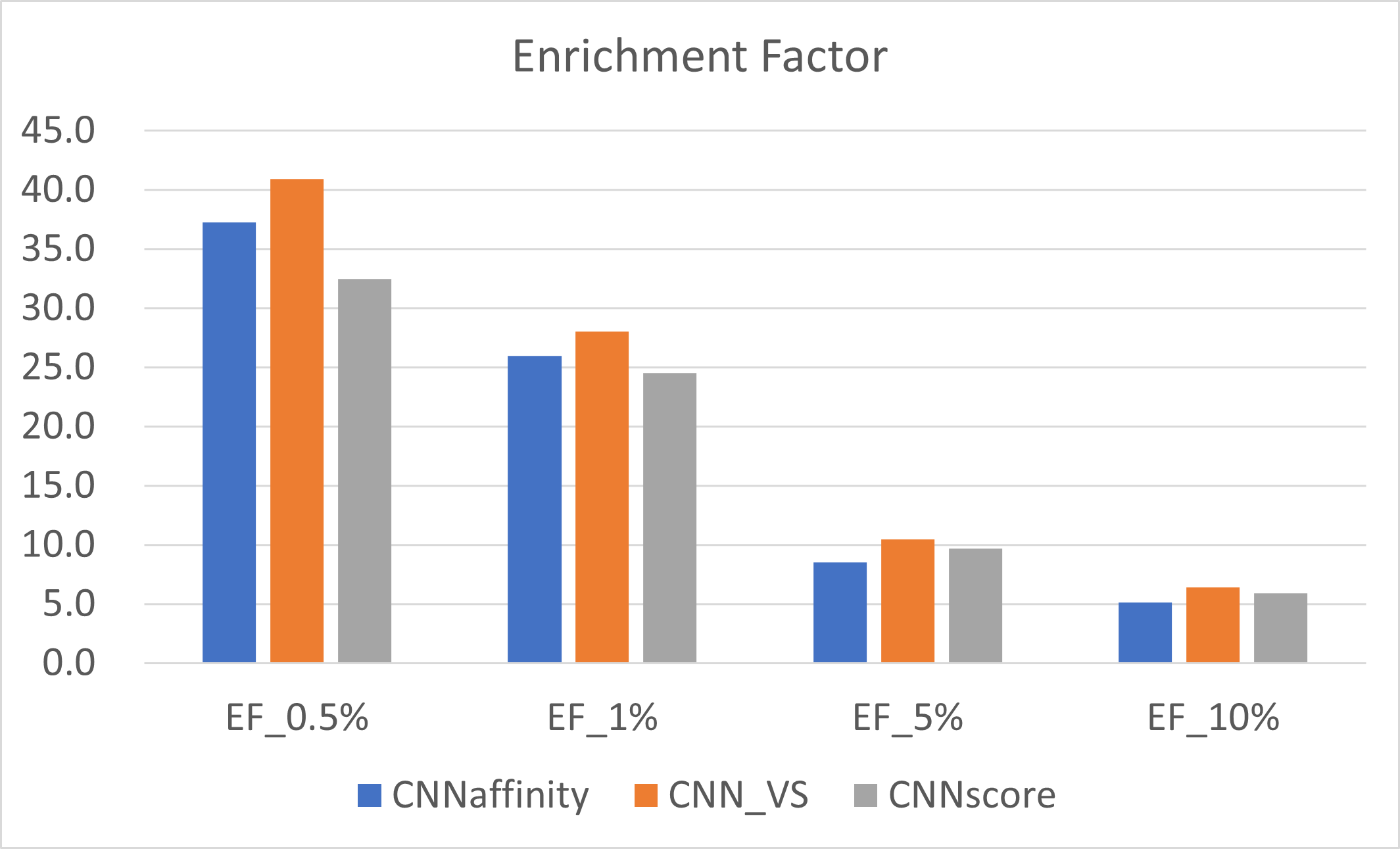
图4. CNNaffinity,CNNscore与CNN_VS重新打分的EFX%比较
比较LF VSscore、LF CNN VS、CNNaffinity,CNN_VS与Glide SP11的ROC曲线,如图5所示,可以发现LF VSscore、CNNaffnity与CNNscore相乘之后的LF CNN VS以及CNN_VS曲线在最右上角,优于其它的方法,接着是Glide SP与CNNaffinity,最下方是LF VSscore。
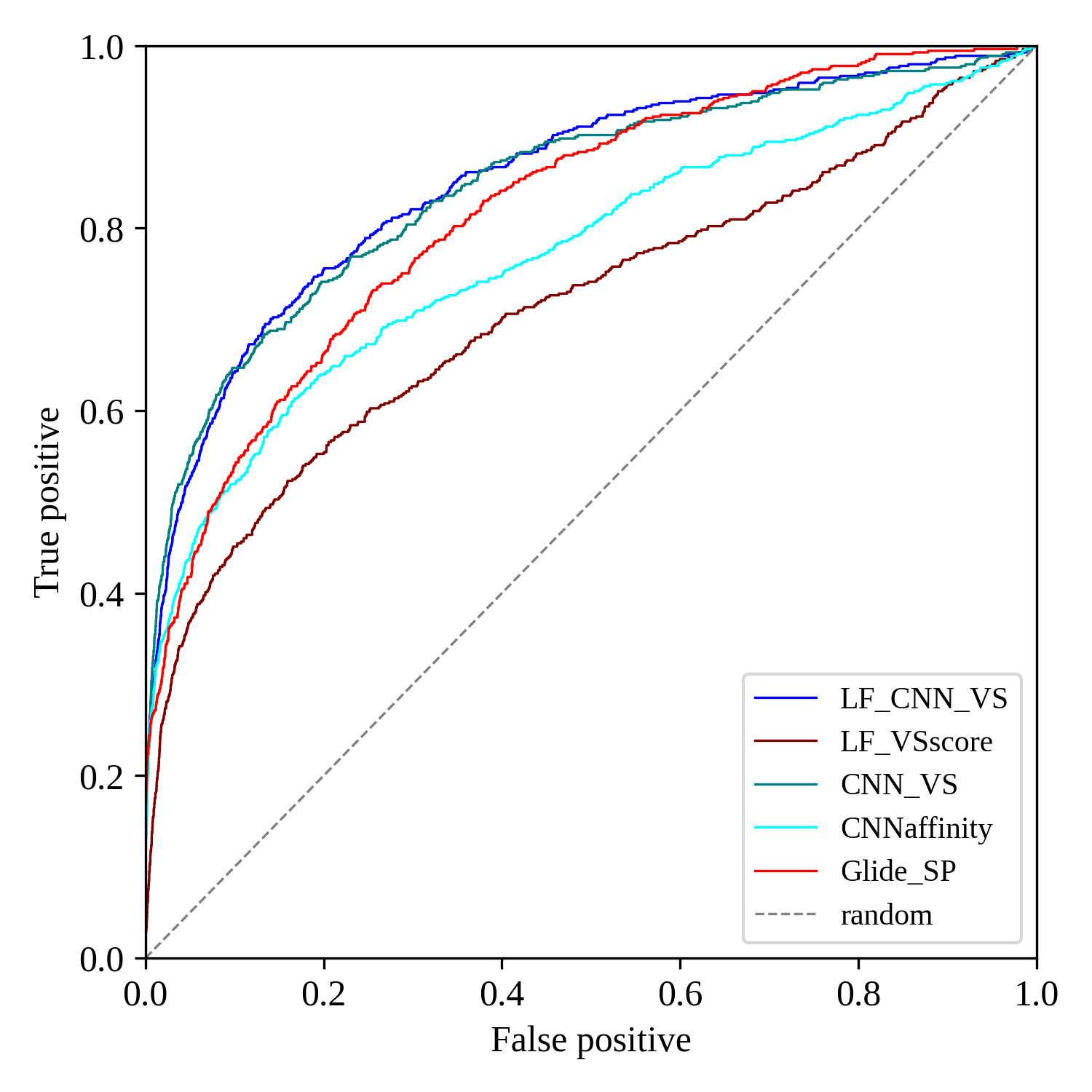
图5. LF VSscore、LF CNN VS、CNNaffinity,CNN_VS与Glide SP的ROC曲线
在ROC曲线中,在非常早期的阶段,LF CNN VS、CNNaffinity,CNN_VS与Glide SP的曲线重合在一起难以区分,图6的半对数ROC曲线可以放大早期阶段的差异。从图6可以看到,在非常早期(假阳性率从0.001-0.01阶段),四种打分函数经过一小段的交叉重合之后趋势翻转,不同的具体阶段性能有差异。具体的讲,在0.001-0.002区间,从高到低的顺序依次为Glide SP、CNN_VS、LF CNN VS与CNN affnity;在0.002-0.004区间,4种打分函数缠绕在一起;在0.004-0.01区间开始分开并翻转,从高到低的顺序依次为CNN_VS、LF CNN VS、CNN affinity与Glide SP。
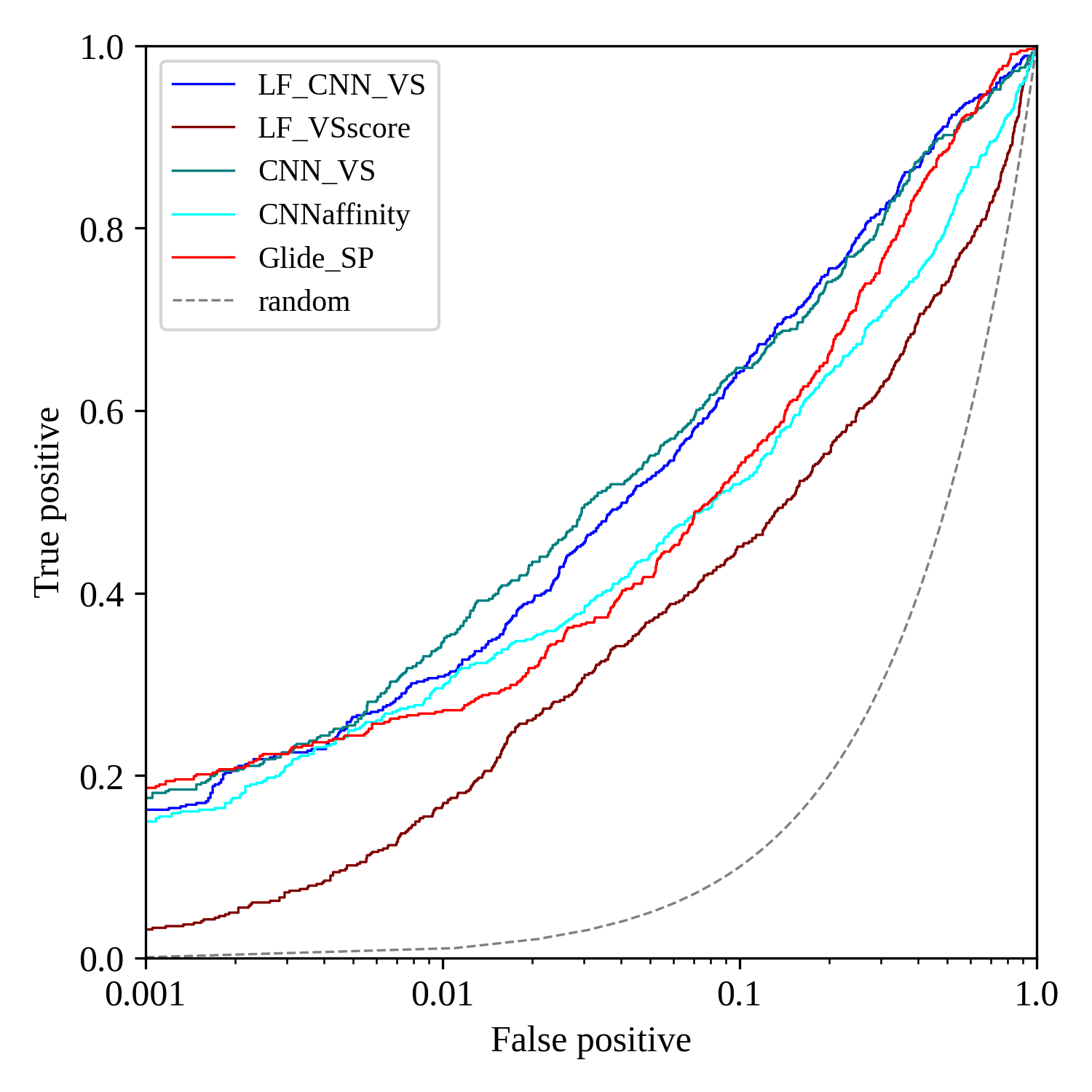
图6. LF VSscore、LF CNN VS、CNNaffinity,CNN_VS与Glide SP的半对数ROC曲线
CNNscore用于对接后处理:作为过滤器提高虚拟筛选的性能
如前所述,CNNscore是个Pose score,评估pose正确的概率。将CNNscore打分值低的pose过滤掉,有可能提高虚拟筛选的性能。接下来,将CNNscore小于0.7的pose从actives与decoys里过滤掉,仅对余下的Pose进行数据后处理,然后用LF_VSscore进行性能统计。
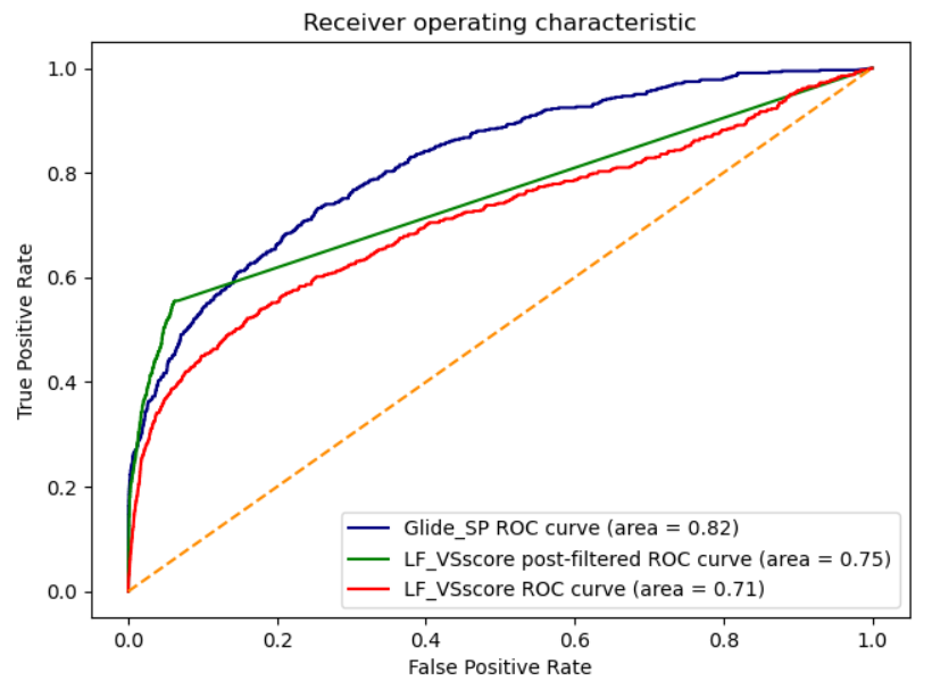
图7. CNNscore用于Pose过滤。LF VSscore、LF CNN VS、CNNaffinity,CNN_VS与Glide SP的ROC曲线
ROC曲线如图7所示,AUC表示的综合性能从高到低依次为Glide SP、CNNscore过滤后的LF VSscore,最后是未经CNNscore过滤的LF VSscore。我们更加关注早期富集能力,也就是假阳性率很低的那个阶段的富集能力。显而易见,经过CNNscore 0.7过滤的LF VSscore绿色曲线在最右上方,其次为红色的glide曲线在中间,最下方为为经过CNNscore过滤的LF VSscore。这说明,CNNscore过滤提高了LF VSscore的早期富集能力,性能不低于文献报道的Glide SP。
结论
本文以DUD-E的EGFR子集为例,在Flare Docking的虚拟筛选模式下对Actives与Decoys进行了分子对接计算,并进一步用GNINA的CNNscore与CNNaffinity进行再次打分,将Flare docking的VSscore与CNNscore进行相乘作为新的打分值,最后对不同的打分值进行了虚拟筛选性能评估,尤其是表示早期富集能力的BEDROC与EF值,并与文献报道的Glide SP进行了比较。
结果表明,Flare docking的LF VSscore与深度学习CNNscore相乘之后的LF CNN VS的富集能力优于各自独立的LF VSscore与CNNscore打分函数;同样,CNNaffinity与CNNscore相乘之后的CNN_VS的富集能力也优于各自独立的CNNaffinity与CNNscore。总的来说,CNNscore与Flare Docking的LF VSscore以及GNINA的CNNaffinity显示出协同增强虚拟筛选性能的能力。在DUD-E的EGFR这个子集上,LF CNN VS与CNN_VS在EBEDROC与EF等关键的虚拟筛选性能指标上与文献报道的Glide SP表现出相似的性能。
此外,GNINA的CNNscore也可以直接用来作为对接后处理的过滤器,在本文中,将CNNscore小于0.7的Pose过滤,可以显著地提供虚拟筛选的早期富集能力。
附件
本文涉及的数据文件下载链接: https://pan.baidu.com/s/1MH2nczRAyeUoUx4vFhXFNg
提取码: c246
参考文献
- a)Wang, Z., Sun, H., Yao, X., Li, D., Xu, L., Li, Y., Tian, S., & Hou, T. (2016). Comprehensive evaluation of ten docking programs on a diverse set of protein–ligand complexes: the prediction accuracy of sampling power and scoring power. Phys. Chem. Chem. Phys., 18(18), 12964–12975. https://doi.org/10.1039/C6CP01555Gb. b)十种分子对接软件的性能评估. http://blog.molcalx.com.cn/2017/02/08/evaluation-ten-docking-program.html.
- Lyu, J., Irwin, J. J., & Shoichet, B. K. (2023). Modeling the expansion of virtual screening libraries. Nature Chemical Biology, 19(6), 712–718. https://doi.org/10.1038/s41589-022-01234-w
- a)Fischer, A.; Smieško, M.; Sellner, M.; A. Lill, M. Decision Making in Structure-Based Drug Discovery: Visual Inspection of Docking Results. J. Med. Chem. 2021, 64 (5), 2489–2500. https://doi.org/10.1021/acs.jmedchem.0c02227. b)基于结构药物发现中的决策:对接结果的手工检查. http://blog.molcalx.com.cn/2021/05/22/visual-inspection-of-docking-results.html
- Ragoza, M., Hochuli, J., Idrobo, E., Sunseri, J., & Koes, D. R. (2017). Protein–Ligand Scoring with Convolutional Neural Networks. Journal of Chemical Information and Modeling, 57(4), 942–957. https://doi.org/10.1021/acs.jcim.6b00740
- McNutt, A. T., Francoeur, P., Aggarwal, R., Masuda, T., Meli, R., Ragoza, M., Sunseri, J., & Koes, D. R. (2021). GNINA 1.0: molecular docking with deep learning. Journal of Cheminformatics, 13(1), 43. https://doi.org/10.1186/s13321-021-00522-2
- Sunseri, J., & Koes, D. R. (2021). Virtual Screening with Gnina 1.0. Molecules, 26(23), 7369. https://doi.org/10.3390/molecules26237369
- Mysinger, M. M., Carchia, M., Irwin, J. J., & Shoichet, B. K. (2012). Directory of Useful Decoys, Enhanced (DUD-E): Better Ligands and Decoys for Better Benchmarking. Journal of Medicinal Chemistry, 55(14), 6582–6594. https://doi.org/10.1021/jm300687e
- DUD-E EGFR subset. https://dude.docking.org/targets/egfr
- Flare V7. https://www.cresset-group.con/software/flare
- 肖高铿. 如何进行虚拟筛选的方法学验证.墨灵格的博客. 2016-09-22. http://blog.molcalx.com.cn/2016/09/22/virtual-screening-methodology-validation.html
- Shen, C.; Hu, Y.; Wang, Z.; Zhang, X.; Pang, J.; Wang, G.; Zhong, H.; Xu, L.; Cao, D.; Hou, T. Beware of the Generic Machine Learning-Based Scoring Functions in Structure-Based Virtual Screening. 2020, 00 (April), 1–22. https://doi.org/10.1093/bib/bbaa070.
- Stroganov et al., Lead Finder: An Approach to Improve Accuracy of Protein-Ligand Docking,
Binding Energy Estimation, and Virtual Screening, J. Chem. Inf. Model. 2008; 48, 2371-2385.
联系我们,获取试用
立即申请免费试用版Flare以进一步探索其完整的分子建模功能组合。作为评估Flare性能的一部分,您将获得安装平台和访问其广泛功能的全面支持。您也可联系我们,开启线上会议为您演示在DUD-E其它靶标上的虚拟筛选性能效果。



