摘要:在本文中,我们演示了如何用Flare的QSAR建模功能,导入RDKit指纹与2D分子描述符用于机器学习方法SVM建立血脑屏障(BBB)透过性分类预测模型,评估模型的性能与可靠性,并用于新化合物的BBB透过性预测。
前言
在基于配体和基于结构的药物设计综合性软件平台Flare中,用户可以使用静电和形状3D描述符构建三维定量构象关系(3D-QSAR)模型。这些模型假设数据集中的配体与感兴趣的生物靶标活性位点相互作用时具有共同的结合模式。
然而,在许多情况下,设计者希望能为那些没有特定结合模式或结合事件的生物学特性构建QSAR模型。例如,他们可能想要使用一组结构多样的配体来模拟ADMET终点,其中可能不存在活性位点结合事件,或者这对于所有配体来说没有一致的结合模式。在这种情况下,需要使用配体的替代物理化学描述符,比如分子量(MW)、油水分配系数(logP)、拓扑极性表面积(TPSA)和可旋转键数量等之类的2D描述符。
Flare最近的更新完全集成了RDKit 2D描述符1的生成,并添加了使用RDKit指纹1进行QSAR模型构建的功能。在上一篇文章中,我们展示了用户如何通过Python脚本导入Flare的2D描述符来构建血脑屏障(BBB)渗透的QSAR模型。在本专题中,我们将重新审视这项研究,使用新的内置功能快速轻松地生成RDKit描述符。
QSAR模型的类型
本研究使用的BBB数据集取自Roy等人的工作2,是一个分类数据集,其中未给出连续的BBB透过性测量值。报告的响应值是一个二元分类:分子要么能够通过BBB给出值1,要么不能通过给出值0。因此,我们用这些分类建立一个分类QSAR模型。
导入RDKit描述符用于构建QSAR模型
首先,将完整的数据集分为训练集和测试集,训练集包含总数据集的70%(1284个配体),测试集包含数据集的剩余30%(551个配体)。 接下来,单击“Column & Activity Editor”中的“Import RDKit descriptors”按钮生成RDKit 2D描述符(图 1)。您可以通过选择“Select All”来导入所有RDKit描述符,或者通过勾选选择框来仅导入您认为必要的描述符。
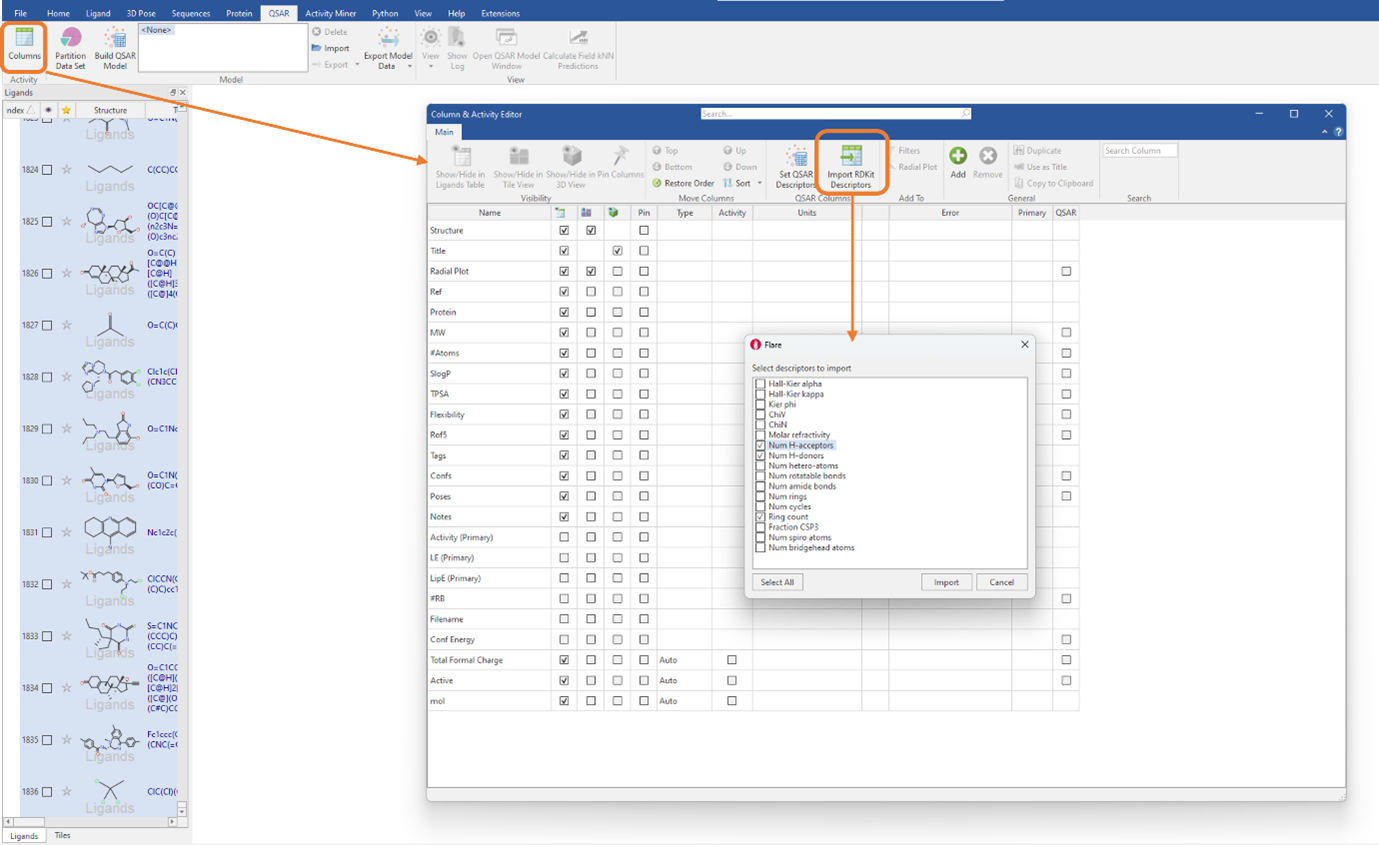
图1. RDKit 2D物化描述符可以通过单击“Column & Activity Editor”按钮导入。
您选择导入的每个RDKit描述符都将作为分子属性列添加到配体表单中。要使用这些描述符来构建QSAR模型,我们需要在表单中选择描述符,然后在“Column & Activity Editor”中单击“Set QSAR Descriptors”按钮(图 2)。在本实验中,我们使用MW、SlogP、TPSA、Flexibility、#RB、Total Formal Charge、NumHAcceptors、NumHDonors和RingCount等描述符。后三个描述符是之前从RDKit导入的描述符(图 1)。
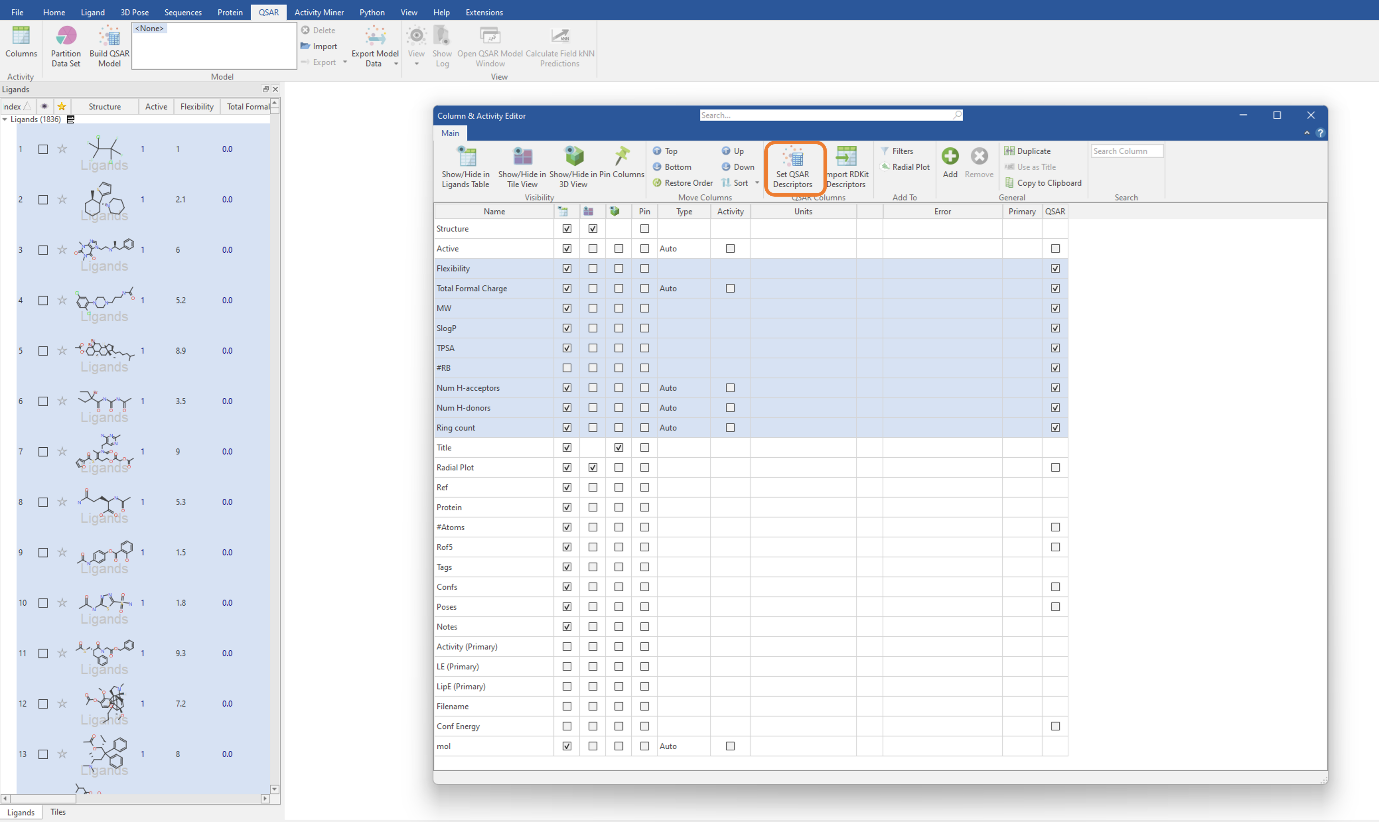
图2. 选择想在分类QSAR模型中使用的RDKit描述符以便导入
为了与之前的研究具有可比性,我们联合使用上面列出的Flare生成的描述符以及从RDKit导入的描述符来构建SVM分类QSAR模型(图 2)。 我们需要确保“静电”和“体积”复选框未被选中。此外,这里值得注意的是,由于我们没有使用静电和形状场点3D描述符,因此我们的配体数据集不需要进行叠合对齐(图 3)。
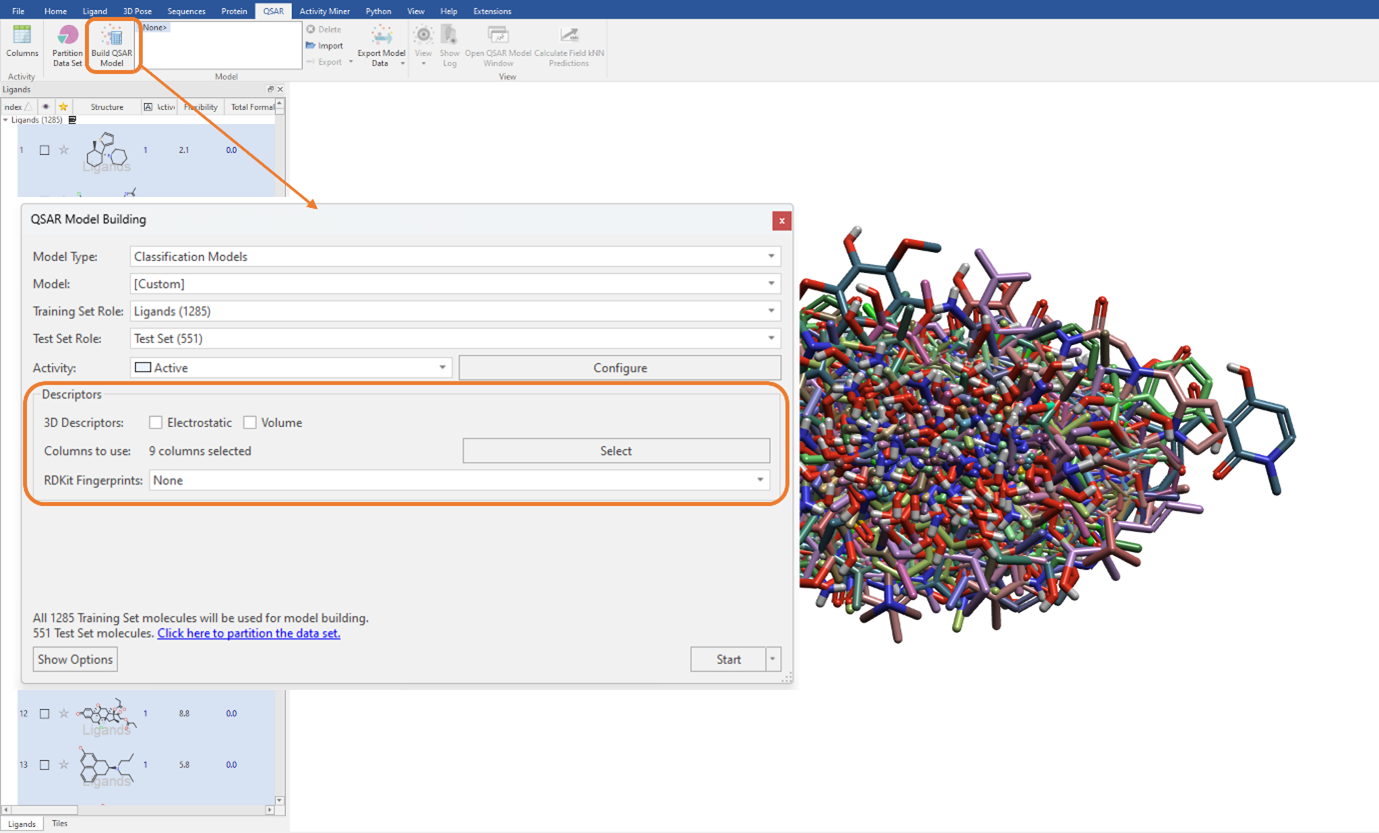
图3. 联合使用Flare生成的描述符与从RDKit导入的描述符建立SVM分类模型
我们可以使用无限最大优化器迭代次数来执行QSAR建模计算,这是大型数据集的默认设置,但可以在“Show Options”菜单中手动编辑(图 3)。分类QSAR模型的性能,即分类模型正确预测测试集中哪个配体被分配到类别0或1的能力,由测试集混淆矩阵总结(图 4)。非对角线元素显示错误预测的频次,即属于类别0的配体被分配给类别1,反之亦然。非对角线值为零表示分类QSAR模型将测试集中的每个配体100%分配为正确的类别。
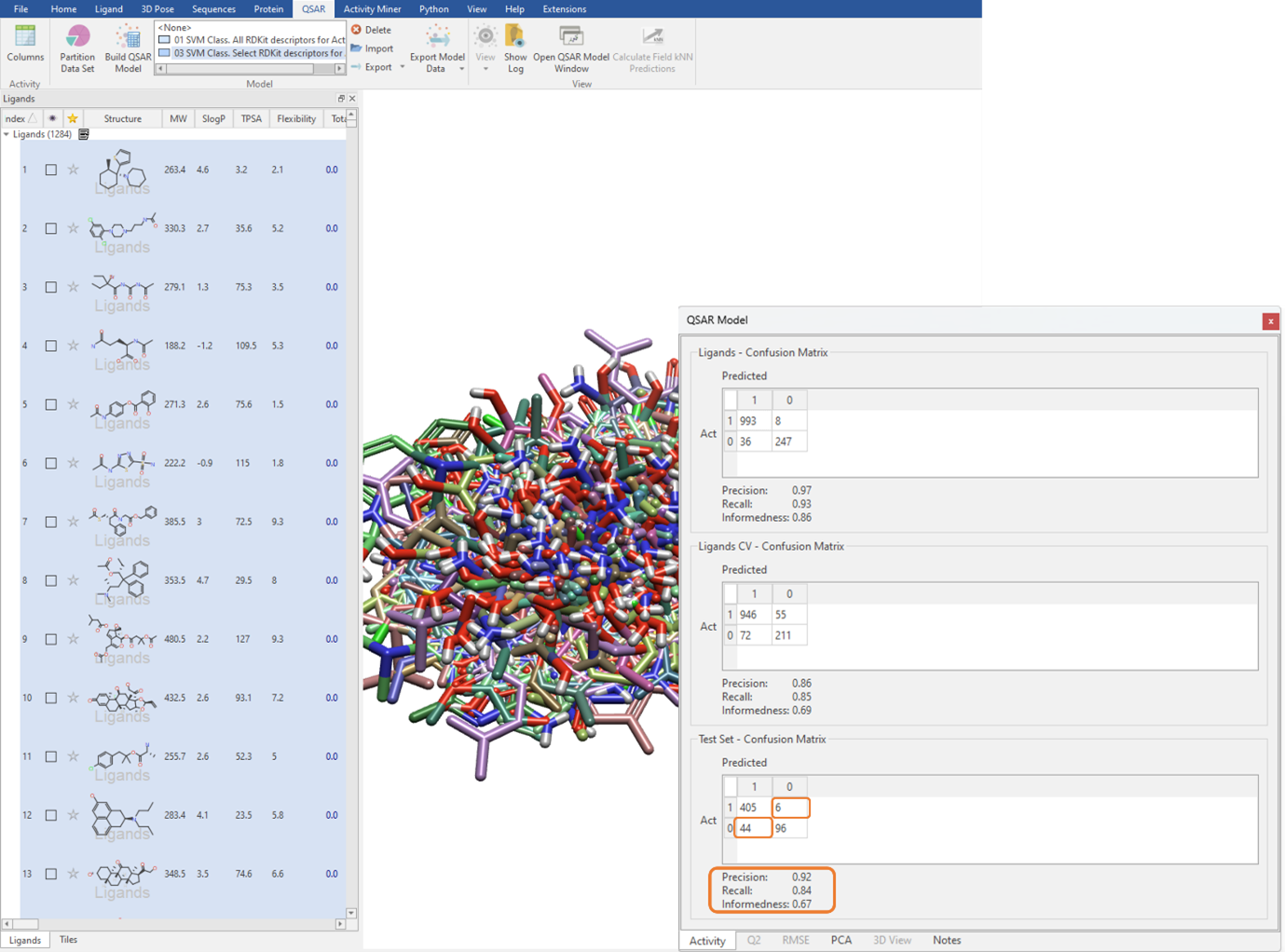
图4. 用测试集的混淆矩阵来识别分类QSAR模型的预测能力
精确度(Precision)、召回率(Recall)和信息度(Informedness)是从混淆矩阵得出的其他统计指标(图 4)。 精确度描述了正确分类的比例。召回率进一步细化了这一点,并报告给定类别的正确分类分配的比例,本质上是正确的类别1预测的数量除以类别1配体的总数。信息度描述了做出明智决策而不是随机猜测的概率。所有这些值越接近1,我们对分类QSAR模型的信心就越大。
QSAR模型在测试集上的精确度为0.92,召回率为0.84,信息度为0.67(图 4),我们可以有统计信心部署此分类QSAR 模型用于预测新配体BBB可渗透或不可渗透的分类。此外,从测试集-混淆矩阵中可以看出,6个活性配体被预测为非活性,44个非活性配体被预测为活性。
结果表明,尽管我们的预测出现了44个假阳性,但只有少量(6个)假阴性。这意味着,考虑到BBB渗透性是我们项目的理想特性,在确定进行测试分子的优先级排序时,我们错过BBB渗透且为项目感兴趣分子的可能性很低。
用RDKit指纹建立QSAR模型
在图3中,您可以看到使用RDKit指纹作为描述符来构建QSAR模型的选项。RDKit指纹是用于评估分子相似性的子结构指纹。通过选择此选项,可以使用这些指纹描述符来补充或代替当前选择的QSAR描述符。同样,与2D物理化学描述符(图 1)一样,只需从“RDKit Fingerprints”下拉菜单中选择所需的选项(图 5),即可无缝地将其用于模型构建。当前的选择包括我们将在本示例中使用的“RDKit”指纹,以及摩根指纹和MACCS密钥。
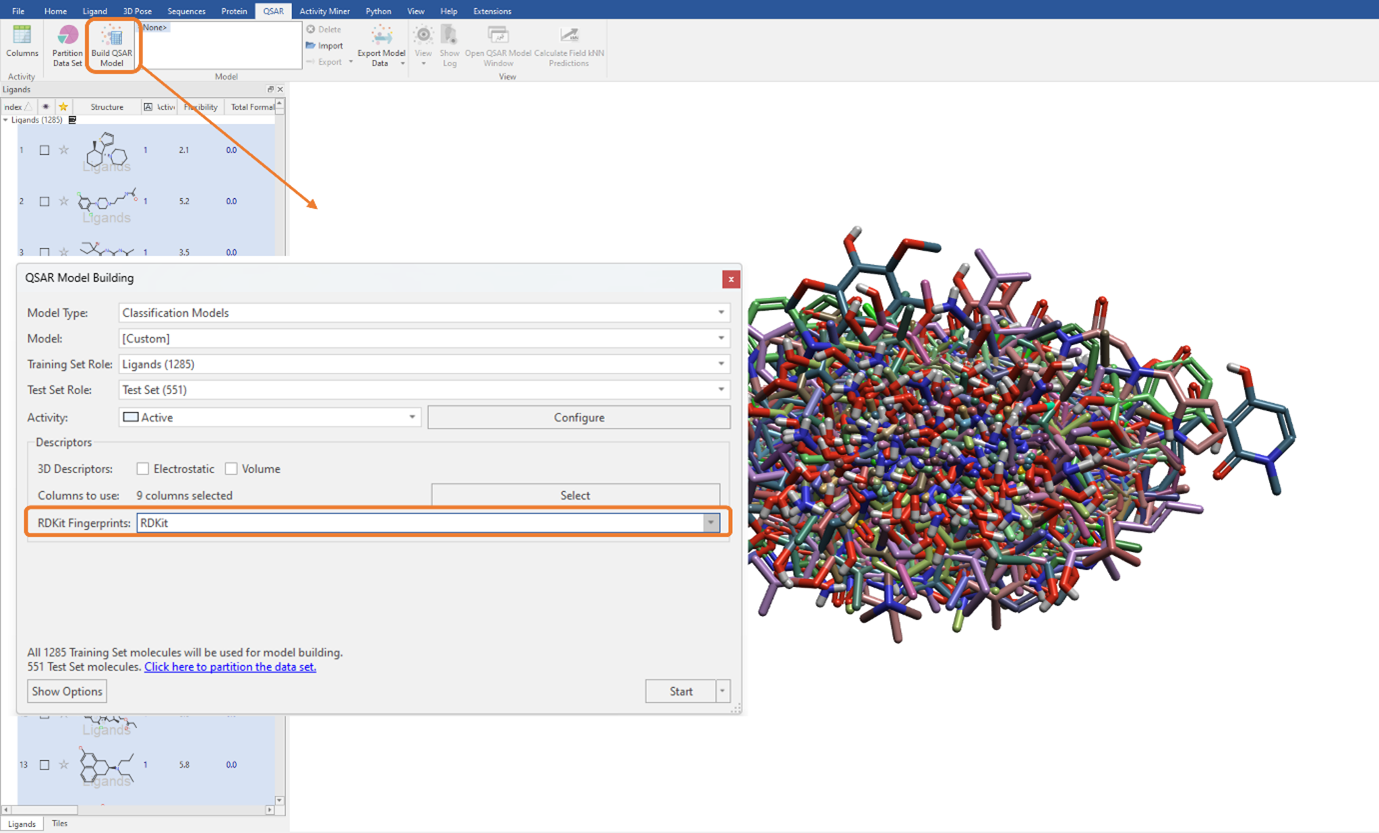
图5. 用RDKit指纹建立分类QSAR模型。注意,我们去掉了对3D与2D描述符的勾选(‘Columns to use’设置为0)并且仅使用RDKit指纹用于QSAR建模
使用RDKit指纹构建分类QSAR模型后,我们获得了0.90的精确度、0.86的召回率和0.72的信息度。这表明与使用RDKit 2D描述符构建的分类QSAR模型相比,使用RDKit指纹的模型预测BBB渗透性的能力略有提高。
结论
在Flare中可以快速、轻松地导入RDKit 2D描述符和指纹并用于QSAR建模。在此示例中,这些描述符成功用于构建分类QSAR模型以及预测配体的BBB渗透性。RDKit描述符或RDKit指纹为静电和形状的Cresset 3D描述符提供了替代方法,可用于构建生物活性和ADMET性质的预测性QSAR模型。
参考文献
- Getting Started with the RDKit in Python — The RDKit 2023.03.1 documentation. https://www.rdkit.org/docs/GettingStartedInPython.html#list-of-available-descriptors (accessed 2023-07-31).
- Roy, D.; Hinge, V. K.; Kovalenko, A. To Pass or Not To Pass: Predicting the Blood–Brain Barrier Permeability with the 3D-RISM-KH Molecular Solvation Theory. ACS Omega 2019, 4 (16), 16774–16780. https://doi.org/10.1021/acsomega.9b01512.
联系我们,获取试用
立即申请免费试用版Flare以进一步探索其完整的分子建模功能组合。作为评估Flare性能的一部分,您将获得安装平台和访问其广泛功能的全面支持,同时可以自由发布生成的任何结果并将其用于进一步的研究。



