摘要:在本文中,我们演示了如何用即将发布的Flare V5增强的QSAR建模功能,使用Flare Python拓展包RDKit计算分子描述符,建立血脑屏障(BBB)透过性分类预测模型。
作者:Giovanna Tedesco, Ryuchiro Hara.(2021/04/27). Building a blood-brain barrier penetration model in Flare. Retrieved from https://www.cresset-group.com/about/news/bbb-penetration-model-flare.
编译:肖高铿/2021-04-29
前言
基于配体的设计平台Forge提供了Field QSAR和机器学习方法,使用基于静电的分子场和空间特性描述符来表征分子并建立生物活性的3D-QSAR模型。此类描述符在特定的配体-蛋白质相互作用建模方面非常高效并得到定量模型,将配体的化学结构与靶标的生物活性关联起来。
然而,当建立不涉及直接配体-蛋白质相互作用,而是被动扩散机制(例如BBB渗透)的ADMET性质的QSAR模型时,分子量(MW)、极性表面积(PSA)和亲脂性(即SlogP)等等的整体分子性质可能更合适。事实上,许多实践者希望进一步扩展这些特性,利用计算的、实验的或自定义的性质来建立QSAR模型。虽然这在Forge中是不可能的,但在Flare V5中将是可能的。即将发布的版本将包含Forge所有功能,并通过Flare的Python API使用,以支持更复杂的QSAR工作流。
数据集
本研究所用的数据集取自Roy等人[1]的文章。在该文章中,BBB透过性数据从不同来源收集而得,包含了1866个化合物。其中1438化合物分类为BBB透过性(BBB+,Class 1);428个化合物分类为非BBB透过性(BBB-,Class 0)。作者将初始的数据集划分为含1400个化合物的训练集(training set)以及含466个化合物的测试集(test set)。
将这些化合物导入到Flare,生成含1376个化合物的训练集(1058个为BBB+/Class 1,318个为BBB-/Class 0)以及含459个化合物的测试集(354个为BBB+/Class 1,105个为BBB-/Class 0)。一些无效SMILES的分子没被导入。
初步的模型
初步的支持向量机(SVM)分类模型仅使用Flare内置的描述符构建,即MW、SlogP、拓扑极性表面积(TPSA)、柔性、可旋转键数和总电荷数。 由于数据集很大,如QSAR Model Building小部件中的策略警告所建议的(图1),优化器迭代的最大数量增加到1000。其他选项均使用默认值。
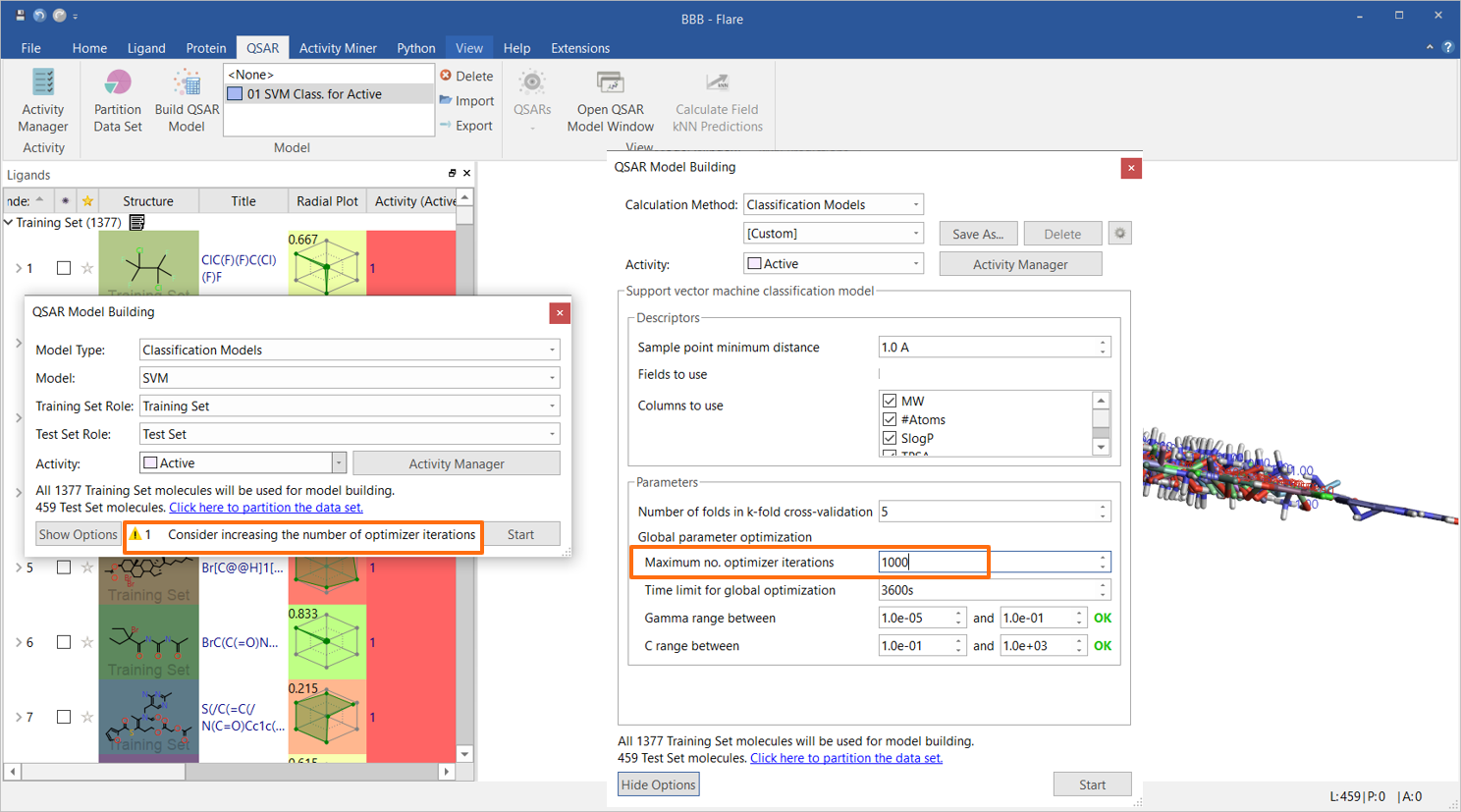
图1. 初步的SVM分类模型用6个Flare内置的描述符构建
混淆矩阵总结了初始模型的性能(图2-左)。 混淆矩阵显示训练集数据的实际类别与模型预测的类别之间的预测频数。如果所有非对角线值均为零,则模型预测都是正确的。从该矩阵得出的其他有用的统计信息如下:
- 精确度(Precision):预测正确的比例。比如,在模型预测为“Class 1”的化合物中,有多少个化合物被正确预测?Precision值的范围从0(意味模型预测完全不对)到1(意味着完美预测)。
- 召回率(Recall):某一类别被正确识别的比例,比如被正确识别为Class 1类别的分子除以Class 1类分子的总数。Recall值的范围从0(意味模型预测完全不对)到1(意味着完美预测)。
- 信息性(Informedness):做出明智决策而非随机猜测的概率,该值范围从0(意味模型预测仅仅与随机猜测一样)到1(表示完美预测)。
混乱矩阵中报告的统计信息是每个类别的各自统计信息的平均值,您可以在项目日志中查看这些统计信息(图2-右)。
对于机器学习模型,训练集/训练集CV的统计数据往往过于乐观。模型的实际预测能力取决于它在测试集上的性能,测试集是一组不包含在模型构建中的分子。
模型对测试集(图2-左)预测结果表明,精确度为0.87,召回率为0.83,信息性为0.66。
模型对测试集预测的精度为0.87、召回率为0.83、信息量为0.66(见图2-左)。这个非常简单的模型已经具有相当好的能力,如图2所示,能够将测试集中的化合物正确地分类为BBB+(class 1)和BBB-(class 0)。这完全并不令人惊讶,因为我们将诸如MW、SlogP、TPSA和总电荷数等等与被动扩散过程有关的描述符用进来了。
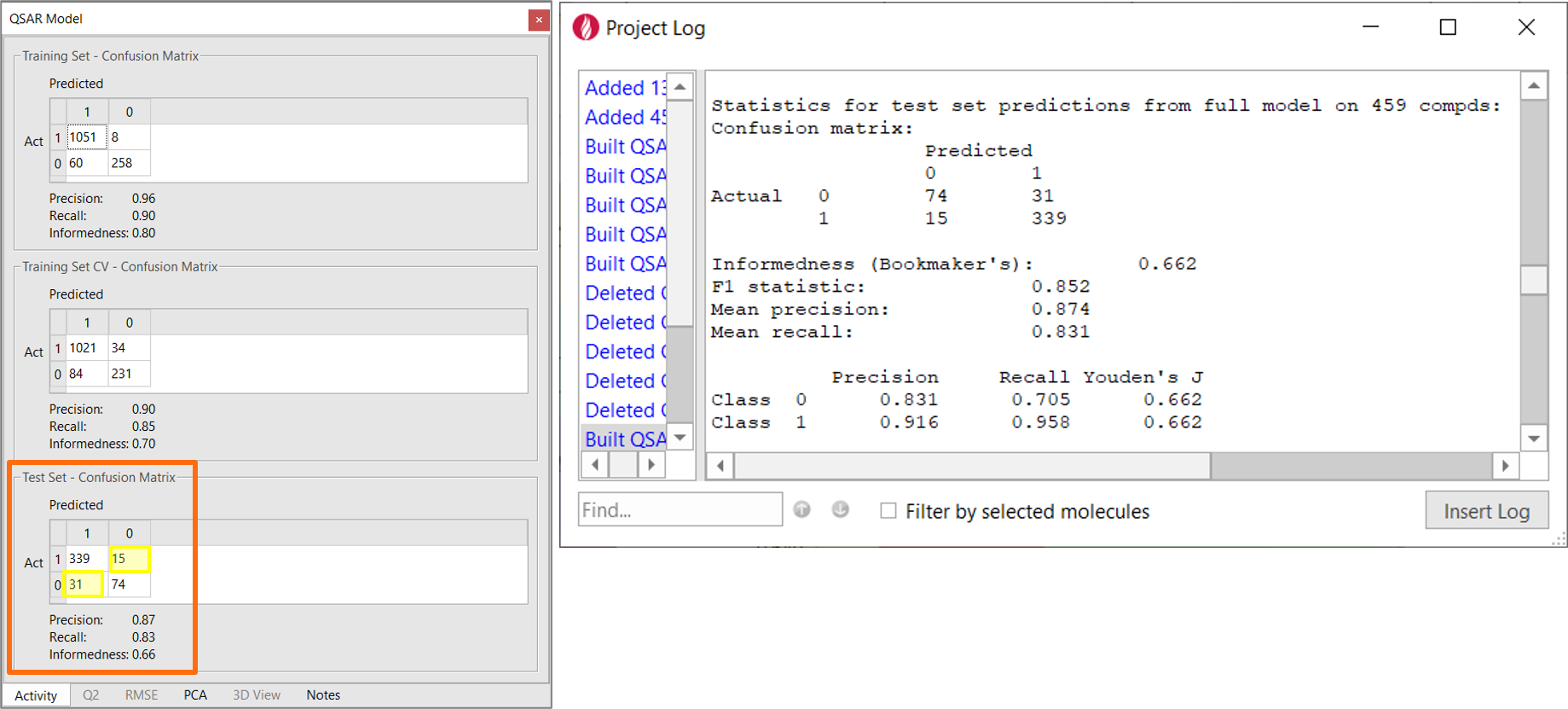
图2. 左:混淆矩阵展示了用Flare内置描述符建立的BBB透过性模型的分类性能。黄色方框里的数值为模型不正确分类的化合物数。右:Flare项目日志里报告的QSAR模型信息。
在Flare中导入自定义的描述符
尽管亲脂性、分子量和极性表面积是决定BBB透过性的重要因素,但如文献所述,众所周知地,诸如氢键供体和受体总数等其它性质也可能起作用。更有效的ADMET性质建模可能还需要使用自定义性质,例如计算的或实验的pKa或LogP/LogD、HOMO、LUMO和去溶剂化等等特性。
当然,可以分别计算这些描述符,然后再导入Flare,但这是一个手动操作且繁琐的过程。Flare Python API可以让这类工作流程自动化,让创建Flare“扩展”变得非常容易。这些扩展将Flare与您喜爱的物化性质计算器连接起来,或者从公司数据库中提取实验数据。
在本研究中,Flare的“Generate features for machine learning”拓展包(图3)可以用来导入RDKit计算的描述符以及其它来源的描述符,将导入的描述符添加为Flare项目的一列。
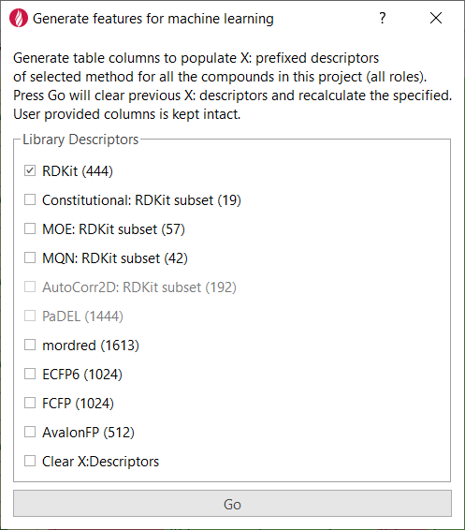
图3. Flare Python API让你写个自定义的Python拓展变得轻而易举
优化BBB透过性模型
导入RDKit的描述符之后,就可以用下述描述符建立新的SVM模型:MW, SlogP, TPSA, Flexibility, #RB, Total Formal Charge, NumHAcceptors, NumHDonors, RingCount。前6个描述符 (MW, SlogP, TPSA, Flexibility, #RB, Total Formal Charge)用Flare计算;后三个NumHAcceptors, NumHDonors与RingCount用RDKit计算。
优化器迭代最大次数设置为1000,其它的参数用默认值。新获得的模型(见图4)性能有所提升。
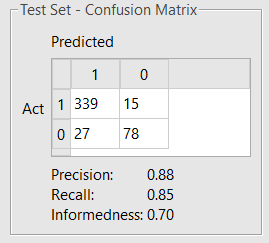
图4. 混淆矩阵展示了用Flare内置描述符与RDKit描述符混合优化过的BBB透过性模型对测试集的分类预测性能
若以更高的模型复杂度与过度训练风险为代价,用444个RDKit描述符进行训练,可进一步提升模型对测试集的预测性能:Precision 0.91, Recall 0.86, Informedness 0.73。
总结
Forge的QSAR方法将完全集成到定于2021年夏季发布的Flare V5版本中,具有处理自定义描述符的新功能。Flare Python API使您能够轻松创建扩展,将Flare连接到您喜爱的理化性质计算器上,或从公司数据库中提取数据。这两个特性可以很容易地构建ADMET性质预测性QSAR模型(比如,用自己的数据集建立BBB透过性预测模型)。
文献
- Roy, D.; Hinge, V. K.; Kovalenko, A. To Pass or Not to Pass: Predicting the Blood-Brain Barrier Permeability with the 3D-RISM-KH Molecular Solvation Theory. ACS Omega 2019, 4 (16), 16774–16780. https://doi.org/10.1021/acsomega.9b01512.



