摘要:本文详述了如何以MTRX849系列化合物为起始分子,以docking为打分函数,用Spark虚拟筛选VeryCommon片段数据库对MTRX849母核进行替换并引入新的相互作用。本算例的结果表明,Spark docking打分函数可以利用蛋白的信息进行骨架跃迁发现具有新相互作用模式的配体。本算例命中的母核恰巧就是百济神州专利WO2021058018母核,虚拟筛选命中的化合物为该专利的实施例32,对KRASG12C的IC50=39.5nM。这进一步证实了本算例采用的Spark docking策略的可靠性。
肖高铿/2021-04-26
关于MRTX849
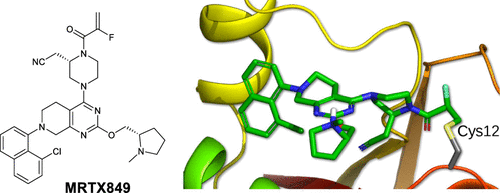
图1. MRTX849的化学结构及其与KRAS的复合物结构(右,PDB 6UT0),图片来源自Jay B Fell等人的文章[1]。
MRTX849(商品名为Adagrasib)的化学结构如图1所示,是一种口服有效、高选择性的KRASG12C共价抑制剂[1],可选择性地修饰KRASG12C中突变的CYS12残基并抑制KRAS依赖性信号传导。MRTX849通过将KRAS分子不可逆地锁定在其非活性状态来最大化抑制作用,从而防止肿瘤细胞生长并导致肿瘤细胞死亡。与现有的KRAS突变体选择性抑制剂相比,MRTX849的半衰期和进入肿瘤的渗透率显著提高,在整个给药期间中都关停了KRAS信号传导,并显示出更高的抗肿瘤活性。

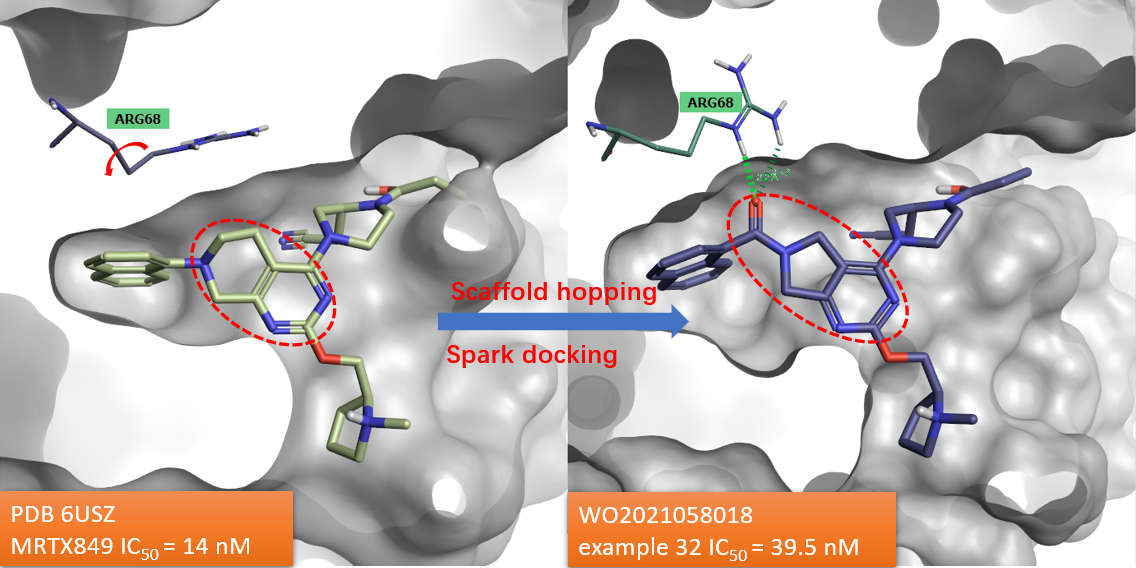
图2. MRTX849同系物7(左)与12a(右)的化学结构,它们与KRAS的复合物结构分别为PDB 6USX与6USZ。其中阴影覆盖部分是这类化合物的公共母核,也是骨架跃迁进行的部分。
Jay B Fell等人[1]发布了MRTX849及其同系物7、12a(见图2)与靶标KRAS的复合物晶体结构,其PDB代码分别为6UT0、6USX与6USZ。这些结构生物学数据为我们对母核(图2中阴影覆盖的部分)进行骨架跃迁提供了起始分子的生物活性构象,并为基于结构的方式进行片段虚拟筛选提供了结构生物学基础。在博文《从活性位点里发现新的蛋白-配体相互作用》[2]里已经详细介绍了Spark的docking方法,并展示了Spark docking在引入新相互作用的能力。本文将使用同样的方法用来设计新的KRASG12C共价抑制剂。
蛋白结构的准备与分子对接的策略:受体立体碰撞消除(SCAR)法
SPARK docking采用Lead Finder作为分子对接计算引擎对片段库进行虚拟筛选。然而Spark Docking并不自动执行共价对接策略,为了避免Michael受体与Cys侧链发生立体碰撞而docking失败,因此本文采用了受体立体碰撞消除(SCAR)法作为共价抑制剂的分子对接策略,具体描述请参见博文《用常规分子对接进行共价抑制剂的虚拟筛选》[3]。总的来说,我们将与配体共价结合的Cys突变为Gly,以便在蛋白结合位点Cys附近里留出足够空间并避免共价抑制的Michael受体与Cys侧链发生立体碰撞。在本算例中,突变用Flare | Protein | Mutation来完成,常用的分子模拟软件基本都可以实现突变操作。
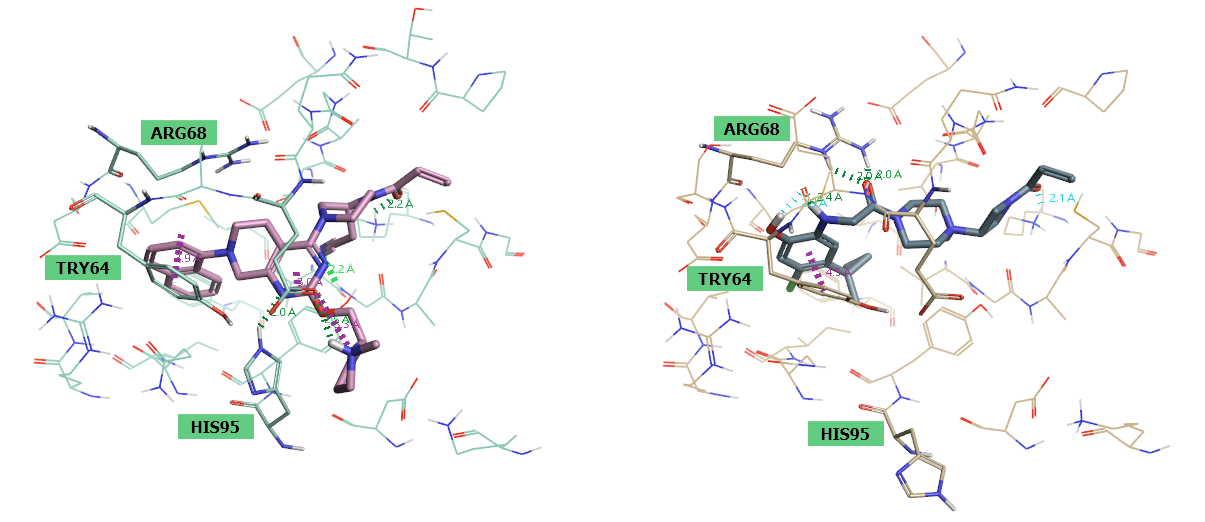
图3. 不同结构类型共价抑制剂结合口袋的比较。左:PDB 6USZ;右:PDB 5F2E。
KRAS结合口袋相当柔性,分析KRAS与不同共价抑制剂的共晶结构会发现:与不同类型的抑制剂结合时,结合口袋的形状与相互作用模式也不同。如图3所示,6USZ代表的MXTR849系列化合物母核附近的结合口袋残基ARG68、TRY64、HIS95的侧链与5F2E中对应的侧链在构象上有很大的差异。其中,5F2E中的ARG68与配体羰基形成氢键相互作用,而在6USZ中该残基不仅取向不同而且与共晶化合物12a也不发生任何极性的相互作用。5F2E共晶配体中的酰基哌嗪环与6USZ共晶配体的母核在空间位置有点重合,这意味着在对MXTR849母核替换的时候,有机会引入与ARG68发生相互作用的新片段。
为了让结合位点ARG68残基有机会与新片段发生氢键相互作用,需要对ARG68的侧链构象进行调整以确保与5F2E中ARG68有相似的侧链构象。在本算例中,我们用Flare | Protein | Edit对该残基进行构象调整。此外,还去除掉蛋白中所有的水分子、金属原子以及其它配体分子。
用Docking作为SPARK打分函数筛选片段库
鉴于MXTR489系列化合物与KRAS的结合模式相同,7、12a以及MXTR849中任意一个都可以作为起始分子来进行骨架跃迁。在本算例中,以12a(PDB 6USZ)为起始分子,并定义了替换片段连接点的原子类型(见图4左上角)。
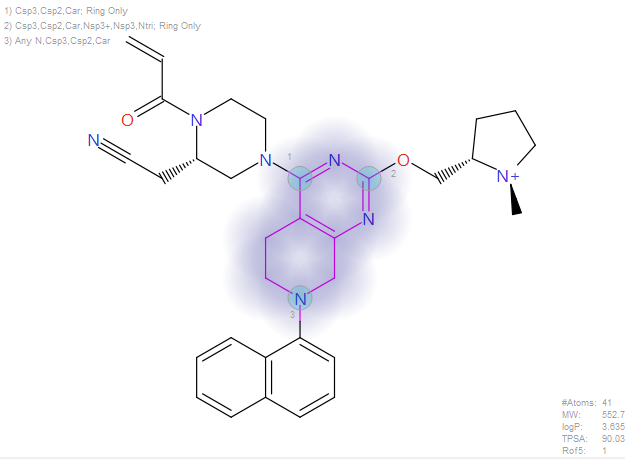
图4. 起始分子与被替换的片段,左上角的文字给出了片段连接点的原子类型。
将6USZ中的Cys突变为Gly、并去除全部的水与其它配体、调整ARG68侧链构象与5F2E中的大体一致后,将CHAIN A蛋白作为docking的受体蛋白,共晶配体12a作为起始分子。然后用SPARK(docking作为打分方法)对VeryCommon数据库进行虚拟筛选(见图5)。选用Very common数据库主要是因为该数据库含有的片段数相对较少,可以在最短的时间内得到计算结果。同时,该库的片段非常常见,每个片段至少在725种不同的分子里出现过,更具体的数据库介绍请阅读博文《SPARK数据库》[4]。
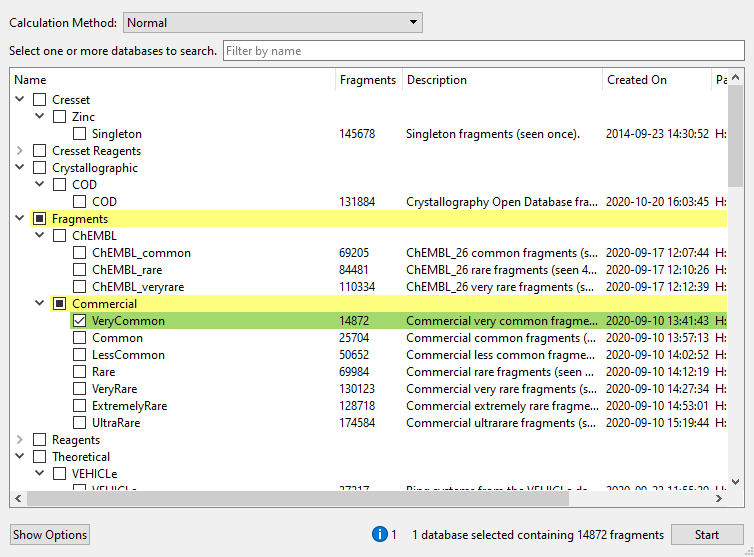
图5. 用SPARK对VeryCommon数据库进行虚拟筛选
结果
本次计算命中了36个化合物,部分化合物的母核及其打分值如图6所示。
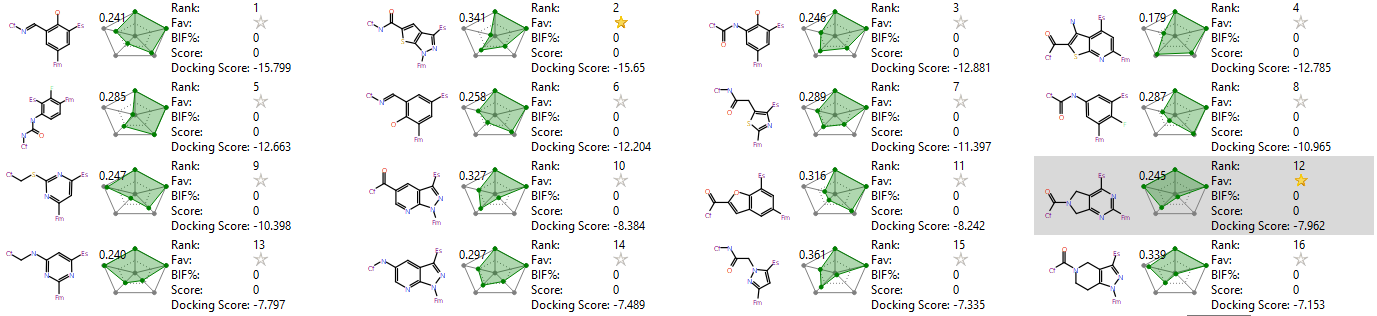
图6. 部分命中的化合物
我们对排名第12的化合物(图6灰色高亮)感兴趣,因为它具备了我们预设的相互作用特征与类药性质。如图7所示,与起始化合物12(左)相比,SPARK虚拟筛选命中的化合物(右)通过新片段与ARG68的氢键相互作用。同时,该命中化合物具有良好的构象稳定性,因为各个两面角都处于概率分布的高频区间。
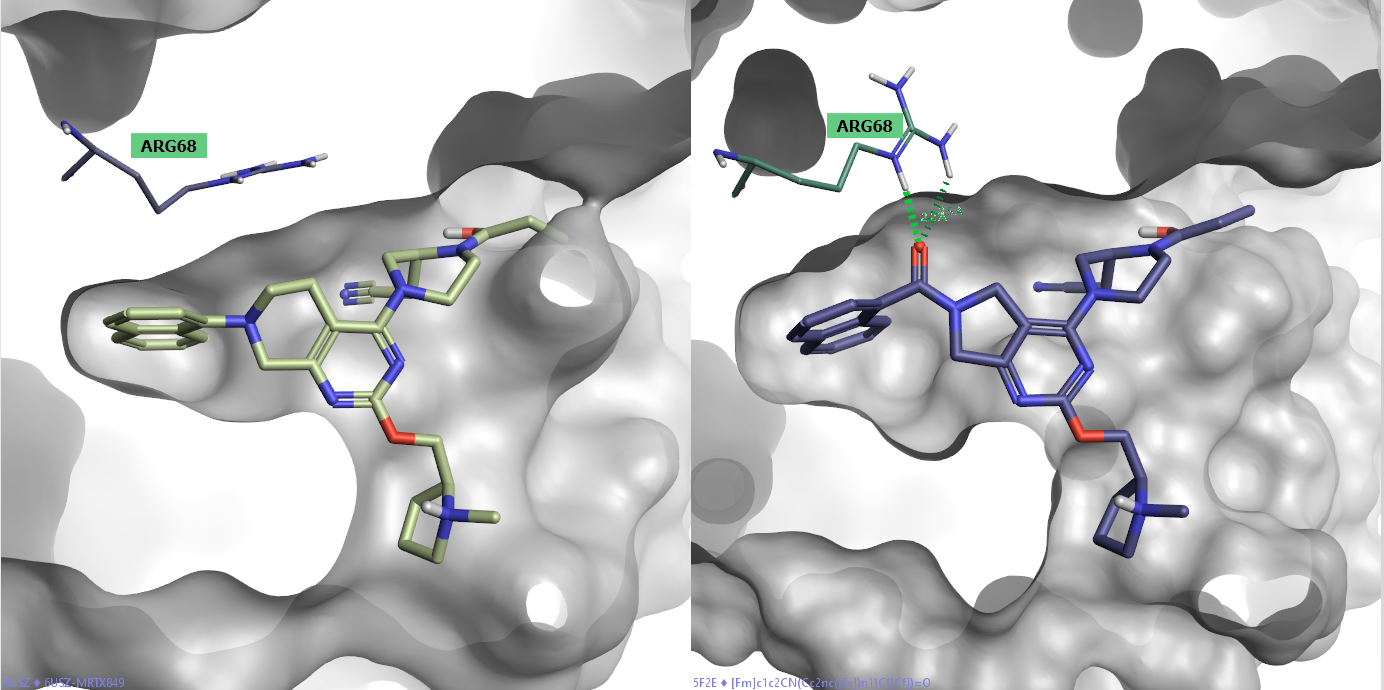
图7. 与起始化合物(左)相比,SPARK虚拟筛选命中的化合物(右)通过羰基引入了与ARG68相互作用的氢键
实际上,如果进一步用与ARG68发生氢键相互作用作为条件,如图8所示,命中的化合物会进一步被过滤为4个化合物。原先排名12的化合物调整为第2,可以被药物化学科研人员重点关注到。
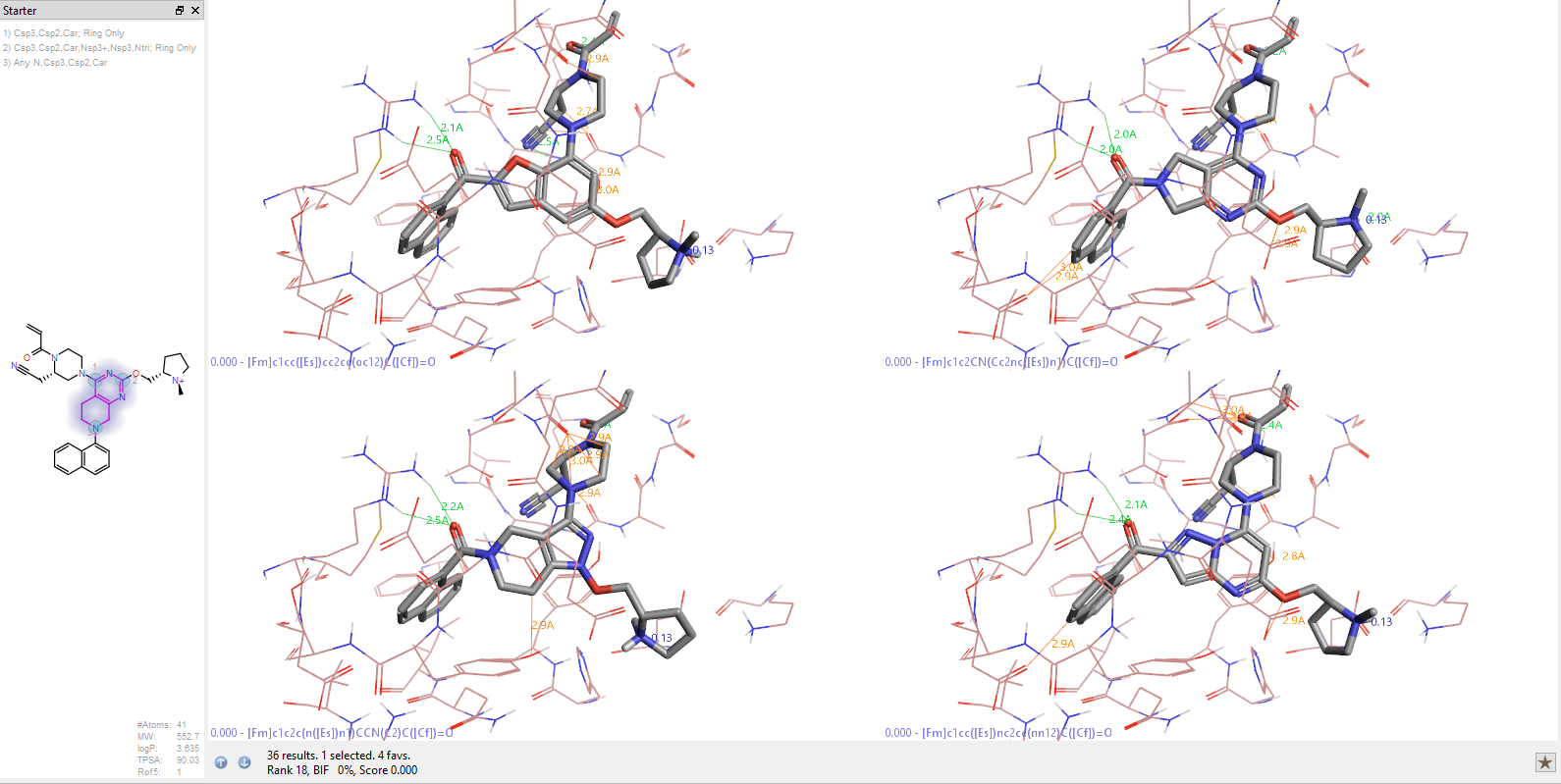
图8. 用ARG68的氢键相互作用作为过滤条件,命中化合物缩减到4个不同的新骨架
还可以进一步用已知的相互作用信息对图8的4个骨架进行排序。比如左上角化合物的苯并呋喃环不与TRY64或HIS95发生极性的氢键作用,排序则被往后调整;左下角化合物因为有N-O键而被排除在外,也可以在定义被替换片段query的时候对原子类型进行合理的约束以避免不合理的键的形成。虽然有N-O不合理的键存在,但这个化合物也提供了足够的idea:具有令人满意的母核,仅需进一步修改N-O键为一个合理键,这普通的药化人员在此提示之下很容易做到。这样,最终仅需考虑图8右边的两个骨架类型。实验与计算药物化学科研人员都可以通过可视化分析对结果进行优先级排序,获得对项目有价值的idea。
最近(2021年4月1日)公开的百济神州(BEIGENE LTD)专利WO2021058018[5]刚好以上述命中的母核为主要技术特征,并且命中化合物与实施例32(图9)完全一致,对KRASG12C的IC50=39.5nM,且其同系物也具有不错的活性。总的来说,百济神州的专利进一步证明了本算例采用的Spark docking策略的可靠性。
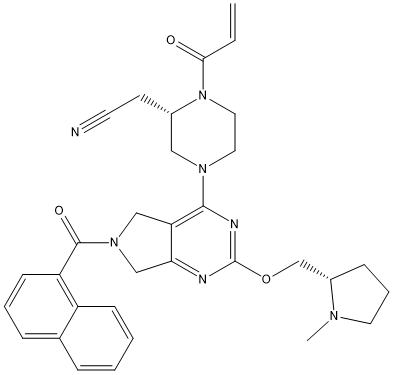
图9. WO2021058018的实施例32,KRASG12C的IC50=39.5nM
实际上,即使是在对完整的片段库进行虚拟筛选,该母核还会被命中并且排名靠前(原始的打分排名第74位),通过与ARG68相互作用等过滤可以进一步提高排名到前30而被药物化学科研人员优先关注到。
小结
本算例几个关键点总结如下:
- 通过比较不同抑制剂的结合位点,发现引入新的相互作用机会
- 受体立体碰撞消除(SCAR)法确保了常规对接方法可用于共价抑制剂的虚拟筛选
- 本算例的结果被最近公开的专利所验证
ARG68侧链与MXTR849系列化合物没有发生氢键相互作用,但在PDB 5F2E中该残基以截然不同的构象与抑制剂发生氢键相互作用。将5F2E的ARG68构象与6USZ的TRY64、HIS95构象组合,为MXTR849的骨架跃迁引入新相互作用提供了机会。
此外,OpenEye的Spruce高通量的蛋白结构准备与LigandScout的动态药效团技术(dynophore)擅长于从复合物晶体结构里发现新的相互作用,相信与Spark docking组合使用会得到意想不到的效果。
在本算例中,受体立体碰撞消除(SCAR)法将Cys突变为Gly以保证潜在的共价抑制剂有机会摆出正确的pose,避免了因为与Cys侧链的碰撞而产生假阴性。
我们感兴趣、排名靠前的母核恰巧是2021年4月1日公开的百济神州专利“KRAS抑制剂”的母核,而且命中的化合物也被实施例32完全一致并具有良好的活性,这进一步证实上述Spark docking的可靠性。
文献
- Fell, J. B.; Fischer, J. P.; Baer, B. R.; Blake, J. F.; Bouhana, K.; Briere, D. M.; Brown, K. D.; Burgess, L. E.; Burns, A. C.; Burkard, M. R.; et al. Identification of the Clinical Development Candidate MRTX849 , a Covalent KRASG12C Inhibitor for the Treatment of Cancer. J. Med. Chem. 2020, 63 (13), 6679–6693. https://doi.org/10.1021/acs.jmedchem.9b02052.
- 肖高铿(2021-01-23).从活性位点里发现新的蛋白-配体相互作用. 墨灵格的博客. Retrieved from : http://blog.molcalx.com.cn/2021/01/23/spark-docking.html
- 肖高铿(2020-03-22).用常规分子对接进行共价抑制剂的虚拟筛选. 墨灵格的博客. Retrieved from :http://blog.molcalx.com.cn/2020/03/22/covalent-inhibitor-docking-vs.html
- SPARK数据库.墨灵格的博客. Retrieved from: http://blog.molcalx.com.cn/2021/04/07/spark-database.html
- Ji Qi,Yu Chao, Wang Ce, Sun Hanzi. WO2021058018. Inhibitors of KRAS G12C
相关主题
肖高铿(2019-06-25).SPARK案例 | 拜尔发现通过干扰RAS-SOS1相互作用能够阻断RAS激活的SOS1抑制剂. 墨灵格的博客. Retrieved from http://blog.molcalx.com.cn/2019/06/25/spark-bayer-ras-sos1-inhibitor.html
获取算例文件、软件试用以及技术服务,请联系我们
下载本算例,重现计算过程与结果:MRTX849-scafflod-hopping.fsp
Cresset不仅提供行业领先的药物设计软件,而且提供专业的药物设计服务:Cresset药物发现服务