膜蛋白的自由能微扰计算——用Flare FEP精确地计算暴露于脂质与GPCR P2Y1界面上配体的结合亲合力
摘要:本案例研究展示了如何用Flare™ FEP1精确计算结合于P2Y1脂质和GPCR界面之间30个配体的结合亲和力。在本文的基准测试研究中,预测的亲和力与实验测量结果一致,与文献中发表的结果一致甚至更好。本文提出的工作流表明,在FEP计算之前仔细地系统准备可以改进计算结果,从而减少获得可靠和准确的预测结合亲和力所需的计算次数。
作者:Lauren Nelson, Jenny Brookes
单位:Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK.
前言
尽管炼金术相对结合自由能( Relative binding free-energy, RBFE)计算有所改进,且应用日益增长,但是RBFE方法在诸如GPCR之类的膜蛋白上的应用仍未得到充分研究与测试2。正如Yang等人3报道的那样,在826种已知的人类GPCR中,大约350种非嗅觉成员被人们认为具有成药性,其中165种为经过验证的药物靶标,并参与了广泛的生理功能。因此,我们的目标是改进这些体系的建模以用于FEP计算。在本案例研究中,我们将Flare FEP1应用于一个精心准备的大型GPCR体系(如图1所示,不包括水有35,895个原子),在R2和MUE方面取得了比Ross等人4用竞争对手软件的结果更好,并与Dickson等人2发表的结果一致。
在本文中,我们使用了Dickson等人2发表的数据,他们研究了GPCR P2Y1嘌呤能受体,一种由百时美施贵宝公司鉴定用于治疗血栓形成的A类7次跨膜GPCR5。与其他GPCR复合物一样,P2Y1提供了研究药物如何与同一GPCR蛋白的不同区域结合并产生相同反应的机会6。有趣的是,除了细胞外结合位点外,P2Y1还有一个跨膜暴露位点位于P2Y1受体的螺旋束外7。这个位于膜和蛋白质之间的配体结合位点(见图1)为精确模拟脂质相互作用提供了有趣的挑战。
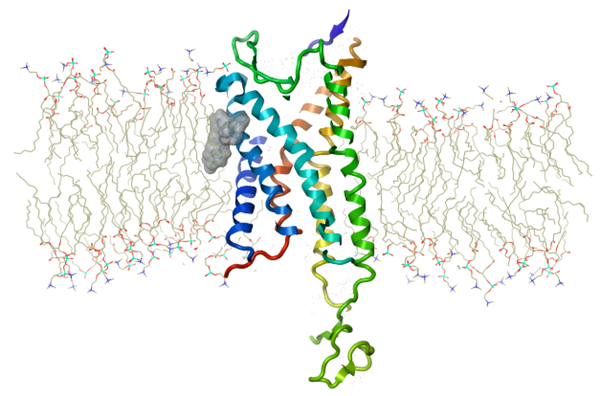
图1. P2Y1的晶体结构(PDB:4XNV)表示为以从N末端到C末端(从蓝色到红色)彩虹飘带,共晶体配体(BPTU)显示为嵌入到蛋白和POPC脂质双层之间的疏水分子表面。除了水之外,共有35,895个原子被建模,包括水盒在内共93,255个原子。这是从Flare进行20ns分子动力学(MD)模拟的输出中提取的一个快照图像。
为了重现Dickson等人2和Ross等人4的研究,我们使用Flare FEP预测30个已知活性同系物拮抗剂的亲和力,图2A显示了这些化合物的公共子结构。BPTU是P2Y1的有效拮抗剂,该系列就是从BPTU开始创建的,与前面提到的跨膜暴露区域结合。在FEP计算之前,使用MD模拟与3D-RISM水稳定性分析来仔细准备配体-蛋白复合物,比起Dickson等人2发表的亲和力预测,我们改善了预测结果,并增强了统计性能,从而可能减少后续更改的数量和完成先导优化工作所需的FEP运行次数。

图2.(A)P2Y1拮抗剂公共子结构;(B)在P2Y1共晶结构PDB 4XNY中的配体BPTU,作为其余29个配体的参比分子在Flare进行叠合。
模拟GPCR-配体复合物
BPTU(图2B)是一个有效的P2Y1 GPCR拮抗剂(Ki = 16 nM ,ΔGbinding = -11.2 kcal/mol)。P2Y1-BPTU复合物的结晶学数据可从PDB 4XNV获得,在Dickson的研究中也用了同一个结构数据。在他们的研究中,使用了一组包含30个拮抗剂的数据集,之前Chao等人5报道过这些化合物与P2Y!受体的实验Ki值。这些化合物的活性跨度为6.0-2000nM,活性数据范围覆盖了推荐的5 kcal/mol的动态范围8。
MD研究与3D-RISM水分析
如之前的Flare FEP案例研究,我们用MD与3D-RISM水分析来准备P2Y1体系。在进行更加复杂的FEP计算之前进行初始的MD与水分子分析步骤从多个方面讲都非常有益,最为重要的一点是获得可靠的建模能力以便抓捕生物活性的配体-蛋白结合模式。在本文的GPCR算例中,配体结合在蛋白/脂质界面上,尤其需要注意在分子动力学模拟过程中这些相互作用的保守性。此外,人们通常认为膜蛋白很干燥,因此对其放置水尤其具有挑战性。然而,某些关键的水分子可能存在于分子体系中,错误放置或没有出现的水分子可导致问题或错误。
鉴于体系的尺寸(总共93255个原子),我们进行了一个MD研究,包含一个POPC脂质双层包住P2Y1受体。在分子动力学模拟研究中,小分子采用最新的力场OpenFF 2.08-10,蛋白采用AMBER ff14SB力场11,采用POPC磷脂膜双层来模拟细胞膜。计算在298K,NPT系综、4fs的时间步长下进行。为了建立体系的稳定性,对轨迹进行了分析,检查了配体的RMSD,取后面的几帧快照进行结构准备并进行3D-RISM分析。对分子动力学模拟的轨迹分析表明,配体的坐标并没有偏离共晶参比分子很多,预期的相互作用得以保留,配体没有偏离结合位点。由于活性位点自身特性的原因(比如跨膜暴露区),需要特别小心,在分子动力学模拟期间,在磷脂双层与小分子之间是否有碰撞或不太可能的相互作用发生。
配体位于跨膜暴露区,用Flare实现的3D-RISM水分子分析方法(采用Cresset XED力场12)对选定的代表性MD快照进行分析以识别在这个显著疏水、封闭的结合口袋中的任何可能的水(图3)。以这种方法,3D-RISM可以用于识别水的位置、结合位点中正确的水合位点、甚至是晶体结构数据中所不能发现的水。这个MD快照,随着配体叠合到参比分子上(见下一节的配体叠合与微调)以及水分子的添加,然后可作为后续Flare FEP计算的输入。
在MD准备阶段,当磷脂膜生成并加到GPCR上的时候,包埋于磷脂-配体-蛋白区内的水分子可能缺失或者错误放置。将3D-RISM分析作为FEP分析体系的一部分使得这样的水分子的放置成为可能。因为3D-RISM分析快速且精确,用一个CPU在中等尺寸的非膜蛋白上仅需几分钟即可完成,对于非常大的膜蛋白体系可能需要更长的时间,但依然仅是FEP机时的一小部分。这是极其有用的准备步骤,可以避免非常耗机时的FEP计算的精确性与稳定性问题。
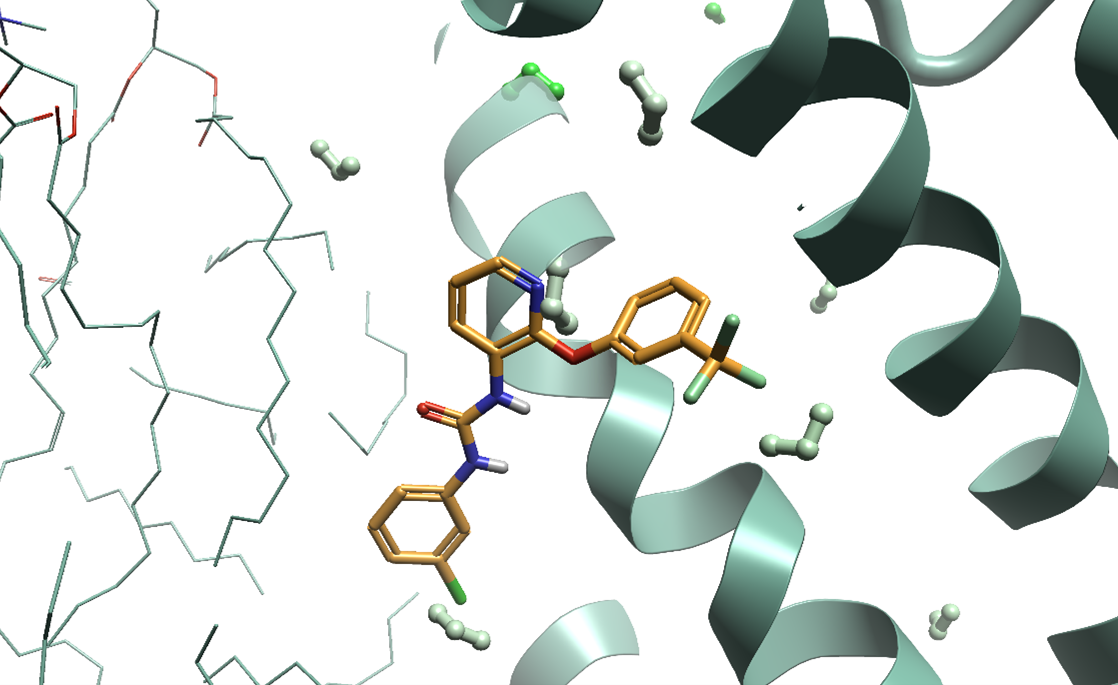
图3. Flare的3D-RISM可用于识别没被共晶数据鉴定出来的结合型水分子,为Flare FEP模拟提供有价值的输入。从红色到绿色的预测水分别对映于ΔG有利、不利的水分子。
考虑到膜蛋白原子输入的不确定性,这个初始准备工作流(比如用分子动力学模拟与3D-RISM分析)可能特别至关重要,用来确保模型包含所有必要的细节以便精确地表征体内体系。
配体叠合
在Flare中使用Cresset基于配体的叠合算法和子结构计算方法将含有30个配体的数据集叠合到选定的MD快照的配体构象上。配体叠合基于它们的最大公共子结构与配体场来给每个配体生成结合模式。检查手性、旋转异构体以及互变异构体状态,生成的紧密叠合最后用于创建理想的FEP计算起点,如图4所示。

图4. 在Flare中准备、叠合好的30个P2Y1配体数据集
Flare FEP计算
相对FEP计算特别适合于精确地计算小分子同系物的ΔGbinding,在本案例研究中展示的分子与共晶参比分子相比结构变化较小,因此FEP计算更加可靠。正如MD计算一样,所有的Flare FEP计算采用Open FF 2.0小分子力场9-10,蛋白AMBER ff14SB蛋白力场11,以及POPC磷脂膜双分子层。
第一个Flare FEP基准测试集总共有31个配体,包含30个已知活性的化合物和一个自动生成的中间体,如图5所示的多连接、闭环的标准微扰图。Flare FEP的中间体自动化是改进微扰网络一个重要特性,最终积极地影响了计算结果,而无需用户干预。为了检查延滞情况,创建了一个90个双向微扰图,基于假设每个微扰使用9个Lambda窗口,总共有810个Lambda窗口。从实践上看,Flare FEP实现了一个自适应Lambda采样以便在FEP计算之前以全自动的方式决定需要多少Lambda窗口实现收敛。双向微扰计算有利于好地误差分析并增强总体精度。
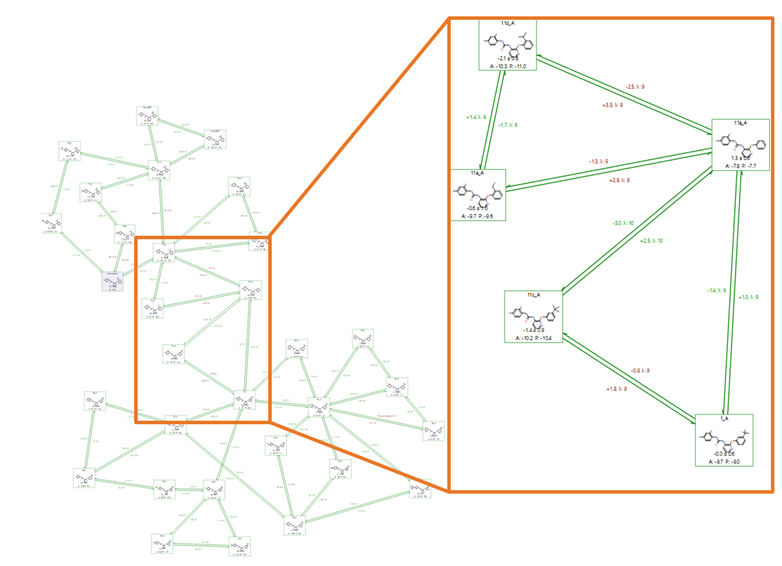
图5. 一个完整的、开箱即用Flare FEP基准测试,使用标准模式的微扰图。桔黄色的方框凸显了连接图上、下区的链接。
在本研究案例中,我们还应用了一个Flare最近引入的特性,即FEP聚类分析工具,图6蓝色方框高亮显示的配体就是用这个工具识别的。这个新特性应用子图分析从完整的基准测试研究里的活性图数据中聚类出内部误差统计更低的任意子图。如果在FEP预测中至少有2组不同预测精度的分子,或者这些分子通过有问题的链接连接时,此时对全部分子的评估会给你带来很大的误差高估,那么此时FEP聚类分析将非常有用。聚类可以让用户聚焦于预测可靠的子集合,在这些子集中,作为更小子集的一部分,预测可能更可靠,具有更高的内部预测精度。
令人感兴趣且易于理解的是,这个配体数据集可分为结构多样的两类。图6蓝色显示的上面那个类(top cluster)以苯环的邻位取代 (R1, 图2A)来区别于其它,而下面那个类由间位取代组成(R2, 图2A)。在这两个类之间通过一个迟滞的链接连接起来。观察这一点,我们的目的是通过手动添加中间体来提高FEP计算中上、下两个类之间的链接,从而改进了最初的开箱即用效果。图6高亮显示了两个手动加入的中间体分子,具有间位甲基的11c_EDIT以及邻、间位甲基的11e_EDIT。第二张微扰图由33个配体的98个微扰组成,总共882个lambda窗口。手动增加两个中间体的目的是增强图中上、下两个类之间的链接,从而减少微扰图对延滞、不精确链接的依赖。
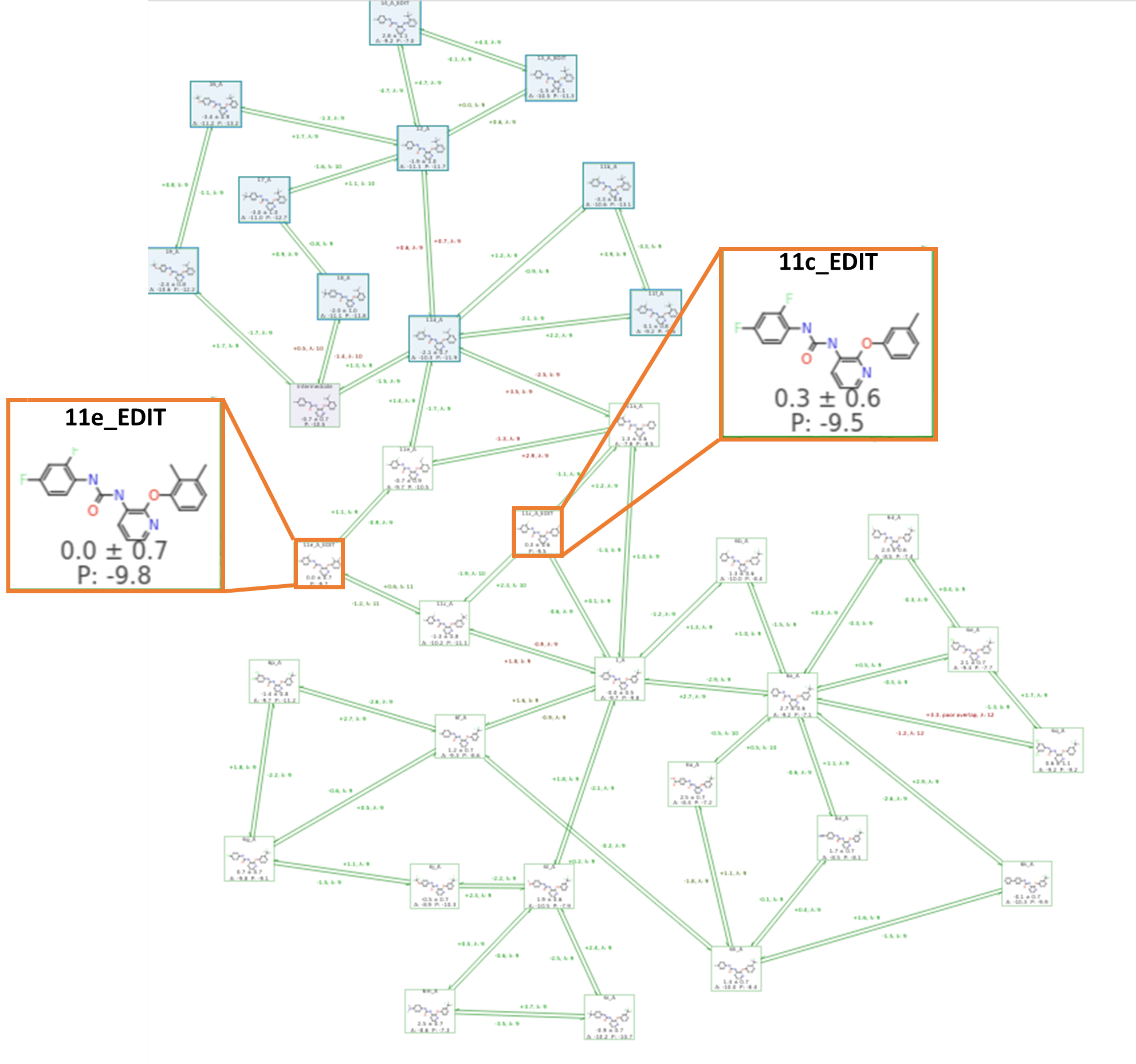
图6. 一个完整的Flare FEP基准测试,使用了带有手动添加中间体(11c_EDIT和11e_EDIT,用橙色框突出显示)的“标准”连接微扰图,以改善上、下两个类之间的链接。上面那个类中蓝色高亮显示是采用新的Flare FEP聚类工具实现的。
结果
总体而言,Flare FEP计算得到良好的结果,与Dickson等人2获得的结果一致,略优于Ross等人4使用竞争对手软件发布的结果(见表1)。当使用Flare FEP(无论是否包括手动加入的中间体,在没有如Ross等人4所建议的那样将配体划分为两个子图)时,完整的P2Y1数据集的R2为0.57,MUE为1.17,RMSD为1.65。
表1. 在P2Y1数据集上,Flare FEP统计结果与已发布数据的比较。RMSDpw*是数据集中所有配体对(成对)的ΔΔG预测值和实验值之间的均方根偏差。
| 方法\指标 | R2 | MUE | Tau | RMSDpw* | 配体数 | 微扰数 |
|---|---|---|---|---|---|---|
| Ross et al.4 | 0.48 | – | – | 1.41 | 30 | 76 |
| Dickson et al.2 | 0.58 | 1.29 | – | – | 30 | ~70 |
| Flare FEP (开箱即用) | 0.57 | 1.17 | 0.56 | 1.65 | 31 | 90 |
| Flare FEP(手动添加中间体) | 0.57 | 1.18 | 0.56 | 1.67 | 33 | 98 |
开箱即用:默认参数,自动生成中间体。
手动添加中间体(11c_EDIT和11e_EDIT)可以更好地连接邻位和间位取代的子集,并没有改善统计结果。作为一种替代方法,如Ross等人4所述,可以将初始微扰图分为两个单独的图(包含邻位和间位取代基)分别运行,以实现对每组化合物更好的统计。
结论
本案例研究表明,Flare FEP可以准确预测与膜蛋白结合的配体的结合亲和力,即使在具有潜在的困难情况下,如变构、脂质暴露的结合实例P2Y1。总的来说,通过应用由初步MD模拟、3D-RISM水分析和基于配体的叠合组成的仔细的体系准备工作流,Flare的开箱即用FEP计算结果与之前对P2Y1数据集2,4的研究报告结果一致,甚至略好,而无需对FEP计算进行详尽且计算成本高昂的微调。
虽然本案例研究侧重于基准测试模式,但这些结果可用于为生产运行模式提供信息。这个完整的30个配体数据集的良好统计数据表明,我们的模型和方法得到了很好的校准,并按预期工作,因此可以合理地预期,当用于预测新设计化合物的亲和力时,这种准备好的体系将在生产阶段继续提供良好的结果。此外,最终确定了两个分子“类别”的事实,使我们能够在生产模式运行中采取更有针对性和有效的设计策略,专注于性能更好的设计。
文献
- Flare™, version 7, Cresset®, Litlington, Cambridgeshire, UK; http://www.cresset-group.com/flare/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation J. Chem. Inf. Model. 2006, 46 (2), 665-676; Bauer M. R., Mackey M. D.; Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes J. Med. Chem. 2019, 62, 6, 3036-3050; Maximilian Kuhn, Stuart Firth-Clark, Paolo Tosco, Antonia S. J. S. Mey, Mark Mackey and Julien Michel Assessment of Binding Affinity via Alchemical Free-Energy Calculations J. Chem. Inf. Model. 2020, 60, 6, 3120–3130
- Dickson, C., Hornak, V., and Duca, J. Relative Binding Free-Energy Calculations at Lipid-Exposed Sites: Deciphering Hot Spots, Journal of Chemical Information and Modeling. 2021 61 (12), 5923-5930 DOI: 10.1021/acs.jcim.1c01147
- Yang, D., et al. G protein-coupled receptors: structure- and function-based drug discovery. Signal Transduction and Targeted Therapy. 2021, 6, 7. DOI: 10.1038/s41392-020-00435-w
- Ross, G. et al. The maximal and current accuracy of rigorous protein-ligand binding free energy calculations. ChemRxiv. Cambridge: Cambridge Open Engage; 2022; This content is a preprint and has not been peer-reviewed.
- Chao, H.; et al. Discovery of 2-(Phenoxypyridine)-3-phenylureas as Small Molecule P2Y1 Antagonists. Journal of Medicinal Chemistry. 2013, 56, 1704−1714.
- Yuan. S; et al. The Molecular Mechanism of P2Y1 Receptor Activation. Angewandte Chemie (International Ed. In English). 2016, 55(35), 10331-10335. DOI: 10.1002/anie.201605147.
- Mañé. N, Jiménez-Sábado. V, Jiménez. M. BPTU, an allosteric antagonist of P2Y1 receptor, blocks nerve mediated inhibitory neuromuscular responses in the gastrointestinal tract of rodents. Neuropharmacology. 2016, 110, 376-385. DOI: 10.1016/j.neuropharm.2016.07.033.
- Hahn, D. et al.; Best Practices for Constructing, Preparing, and Evaluating Protein-Ligand Binding Affinity Benchmarks [Article v1.0]. Living Journal of Computational Molecular Science. 2022, 4 (1), 1497. DOI: 10.33011/livecoms.4.1.1497
- Qui Y. et al. Development and Benchmarking of Open Force Field v1.0.0-the Parsley Small-Molecule Force Field. Journal of Chemical Theory and Computation. 2021, 17 (10), 6262-6280. DOI: 10.1021/acs.jctc.1c00571
- Wagner J. et al. openforcefield/openff-forcefields: Version 2.0.0 “Sage” (2.0.0). 2021. DOI: 10.5281/zenodo.5214478
- Maier, J. et al. ff14SB: Improving the Accuracy of Protein Side Chain and Backbone Parameters from ff99SB. Journal of Chemical Theory and Computation. 2015, 11, 8, 3696-3713 DOI: 10.1021/acs.jctc.5b00255
- Vinter, J. Computer-Aided Molecular Design, 1994, 8, 653-668
获取试用、商务合作,请联系我们
立即申请免费的Flare试用版,开始用Flare FEP来分析您自己的项目!此外,我们还提供FTE技术服务,由我们的专家帮您完成计算任务。



