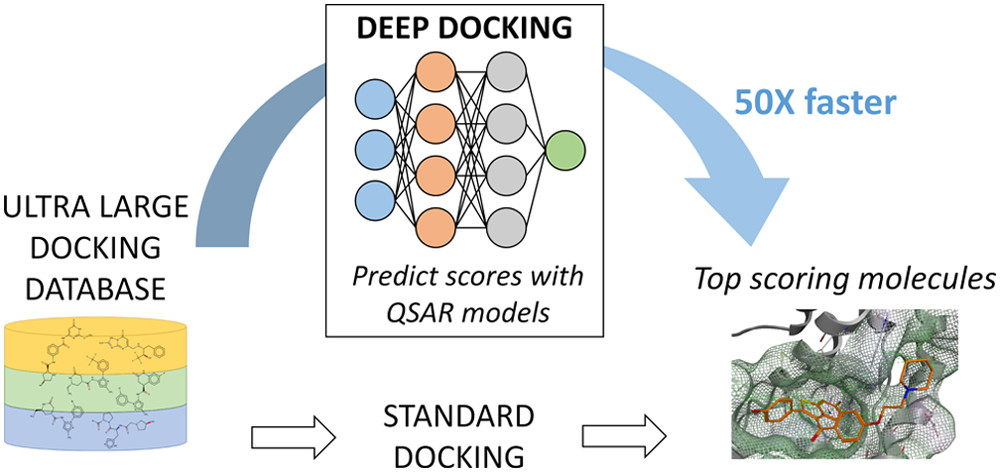
摘要:药物发现是一个严谨的过程,需要数十亿美元的投资和数十年的研究才能将分子从实验室带到病床边。虽然虚拟对接可以显著加速药物发现过程,但它最终落后于当前化学数据库的扩展速度,这些数据库已经超过了数十亿个化合物。最近可供购买的小分子数的激增带来了巨大的药物发现机会,但也要求更快的筛选方案。为了应对这一挑战,我们在此引入了Deep Docking(DD),这是一种新的深度学习平台,适用于以快速、准确的方式对接数十亿个化合物结构。该方法利用定量构效关系(QSAR)深度模型,在对接子集上训练化学库中的对接打分值,以近似未处理化合物实体的对接结果,并因此以迭代方式去除结合不利的分子。使用DD方法与FRED对接程序相结合,使得对ZINC15库中的13.6亿个分子针对12种主要靶标蛋白进行快速而准确的对接打分计算成为可能,并证明可减少高达100倍的数据量并富集高分分子6000倍而无明显有利对接分子的损失。DD协议可以很容易地与其他任何对接程序结合使用,并已公开发布。
原文:Gentile, F. et al. (2020) “Deep Docking: A Deep Learning Platform for Augmentation of Structure Based Drug Discovery,” ACS Central Science, 6(6), pp. 939–949. Available at: https://doi.org/10.1021/acscentsci.0c00229.
前言
药物发现是一个昂贵且耗时的过程,面临诸多挑战,包括高通量筛选的低命中率等1,2。计算机辅助药物发现(CADD)的方法可以显著加快这种筛选的速度,并能大幅提高命中率3。分子对接通常用于处理虚拟库,该库包含数百万种分子结构,针对具有已知三维结构的各种药物靶点进行处理。
随着自动化合成和可供购买化学品的激增,这为虚拟筛选(virtual screening,VS)方法,特别是对接方法提供了巨大的机会,但也带来了全新的挑战。例如,广泛使用的ZINC数据库从2005年4的70万条记录增长到2019年5的13亿多分子,这是惊人的1000倍增长。目前全球范围内仍然缺乏对这些数据库进行筛选的经验,并且对这些大型库进行对接比之对较小的库进行对接的优势仍存在争议6。然而,很少有近期发表的作品提倡扩大VS以涵盖超大型化学库。在Lyu等人7的一项开创性研究中,作者报告了针对1.7亿个按需制造分子结构的对接,显示这种数据库的VS能够发现高活性抑制剂以及常规筛选库存库中不存在的新化学类型。后来,其他9,10涉及大量分子的对接研究也得出了类似的结论。
鉴于当前对接程序的状态和CADD科学家可用的计算资源,现代对接计算很少超过1亿个分子,并且目前的化学空间对基于结构的药物发现仍然基本不可访问。解决这种差异的一种常见方法是使用预先计算的物理化学参数和类药的标准(如分子量、体积、辛醇-水分配系数、极性表面积、旋转键数量等)来筛选大型化学库为可管理的类药、类先导化合物、类片段和类苗头的子集(以及其他)11。虽然这种方法可以有效地将超大的对接数据库减少为可管理的子集,但许多潜在有用的化合物和新型或非传统的化学类型,尤其是从这些大型集合中出现的,可能会丢失6。为了充分利用可用和新兴的“按需制造”的化学品,必须最大限度地增加针对靶标的实际评估数据条目数。同样重要的是要注意,常规的对接工作流程明显忽视了负面结果。典型的对接活动依赖于完成完整的对接运行并选择一个非常狭窄的有利对接分子(虚拟苗头化合物)用作下一步评估的子集。因此,绝大多数对接数据(无论是有利还是不利的)都没有以任何方式或形式被利用,而它们可能代表了一种非常相关、格式良好且内容丰富适于机器学习算法的输入。
之前,我们团队和其他人12对通过浅层定量结构-活性关系(QSAR)模型预测对接打分值的可能性进行了探索,这些方法包括使用支持向量机或随机森林以及符合预测器的3D-“归纳”描述符13,14。然而,在这些方法中没有一种能够提供足够的速度提升来处理数十亿个分子,并且这种研究最多仅限于数百万个化合物。另一方面,深度学习(DL,Deep learning)特别适合大型数据集处理15,并且由于其相对于传统机器学习技术的优越性能,该方法在药物发现领域迅速引起关注16-18。因此,我们预计使用DL可以释放对接和QSAR方法之间的全部潜力和真正协同效应,并将充分利用超大规模对接数据库的数据。
结果
在本研究中,我们引入了快速计算和靶标无关的QSAR描述符(如2D分子指纹)的应用、对接数据库迭代和快速随机采样的应用以及主要使用DL来预测每次迭代步骤中未处理数据库条目的对接分数。因此,DD实现了超大对接数据库减少100倍,并且对排名靠前的苗头化合物富集超过6000倍,同时避免有利虚拟苗头的显著损失,如下文所述。
DD pipeline
本质上,DD管道(图1)依赖于以下连续步骤:
- (a)对超大型对接数据库(如ZINC15)的每个条目计算一组标准的基于配体的QSAR描述符(如分子指纹);
- (b)随机从数据库中采集一个合理大小的训练子集,并使用传统的对接协议将其对接到靶标上;
- (c)然后通过DL模型将生成的训练化合物的对接分数与它们的二维分子描述符相关联;然后使用对接分数截止值(通常是负数)来划分虚拟苗头化合物(得分低于截止值)和非苗头化合物(得分高于截止值)的训练化合物;
- (d)结果得到的QSAR深度模型(在经验对接分数上进行训练),用于预测尚未处理的数据库条目的对接结果。然后随机抽样预定义数量的预测虚拟苗头化合物并用于训练集扩充;
- (e)重复迭代步骤b-d直到达到预定义的迭代次数,和/或超大型对接数据库中的已处理条目收敛。
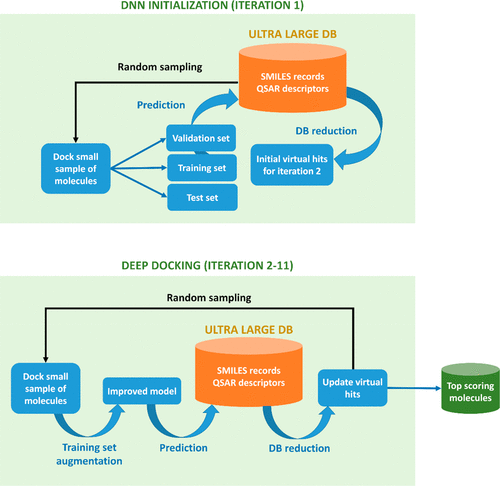
图1. DD管道的示意图。(上)DD初始化:从超大型对接数据库中随机提取一小部分分子,并将其与靶标进行对接。然后,生成的对接分数用于训练QSAR深度模型。创建的QSAR解决方案随后用来预测数据库剩余部分的对接结果,并返回所需的虚拟苗头化合物以开始第2轮迭代。 (下)DD筛选:从第2次迭代开始,通过用前一次DD迭代中随机采样的QSAR预测虚拟苗头化合物来逐步改进深度模型(这些虚拟苗头化合物也被选作实际对接)。该循环重复预定义次数的迭代后,DD会返回数据库中的高分分子。这个最终库可以经过处理以去除残留低得分实体。或者,步骤2-11可以一直执行到一个超大型对接数据库收敛为止。
在DD中,虚拟命中召回率(Hit recall,即从数据库检索到的实际虚拟苗头化合物的百分比)是通过一个概率阈值隐式设置的,该阈值被选择为包括验证集中的90%实际虚拟苗头化合物。然后,相同的阈值应用于独立测试集,并评估虚拟命中召回率以评估模型的泛化能力。如果验证和测试集的召回率一致,则将模型应用到数据库的所有条目(更多详细信息可以在方法中找到)。虽然可以通过使用例如符合预测器来显式地支持召回值14,19,但是我们没有观察到DD结果性能有显著差异。
执行DD管道的脚本,以及有关如何设置运行和额外工具以促进HPC集群自动化的说明,可在GitHub上公开获取:https://github.com/vibudh2209/D2。
超大对接数据库的采样
选择一个代表性和平衡的训练集是任何建模工作流的关键步骤。在采样化学空间的情况下,适当的DD训练集应该有效地反映数据库的化学多样性。可以预期的是,扩大采样的规模和预先对对接数据库聚类最终会改善甚至收敛化学空间覆盖范围。另一方面,目前以任何方式对数十亿个化学结构进行聚类都是不可行的,并且已经表明,在对接之前对大型库预先聚类可能会显著降低活性化学类型的排名,从而阻碍新型抑制剂或激动剂的发现7。此外,偏向于那些由DD高度排名为潜在虚拟苗头分子的采样可能将排名低但真阳性分子排除在用于模型训练的分子之外;因此在所有的DD迭代中我们选择了随机采样。最后,DD训练集的大小(例如实际对接的数量)会对计算运行时间产生关键影响,并应仔细控制。
为了建立ZINC15数据库的最佳采样,我们评估了DD训练集大小与测试集中召回率的均值和标准差之间的关系,这反映了模型性能的一致性和泛化能力。为此,我们对四个主要药物靶标家族中的12个蛋白质靶标进行了评估,包括AR、ERα和PPARγ等核受体;CAMKK2、CDK6和VEGFR2等激酶;ADORA2A、TBXA2R和AT1R等GPCR;Nav1.7、GLIC和GABAA等离子通道,更多关于所选靶标的详细信息见表S1。对于全部12个研究的靶标,我们调查了样本大小与结果测试集召回率之间的关系,在训练集大小在250,000到1百万分子之间时,这些值似乎收敛至0.90(图2a)。我们也观察到当样本量约为1百万时,标准偏差收敛于0(图2b)。因此,我们将1百万分子设定为DD工作流程的标准采样(更多信息可以在方法中找到)。
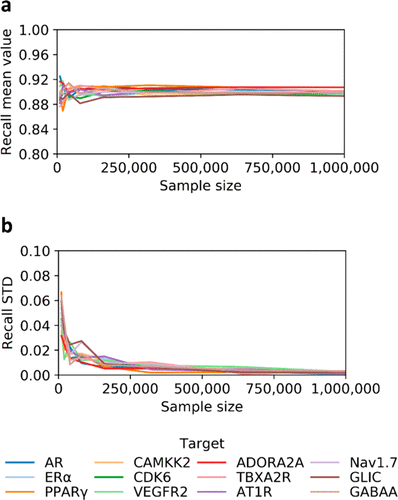
图2. 训练集样本大小对模型泛化能力的影响。(a)使用不同样本大小计算的测试集召回率的平均值。当训练集规模在25万至1百万分子之间时,所有靶标的值接近0.90。(b)标准偏差(STD)的变化趋近于零,对于1百万个分子的样本量而言。我们为每个靶标运行了一次迭代,并在每次采样大小下重复了五次计算。
DD虚拟筛选降低了ZINC15的化合物数量
DD方法的主要目标是将数十亿条记录的超大型对接数据库减少到可管理的几百万分子子集,但该子集仍涵盖了绝大多数虚拟苗头化合物。然后可以使用一个或多个对接程序正常地将最终的分子子集对接到靶点上,或者用其他VS手段进行后处理。DD方法依赖于通过扩展其训练集来迭代改进深度神经网络(DNN)的训练,其中预测的苗头分子来自每个先前的迭代,而决定性的截止值也逐渐变得更加严格。我们广泛评估了这种DD协议的表现,使用对接程序FRED21,通过筛选ZINC15中全部的13.6亿个分子对上述12种蛋白质靶标进行了筛选。值得注意的是,DD本身并不是一种对接引擎,而是一种DL打分预测器,用于与任意对接程序结合使用,以快速消除先验不利的“无法对接”的分子实体,并因此大大提高了实际对接的速度。
为了展示DD的力量,我们用一组固定的参数(如迭代次数、召回值等)测试了DD管道,以便用这12个研究体系提供客观的比较。预计DD用户可能希望使用不同于我们的不同模拟参数,这些参数最适合他们的时间和资源分配:例如,在具有许多CPU和少量GPU的计算集群中,较少的迭代次数与每次迭代更多的对接以及更少的DL周期可能是最佳选择,反之亦然。
对于每个靶标,我们总共运行了11次DD迭代——一次初始训练步骤(需要对接3百万条记录以构建1百万个训练、验证和测试集)以及10次连续的迭代对接步骤,每次对接1百万分子。因此,对于每个靶标,实际上只对接了代表ZINC15中13亿条记录不到1%的1300万个分子结构。图3a展示了用于区分每轮中的苗头和非苗头对接打分的阈值。阈值在每一轮中变得更为严格(详见方法),这些值根据定义虚拟苗头化合物的标准而减少。所有靶标的大部分非苗头化合物在第一轮中被除去,而在后续步骤中丢弃较少的分子,这符合预期,因为在开始时存在较多不利化合物。我们观察到,下降率和识别出的苗头化合物数量取决于靶标(图3b)。从DD进展分析得出的另一个值得注意的结论是,训练集在每一轮迭代后都有效改善,因为添加到训练中的分子的对接打分在每一次建模循环后变得更负(更有利)(图3c)。这一观察标志着DD在识别和排除低分分子方面表现得越来越有信心,并且相应地增强了有利的训练集。因此,我们预计DD很可能在每一轮迭代后提高虚拟苗头化合物物的富集度,如图3d所示,该图显示了在测试集中由DNN模型排名前100位的分子的富集值。正如数据所表明的那样,在所有靶标上这些值在每一轮迭代后都会增加,这也表明每当用来自之前每一轮DD的分子来扩充训练集时,模型的表现也会有所提升。值得注意的是,在最后一轮迭代中,模型检索到了数据库中得分最高的分子的一小部分(0.01%),其富集值显著增加。进一步证明DNN模型迭代改进的证据是由曲线接收器工作特征下的面积(AUC ROC)值和完整的预测数据库富集(full predicted database enrichment,FPDE)值提供的,这些值在表S3中为研究的所有12个靶标提供了所有迭代的结果,以及模型识别高分分子的精度,也像预期一样在每一轮中得到改善(图S2)。
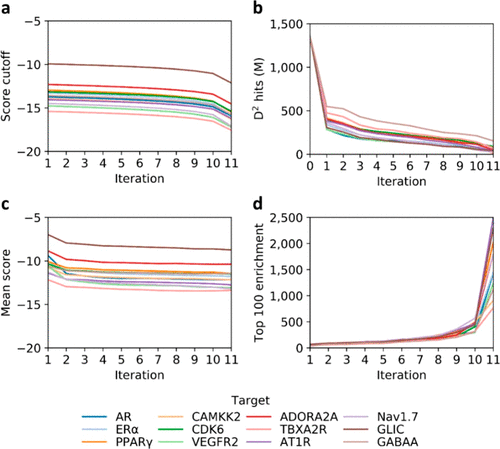
图3. 针对12种药物靶点的DD性能统计。(a)用于选择每个迭代中虚拟苗头化合物打分截止值的变化。(b)在每次迭代后预测为虚拟苗头分子的数量变化。 (c)训练集扩增所用随机选取分子对接打分均值的迭代改进。(d)计算每迭代一次测试集中前100个排名最高的预测虚拟苗头化合物的富集值。
分析DD的性能
如前所述,DD方法的主要目标是将超大型对接数据库减少到一个高度富集的分子库中,这些分子可以使用传统的对接程序和计算资源进行处理。在研究12个精选的靶标时,我们观察到最终剩余子集的大小范围为原始超大型对接数据库的1%至12%(图4a)。预计这些剩余下来富集且DNN排序后的库然后可以通过后处理来去除残留打分低的分子。或者,DD可以在超大型对接数据库收敛之前完成。
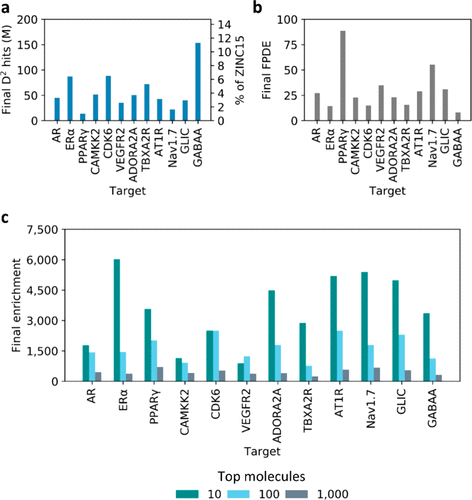
图4. DD运行后最终数据集的大小和富集值。(a)第11次DD迭代后预测为虚拟苗头化合物的分子总数。值以ZINC15条目保留百分比的形式报告(右侧纵轴)。 (b)来自DD实验最后几次迭代的FPDE值。(c)前10,前100和前1000个选定虚拟苗头化合物(在测试集中)的富集值。
DD在PPARγ蛋白中表现出最佳性能,数据库被缩小到其大小的1%。因此,在考虑对接需要训练模型以及对最终子集进行后处理的情况下,针对该靶点的ZINC15的DD筛选只需要对接比传统VS少50倍的分子。另一方面,DD在GABAA靶点上效果最差,由于模型精度低,在最后一次迭代后仍有12%的ZINC15分子未被识别。这些结果清楚地表明,与任何其他计算工具一样,DD的表现取决于靶点。令人鼓舞的是,在全部情况下,召回值都始终转移到测试集中(见表S3)。我们还比较了基于测试集的每个迭代预期返回的分子数量和当将DD模型应用于ZINC15时实际返回的数量,并观察到没有显著差异(图S3)。综合来看,这些结果表明所有底层DL模型以一致的方式泛化。为了进一步评估总体DD性能,我们评估了最终FPDE值,范围从8到89,这表明DD以靶标依赖的方式富集了高对接打分的分子的最终子集。正如所料,FPDE值符合预测虚拟苗头化合物数的趋势(图4b)。
我们还评估了在最终迭代后,在测试集中的前10,100和1000个预测虚拟苗头化合物中识别的富集值,如图4c所示,观察到的值范围从240到6000。当评估排名较高的结构时,所有情况下这种富集都持续下降,这表明真正的苗头化合物高度集中在DD排名高的顶部,并且排名底部的分子大多是假阳性。重要的是要注意,我们在DD运行中设置了严格的0.90召回率值,以保留最后子集中绝大多数的虚拟苗头化合物。然而,可以降低召回率阈值来牺牲DD工作流中虚拟苗头化合物的保留,但要显著减少最终保留未处理分子的数量,并缩短运行时间。
为了调查降低虚拟命中召回率的效果,我们对两个体系进行了DD计算,即PPARγ和GABAA(在上述11次DD迭代后仍有大量分子未处理的最佳和最差靶标),使用了0.75和0.60的召回值。令人鼓舞的是,在两种情况下,与0.90召回的DD计算相比,剩余条目的数量显著减少(图5a)。对于PPARγ系统,当召回率为0.75时,ZINC15的规模缩小了800倍,而当召回率为0.60时,则缩小了2300倍。同样地,我们在GABAA靶点上观察到,分别以0.75和0.60的召回率进行操作时,其规模减少了66倍和654倍。重要的是,当我们使用较低的召回率时,分子子集的富集明显增加,FPDE值达到1450,再次清楚地表明随着远离DNN排名顶部,虚拟苗头化合物密度迅速下降(图5b)。因此,可以根据用户所需的加速速度或可用的计算资源来选择召回值,以便进一步减少DD运行结束时需要的对接和/或后期处理的量。
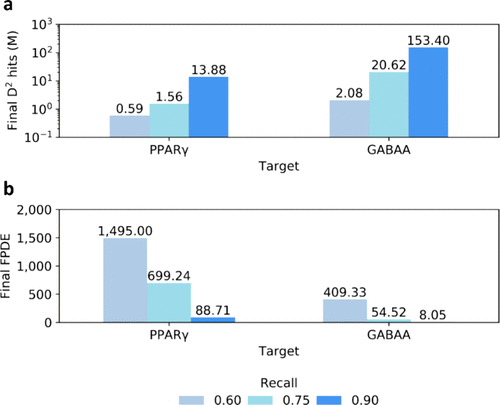
图5. DD样本量减小的能力取决于召回值。(a)由DD预测的虚拟苗头化合物数量(以样本量的对数值表示),以及(b)通过改变虚拟命中召回率获得的PPARγ和GABAA体系的FPDE值。
综上所述,上述分析表明DD流程可以高效地丢弃超大对接数据库中大部分不合格的分子,而不会丢失超过预定义百分比的虚拟苗头化合物。我们认为这使得DD方法成为一种有效的手段来进行涉及数十亿小分子结构的大规模VS计算,并且是一种有效的方法来替代需要大量计算资源的蛮力方法。
DD虚拟筛选与活性配体
我们还研究了DD方法如何处理超大型对接数据库中存在的活性配体。对于五个被调查的体系,即AR、ERα、PPARγ、VEGFR2和ADORA2A,DUD-E22提供了确认的活性配体集(有关数据集的详细信息见表S2),我们将这些配体与来自ZINC15的一百万随机化合物(视为非活性分子)一起对接到各自的靶点上。FRED在不同靶标上的对接性能变化很大,AUC ROC值范围从0.52到0.91(图6a)。同时,我们评估了化合物的DD排名(以召回率为0.90进行评估)与其对接打分值的相关性,特别是它们距离用于定义虚拟苗头化合物和非苗头化合物分子的最后迭代中的打分阈值的距离,使用每个靶标中100万个ZINC15分子的随机样本进行了对接(图6b)。DD似乎明显偏向于高对接打分值的最终集合,并且由于基于AUC值,活性配体比非活性配体打分更高,因此我们期望活性配体会以低于非活性分子的速度被淘汰。因此,我们将活性配体的排名与其得分距虚拟苗头化合物阈值的距离绘制成图(图6c)。正如预期的那样,DD对具有许多高打分活性和良好AUC值的体系的效果特别好,例如AR和ERα,在这些体系中所有活性、最高打分的配体都被保留下来。有趣的是,DD为所有的靶标保留了活性配体的绝大部分以及非苗头化合物,大约是剩余ZINC15非苗头化合物命中率的10倍。
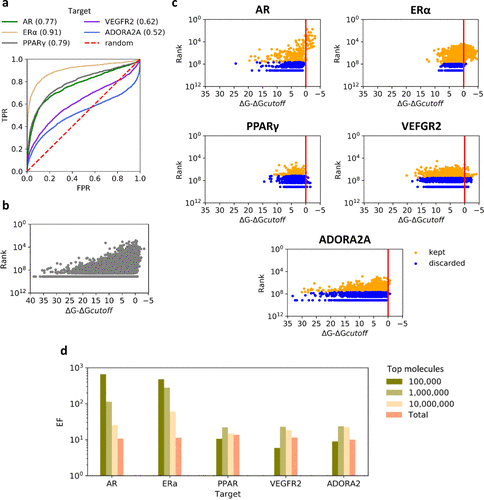
图6.(a)活性配体与五个靶点FRED对接的ROC曲线。活性化合物与一百万随机采样的ZINC15分子混合。每个靶标的AUC值在括号中报告。(b)DD排名(对数尺度)与距离分数从一百万个ZINC15化合物对接到五个靶点的随机样本的截止值的距离。(c)DD排名(对数尺度)与活性配体打分截止距离。保留(召回)的配体用黄色圆点表示,丢弃的配体用蓝色圆点表示。(d)当考虑前100000、1000000、10000000和所有DD苗头化合物时,活性配体的富集因子(EF,对数尺度)。
然后,我们对这五个体系计算了前10万、100万和1000万排名分子中活性化合物的富集因子(enrichment factors,EFs)。不出所料,最高值出现在AR和ERα上,其中前10万个分子的EFs令人鼓舞地高,分别为660和477,随着评估更大比例的打分最高的分子而持续下降(图6d)。对于其他三个体系,在考虑不同大小时,并未观察到明显的富集趋势。然而,所有考虑的分子子集都富集活性配体,显示出至少六倍的EFs。因此,当可用的计算资源有限时,选择一个DD分子排名靠前的子集似乎是降低召回值的有效替代方案,以限制最终对接的数量。此外,所有体系的所有DD苗头化合物的EFs都在11至14之间。因此,DD不仅能够从超大型对接数据库中提取受控部分的虚拟苗头化合物,而且还能使最后的子集富集活性苗头分子。值得注意的是,无论其分数如何,保留活性这一意外的DD特征也导致了为靶标富集近似随机对接性能,例如ADORA2A体系。
讨论
随着合成流程的自动化程度不断提高,现代药物发现活动的重点将转向对包含数十亿化学结构的日益庞大的分子库进行筛选。为了加强这种机会,对接协议在更大的按需数据库上表现出改进性能,有效地为药物发现努力提供了新颖的、结构多样的和非传统的化学类型7。然而,值得注意的是,在现代对接活动中投入的时间和资源大多用于处理不利的分子结构,而新兴的“负面”数据也未被以任何方式或形式利用。
因此,为了跟上不断扩大的化学大数据空间的步伐,并充分利用对接程序“飞速”产生的结果,我们开发了Deep Docking协议DD,这是一种基于深度神经网络的方法,用于处理大型化学库,同时使用传统的计算和软件资源。该方法依赖于迭代对接一个父代超大数据库(如ZINC15)的一小部分,使用选择的对接程序,并利用生成的打分值(有利和不利)来训练基于配体的QSAR模型。然后,该模型能够近似未处理数据库条目的对接结果。重要的是,DL允许使用简单的与蛋白质无关的2D描述符,例如Morgan指纹,以捕获对接打分值。我们已经证明这种策略可以产生一个可管理的小子集数据库,高度富集了“可对接”的分子结构。
我们通过将ZINC15中全部的13.6亿条记录与12种主要药物靶点进行筛选,证明了DD的威力。在检索到一定比例的有利对接分子的同时,原始超大对接数据库得以显著减少。同时,在不花费时间和资源的情况下,大多数打分低的分子都被移除,并且生成的ZINC子集富含潜在虚拟苗头化合物。值得注意的是,使用DD对一个超大的对接数据库进行筛选需要对接比传统对接少50倍的分子,而损失的虚拟苗头化合物仅占约10%。我们也展示了DD可以进一步缩小超大的对接数据库至几十万分子,但仍能检索出相当一部分(60%)的高分苗头化合物。此外,即使只考虑一小部分排名靠前的分子,DD似乎也能使最终子集富集活性配体。这一意外的结果表明,真正的结合剂携带某种化学特征,这些特征与结合口袋互补,模型能够通过QSAR描述符捕捉此类特征。
结论
在本研究中,我们以一种新颖的方式介绍了深度学习在基于结构的药物发现中的应用。开发的Deep Docking方法利用QSAR模型,在分子数据库的小子集的实际对接打分上进行训练,以预测其余部分的对接打分值。这种迭代使用的方法(具有预定义的召回参数)可以显著节省对接运行时间,而不会显著地损失潜在“可对接”的实体或活性配体。使用Deep Docking绕过了大规模对接活动的计算限制,并使数十亿条目分子数据库即使在有限的计算资源下也可访问。
总的来说,我们的研究结果强烈地支持使用深度学习来探索不断扩大的化学空间以寻找新的治疗药物。
方法
QSAR描述符
从ZINC155下载了13.6亿个分子结构的SMILES。使用RDKit软件包23生成大小为1024位、半径为2的Morgan指纹。
蛋白靶标
AR24,ERα25,PPARγ26,CAMKK227,CDK628,VEGFR229,ADORA2A30,TBXA2R31,AT1R32,Nav1.733,GLIC34和GABAA35等的X-衍射结构以及共结晶配体从PDB36中提取。有关所选靶标结构的详细信息总结在表S1中。
分子对接
PDB结构使用Schrödinger套件中的Protein Preparation Wizard模块进行优化37,并使用OpenEye的应用程序MakeReceptor准备对接网格38。使用QUACPAC8处理SMILES以生成pH为7.4时的优势同分异构体和离子化状态。使用OMEGA的pose模块39,40为FRED对接生成多个3D最优构象。对接模拟使用FRED程序和OpenEye的Chemgauss4打分函数进行21。
数据库采样
通过为每个靶标运行一个DD迭代,使用不同大小样本量的训练、验证和测试集(10,000,20,000,40,000,80,000,160,000,320,000,640,000和一百万个分子)进行训练,确定了用于训练集的最优分子数量。对于每个样本量,计算被重复五次独立地执行。然后通过评估所有靶标在测试集中召回值的平均值和标准偏差来选择最佳采样大小。
DD工作流
用于DL模型的初始训练、验证和测试集由在第一个DD迭代期间从ZINC15中随机采样的1百万个分子组成。将每个数据集对接到靶标上,然后根据生成的对接打分值将其分为虚拟苗头化合物和非苗头化合物。确定分子类别的打分阈值是为了将验证分成1%最高打分分子(虚拟苗头化合物)和99%非苗头化合物。每个集合中的对接分值等于或优于阈值的分子被分配到虚拟苗头化合物类别,而其余分子则分配到非苗头化合物类别。DNN模型使用处理后的条目类别和分子描述符进行训练,然后基于分子描述符来预测整个ZINC15中的虚拟苗头化合物和非苗头化合物。从第二个迭代开始,训练集扩展为1百万个分子,这些分子是从前一个迭代中预测的苗头化合物中随机抽样得到的,同时验证集和测试集在整个DD运行过程中保持不变。每次迭代后逐渐降低打分阈值(对应于更高的靶标结合亲和力预测值),以继续选择更好的化合物。这种减少是通过线性降低验证集中分配给虚拟苗头化合物类别的高分分子百分比来进行的,在第一轮迭代中为1%,在最后一轮迭代中为0.01%。为了防止在初始迭代时出现大的变化,从而限制模型全面探索化学种类,因此选择了线性的打分阈值的变化。因此,在第2次迭代中,阈值对应于排名前0.9%的化合物的最高(最差)对接分值,在第3次迭代中它对应于排名前0.8%的化合物的最高对接分值,依此类推。
性能评估指标
所有评估指标都是在测试集上计算的。精度是这样计算的:
$$
precision = {TP \over {TP + FP} }\cdots(1)
$$
其中,TP(真阳性)是DNN正确预测的虚拟苗头化合物;FP(假阳性)是实际非苗头化合物,但被DNN错误分类为虚拟苗头化合物。
召回率计算如下:
$$
recall = {TP \over {TP + FN} }\cdots(2)
$$
其中,FN(假阴性)是DNN错误丢弃的虚拟苗头化合物。
富集值计算为:
$$
top\ N\ enrichment = {{TP|_{top\ N}} \over {TP|_{random\ N}} }\cdots(3)
$$
其中,\(TP|_{top\ N}\)表示在DNN排名靠前的N分子中发现的TP数量,而\(TP|_{random\ N}\)表示在随机采样的N个分子中发现的TP数量。将N设置为等于10、100和1000的值。
FPDE是精确度(等式1)与随机精度之比:
$$
random\ precision = {{TP|_{database}} \over {total\ molecules|_{database}} }\cdots(4)
$$
深度学习
使用Keras Python库41构建和训练前馈DNN模型42。模型超参数被设置为隐藏层和神经元的数量,丢弃频率以及少数类的过采样和类别权重,以处理高度不平衡的数据集(1-0.01%的虚拟苗头化合物)。对于DNN概率(指示分子成为虚拟苗头化合物的可能性)建立了较低的阈值,并将其用作预测时将分子分配到虚拟苗头化合物类别的标准。每次选择阈值是为了检索验证集中90%的实际虚拟苗头化合物(即得分最高的分子)。 通过运行基本网格搜索(grid search)来确定在测试集中提供最高FPDE值的一组超参数来进行模型选择。然后用最佳模型对所有ZINC15条目进行预测,以预测虚拟苗头化合物和非苗头化合物。
Active Ligands
相应靶标的活性化合物是从DUD-E22中获得的。如果尚未出现在ZINC15中,将化合物的SMILES以及计算的相应Morgan指纹添加到其中。并根据之前描述的方式进行结构准备,并与各自的靶点对接。通过虚拟苗头化合物相似性DD概率对前N个分子进行排序后,富集因子(Enrichment factors)根据下式进行计算:
$$
EF = {{actives|_N} \over {actives|_{database} \left( {N \over {mol|_{database}}}\right) } }\cdots(5)
$$
其中,\(actives|_N\)是打分靠前N个分子中发现的活性配体的数量,\(actives|_{database}\)是活性配体总数,\(mol|_{database}\)是数据库大小。
硬件
我们使用了6个Intel(R) Xeon(R) Silver 4116 CPU @ 2.10 GHz(总共60个核心)进行对接,以及4个Nvidia Tesla V100 GPU和32 GB内存用于DNN模型的训练和推理。
支持信息
略
文献
略
推荐读物
- Yaacoub, J.C. et al. (2022) “DD-GUI: a graphical user interface for deep learning-accelerated virtual screening of large chemical libraries (Deep Docking),” Bioinformatics, 38(4). Available at: https://doi.org/10.1093/bioinformatics/btab771.
- The ZINC20 database in DD format has been deposited at https://files.docking.org/zinc20-ML.
- Gentile, F. et al. (2022) “Artificial intelligence–enabled virtual screening of ultra-large chemical libraries with deep docking,” Nature Protocols, 17(3), pp. 672–697. Available at: https://doi.org/10.1038/s41596-021-00659-2.
联系我们
想在自己的项目中亲自使用OEDocking/FRED进行Deep Docking计算,请联系我们获取免费的试用版;你还可以采购软件或委托研究与我们进行项目合作。
- 电邮:info@molcalx.com
- 电话:020-38261356
原创文章,作者:小墨,如若转载,请注明出处:《Deep Docking——一个增强的基于结构药物发现深度学习平台》http://blog.molcalx.com.cn/2025/01/30/deep-docking-a-deep-learning-platform.html



