摘要:本文以化合物31作为查询分子,对ChEMBL35数据库进行基于形状的虚拟筛选,然后对筛选结果进行了化学基因组分析,预测了化合物31的可能靶标,提供了一种基于3D形状相似性进行靶标预测的方法。
1 前言
近期,我们在一篇博客文章中探讨了查询结构(query)的准备对基于形状的虚拟筛选结果所产生的显著影响1。该研究以阿斯利康近期公开的ROS1抑制剂——化合物31(图1)为例,对ChEMBL35数据库进行了虚拟筛选。结果表明,采用生物活性构象构建的查询结构能够有效召回已知的ROS1抑制剂;而若使用非活性构象作为查询,则难以识别出具有真实生物活性的化合物。
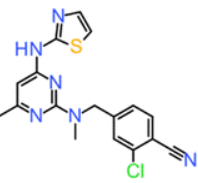
图1. 化合物31的化学结构式(ROS1 IC₅₀ = 97 nM)
鉴于本研究中所使用的ChEMBL数据库是一个成熟的化学基因组学资源,这一结果自然引出了一个关键问题:是否可以通过3D结构相似性推断潜在的靶标?换言之,若某化合物与已知靶标配体在三维形状与药效团特征上高度相似,其是否可能作用于相同或相近的靶点?基于此,本文旨在对上述虚拟筛选结果进行系统的化学基因组学解读,并简要回顾相关方法学基础。
2 方法学背景
我们在2017年的一篇技术博客中2曾详细介绍如何利用OpenEye公司开发的ROCS(Rapid Overlay of Chemical Structures)软件,基于分子三维形状与药效团特征,对PDB中共晶结构中的生物活性配体进行虚拟筛选,从而实现靶标预测(target fishing)。该方法已通过我们搭建的在线服务平台,为国内多家学术机构与制药企业完成了多项靶标识别与脱靶效应分析任务,部分成果已经发表文章3-5。
3 蛋白–配体识别的基本假设
蛋白–配体相互作用的核心物理基础在于三维互补性:即分子的形状与静电分布若能与蛋白结合口袋高度匹配,则该分子很可能成为该蛋白的有效配体。药效团可视为静电相互作用的一种简化模型。因此,具有相似三维形状与药效团特征的化合物,往往倾向于结合至相同的结合位点,并表现出相似的生物学活性。
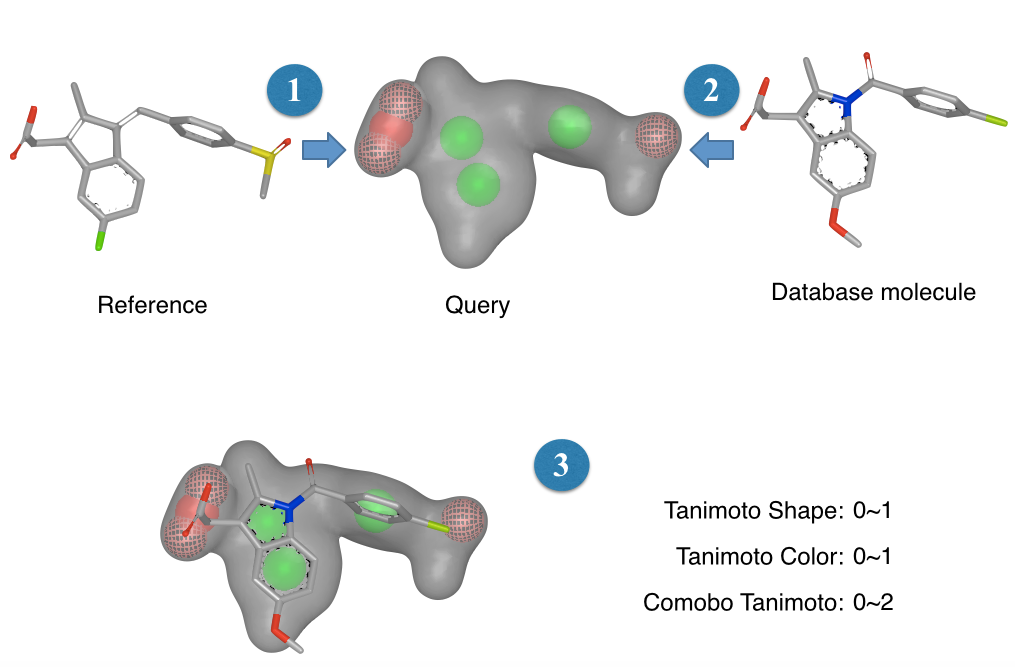
图2. 基于ROCS的靶标预测原理示意图
ROCS通过将待测分子转化为三维形状与颜色(即药效团)表示的查询式(query),随后将其与数据库中已知靶标的配体进行叠合比对,计算其相似性得分。主要输出指标包括:
- Tanimoto Shape(范围:0–1):衡量形状相似性;
- Tanimoto Color(范围:0–1):衡量药效团相似性;
- Combo Tanimoto(范围:0–2):为前两者之和,值越接近2,表示整体3D相似性越高。
在此框架下,与查询分子在3D空间高度相似的已知配体所对应的靶标,即为预测的潜在靶点。
4. 化合物31的靶标预测:基于ChEMBL35的化学基因组学分析
尽管前文1中并未直接使用ROCS,而是采用了其开源近似算法ROSHAMBO2,我们仍以化合物31为参比分子,对ChEMBL35数据库进行了大规模虚拟筛选,获得了Tanimoto Combo相似性得分最高的10,000个命中化合物。随后,我们提取这些化合物在ChEMBL中对应的靶标信息,并对靶标进行聚合分析:每个靶标以其最相似的配体为代表,该配体的相似性得分即作为该靶标的预测置信度。
在本文中,我们采用RefTverskyCombo作为主要打分标准进行结果展示。选择该指标的原因在于其在虚拟筛选任务中的优异表现6——在DUD-E基准数据集上,RefTverskyCombo的性能优于当前主流的商业软件、开源工具及多种AI驱动方法7。
需要特别指出的是,本次分析未对原始预测结果进行任何后处理优化,例如:
- 未排除构象不稳定的预测;
- 未过滤缺乏关键相互作用(如氢键、π–π堆积等)的匹配;
- 未考虑空间位阻或立体冲突等问题。
因此,以下结果应视为初步预测,需结合结构生物学与实验验证进一步评估。
5 结果与讨论
5.1 化学基因组分析概要
本次化学基因组学分析基于 ChEMBL 35 数据库,采用 RefTverskyCombo 相似性算法,对输入文件 CONF_34_VS_hits_score.csv 进行解析,最终生成 792 个靶标 的预测结果。核心数据统计如下:
- 相似性文件记录数:10,000 条
- 靶标文件记录数:6,461 条
- 构建化合物相似性映射:10,000 个
- 匹配靶标记录:6,461 条
- 输出报告位置:target_prediction_report.csv
5.2 关键统计指标
预测结果覆盖792个靶标,相似性得分呈现明显梯度分布(表1)。
| 指标 | 数值 |
|---|---|
| 总靶标数 | 792 |
| 最高相似性打分 | 1.968 |
| 最低相似性打分 | 1.230 |
| 平均相似性打分 | 1.590 |
5.3 打分靠前预测的靶标详细信息
靶标ID: CHEMBL2815
靶标名称: Nerve growth factor receptor Trk-A
物种: Homo sapiens
相似性打分: 1.968000
相似性区间: [1.560000, 1.968000]
平均相似性: 1.776758
化合物数量: 33
最佳化合物ID: CHEMBL5201147
最佳化合物SMILES: Cc1cc(Nc2nc(NCc3ccc(C#N)cc3)nc3ccccc23)n[nH]1
活性类型: IC50
活性值: 83.0 nM
靶标ID: CHEMBL1957
靶标名称: Insulin-like growth factor I receptor
物种: Homo sapiens
相似性打分: 1.933000
相似性区间: [1.403000, 1.933000]
平均相似性: 1.588070
化合物数量: 43
最佳化合物ID: CHEMBL3409725
最佳化合物SMILES: Cc1ccc(CNc2nc3c(c(Nc4cc(C5CC5)[nH]n4)n2)CCC3)cc1
活性类型: IC50
活性值: 149.0 nM
靶标ID: CHEMBL6144
靶标名称: G protein-coupled receptor kinase 6
物种: Homo sapiens
相似性打分: 1.929000
相似性区间: [1.720000, 1.929000]
平均相似性: 1.820500
化合物数量: 16
最佳化合物ID: CHEMBL4874424
最佳化合物SMILES: CCc1cc(Nc2nc(NCc3ccc(Cl)cc3)nc3cccc(OC)c23)n[nH]1
活性类型: IC50
活性值: 16.0 nM
靶标ID: CHEMBL4722
靶标名称: Serine/threonine-protein kinase Aurora-A
物种: Homo sapiens
相似性打分: 1.888000
相似性区间: [1.412000, 1.888000]
平均相似性: 1.719333
化合物数量: 42
最佳化合物ID: CHEMBL559900
最佳化合物SMILES: Cc1cc(Nc2nc(Sc3ccc4ccccc4c3)nc3ccccc23)n[nH]1
活性类型: Ki
活性值: 1.0 nM
靶标ID: CHEMBL1844
靶标名称: Macrophage colony stimulating factor receptor
物种: Homo sapiens
相似性打分: 1.880000
相似性区间: [1.442000, 1.880000]
平均相似性: 1.639200
化合物数量: 5
最佳化合物ID: CHEMBL1997924
最佳化合物SMILES: c1ccc(CNc2nc(Nc3cc(C4CC4)[nH]n3)c3sccc3n2)cc1
活性类型: Ki
活性值: 39.81 nM
靶标ID: CHEMBL1907600
靶标名称: Cyclin-dependent kinase 5/CDK5 activator 1
物种: Homo sapiens
相似性打分: 1.880000
相似性区间: [1.427000, 1.880000]
平均相似性: 1.618500
化合物数量: 10
最佳化合物ID: CHEMBL1997924
最佳化合物SMILES: c1ccc(CNc2nc(Nc3cc(C4CC4)[nH]n3)c3sccc3n2)cc1
活性类型: IC50
活性值: 101.1 nM
靶标ID: CHEMBL5568
靶标名称: Proto-oncogene tyrosine-protein kinase ROS
物种: Homo sapiens
相似性打分: 1.880000
相似性区间: [1.488000, 1.880000]
平均相似性: 1.681133
化合物数量: 3
最佳化合物ID: CHEMBL1997924
最佳化合物SMILES: c1ccc(CNc2nc(Nc3cc(C4CC4)[nH]n3)c3sccc3n2)cc1
活性类型: Ki
活性值: 1.0 nM
5.4 靶标预测准确性评估
在本次靶标预测分析中,化合物31的已知靶标ROS1在预测结果中位列第16位,共发现3个化合物表现出显著的三维结构相似性,其RefTverskyCombo评分值介于1.49至1.88之间。其中,与ROS1激酶结构域具有最高3D匹配性的已知抑制剂为CHEMBL1997924(RefTverskyCombo = 1.88,ROS1 Ki = 1.0 nM)。如前文所述1,化合物31不仅在三维结构上与CHEMBL1997924表现出高度相似性,还成功复现了后者与ROS1激酶结构域相互作用的关键药效特征。基于此双重证据,我们接受ROS1作为化合物31的潜在靶标预测结果。
5.5 反靶标与脱靶效应预测
TrkA作为与ROS1密切相关的反靶标(anti-target),在ROS1抑制剂发现项目中需避免其相互作用以实现选择性抑制。鉴于TrkA与ROS1在结构上的同源性,以及其药理学特性和安全性效应对ROS1抑制剂后续评估的影响,TrkA可作为激酶组选择性评估的代表性靶标。
在本次预测分析中,TrkA作为潜在靶标位列预测结果首位。与化合物31最佳匹配的化合物为CHEMBL5201147,其RefTverskyCombo评分值为1.97,TrkA IC50 = 83.0 nM。这一结果为项目提供了重要警示:在化合物合成与生物活性测试前,需评估其对反靶标的潜在作用。值得注意的是,尽管计算预测提示TrkA结合风险,后续实验验证表明化合物31对TrkA并未表现出显著的抑制活性。这并非是个错误,在3D相似的化合物中,比如CHEMBL1983923,就是个已知的ROS1与TrkA抑制剂。
5.6 预测结果后处理的必要性
需要强调的是,在本次计算分析中,药效团特征由软件算法自动从参比配体中提取,未对关键相互作用位点进行加权处理。此种处理方式可能导致化合物整体相似性得分较高,但局部关键药效特征缺失的情况。
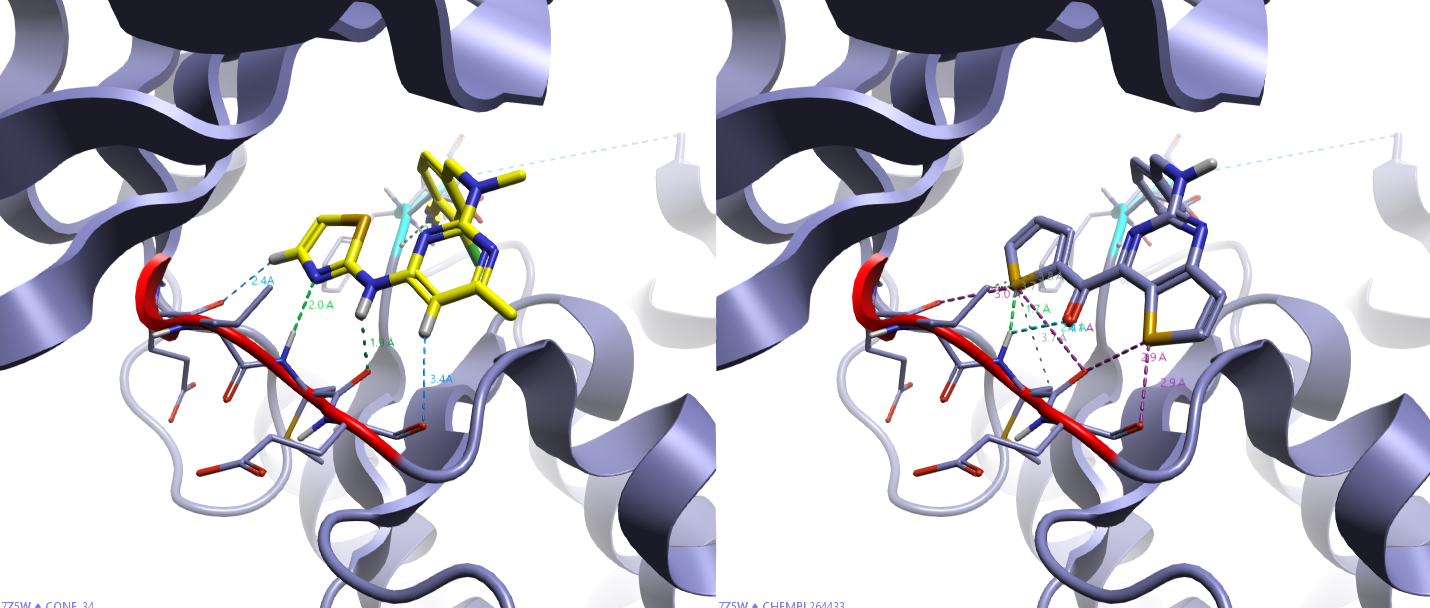
图3. 化合物31(左,ROS1抑制剂)与CHEMBL264433(右,A2A配体)3D叠合图。尽管CHEMBL264433与化合物31整体形状相似性较高(Tc = 1.52),但缺乏与激酶铰链区形成氢键的关键供体基团(红色飘带区域),提示其功能匹配性存疑。
以A2A受体为例,ROSHAMBO2默认以Tanimoto_Combination为打分函数,A2A受体排序第5:
靶标ID: CHEMBL251
靶标名称: Adenosine A2a receptor
物种: Homo sapiens
相似性打分: 1.519850
相似性区间: [1.200071, 1.519850]
平均相似性: 1.292072
化合物数量: 51
最佳化合物ID: CHEMBL264433
最佳化合物SMILES: O=C(c1cccs1)c1nc(NCc2ccccc2)nc2ccsc12
活性类型: Ki
活性值: 25.0 nM
其最佳匹配配体CHEMBL264433虽与化合物31具有较高的3D相似性(Combo = 1.52),但如图3所示,该分子缺乏与激酶铰链区形成关键氢键的供体基团。若将CHEMBL264433视为ROS1抑制剂候选物,则需谨慎评估其结合能力;反之,若考虑将化合物31用于靶标A2A,则需验证其额外氢键供体是否与A2A结合口袋的空间和静电特性兼容。
因此,在严谨的药物发现项目中,建议对初步预测结果进行以下后处理:
- 在靶标结合位点里进行视觉分析,评估结合模式合理性;
- 系统分析关键相互作用是否满足,包括氢键、疏水作用、π-π堆积等;
- 排除存在显著立体化学冲突的预测结果;
- 评估化合物构象能量,排除存在显著构象张力能的匹配结果。
7. 文献
- 查寻分子的准备对基于形状虚拟筛选的影响. http://blog.molcalx.com.cn/2025/10/11/the-impact-of-query-conformation-on-shape-based-virtual-screening.html
- 基于ROCS的靶标预测方案与教程. http://blog.molcalx.com.cn/2017/01/31/rocs-target-fishing.html
- Tu, Y.; Wang, K.; Tan, L.; Han, B.; Hu, Y.; Ding, H.; He, C. Dolichosin A, a Coumestan Isolated from Glycine Tabacina, Inhibits IL-1β-Induced Inflammation in SW982 Human Synovial Cells and Suppresses RANKL-Induced Osteoclastogenesis: From Network Pharmacology to Experimental Pharmacology. J. Ethnopharmacol. 2020, 258 (April), 112855. https://doi.org/10.1016/j.jep.2020.112855.
- Liu, J.; Yang, J.; Hou, Y.; Zhu, Z.; He, J.; Zhao, H.; Ye, X.; Li, D.; Wu, Z.; Huang, Z.; et al. Casticin Inhibits Nasopharyngeal Carcinoma Growth by Targeting Phosphoinositide 3-Kinase. Cancer Cell Int. 2019, 19 (1), 348. https://doi.org/10.1186/s12935-019-1069-6.
- Wang, Y.; Hu, B.; Peng, Y.; Xiong, X.; Jing, W.; Wang, J.; Gao, H. In Silico Exploration of the Molecular Mechanism of Cassane Diterpenoids on Anti-Inflammatory and Immunomodulatory Activity. J. Chem. Inf. Model. 2019, 59 (5), 2309–2323. https://doi.org/10.1021/acs.jcim.8b00862.
- Horvath, D., Marcou, G. and Varnek, A. (2013) “Do Not Hesitate to Use Tversky—and Other Hints for Successful Active Analogue Searches with Feature Count Descriptors,” Journal of Chemical Information and Modeling, 53(7), pp. 1543–1562. Available at: https://doi.org/10.1021/ci400106g.
- 在基于配体3D相似性的虚拟筛选中使用Tversky. http://blog.molcalx.com.cn/2023/04/17/using-tversky-in-vs.html



