摘要:本文介绍了基于配体3D相似性计算两种主要方法Tanimoto与Tversky,并将ROCS Tversky相似性方法与文献报道的9种其它基于配体3D相似性方法与深度学习3D相似性方法在DUD-E数据集上的虚拟筛选性能表现进行了比较。结果表明ROCS Tversky相似性方法在总体虚拟筛选性能(ROC AUC)与早期富集能力(EF1%)上均表现最优,并建议不妨大胆地在虚拟筛选中使用Tversky相似性方法。
肖高铿/2023-03-16
前言
测量分子相似性或不相似性有两个基本组成部分:分子特征(如形状和颜色)的表示和用于量化两种表示之间相似程度的相似性系数。不同的相似性系数量化了不同类型的结构相似性。在基于形状的相似性计算中有三个基本术语:
- selfA: Self overlap or self color score for molecule A
- selfB: Self overlap or self color score for molecule B
- overlapAB: Overlap or color score between molecules A and B
基于配体3D形状的虚拟筛选方法ROCS采用了两方法来衡量两个分子之间的3D相似性程度:Tanimoto系数与Tversky系数1。
Tanimoto系数计算公式如下:
$$Tanimoto_{A,B}={overlapAB \over {selfA + selfB -overlapAB}}$$
Tanimoto相似度计算是对称的,对于形状和颜色,其值始终在0.0和1.0之间。
Tversky系数计算公式如下:
$$Tversky_{A,B}={overlapAB \over {\alpha*selfA + \beta*selfB}}$$
其中α+β=1,ROCS默认α或β=0.95以使得Tversky相似度计算是不对称的。当参数α=β=0.5时,其计算是对称的,并且在一定程度上与Tanimoto相似度计算是相同的。α对参比分子的贡献进行加权,α越大,参比分子的自叠合(self overlap)权重越大;β对数据库分子的贡献进行加权,β越大,数据库分子的自叠合(self overlap)权重越大。
与Tanimoto相似性一样,对于形状,Tversky相似性的值始终在0.0和1.0之间。然而,对于颜色(color)来说,这可能并不总是如此。根据分子A和B之间的颜色原子的数量和类型,|overlapAB|有可能比|selfA|大,并且随着α值的变化,有时会导致TverskyA,B大于1.0。
ROCS计算得到两个Tversky值:1)RefTversky,以参比分子为主Self overlap,也就是α=0.95,偏向参比分子;2)FitTversky,以数据库分子为主self overlap,也就是β=0.95,偏向数据库分子。
相应的,ROCS计算会得到三种Combo打分:
- TanimotoCombo = ShapeTanimoto + ColorTanimoto
- RefTverskyCombo = RefTversky + RefColorTversky
- FitTverskyCombo = FitTversky + FitColorTversky
虽然人们没有达成一致认为一种方法优于另一种方法,但是已有的研究证明Tversky方法在虚拟筛选性能上比Tanimoto方法有很大的提升2,3。
目前已经有众多的验证性文献报道了ROCS的虚拟筛选性能,比如Kearnes3、Koes4与Jiang等人5就用DUD-E等基准数据集系统地对ROCS虚拟筛选性能进行了评估并与其它基于配体3D相似性的方法进行了比较。尽管如此,大部分虚拟筛选方法的性能评估与比较仅针对Tanimoto方法,而对Tversky方法的性能与比较研究则很少见。
本文的目的是再次强调Tversky方法——尤其是RefTverskyCombo方法——的虚拟筛选性能,并引用Jiang5与郑行6等人报道的基于配体3D相似性虚拟筛选方法在DUD-E数据集上的性能表现进行了比较。
结果
最近,Jiang等人5对9种基于配体的3D相似性方法在DUD-E数据集上进行了比较,郑行6等人则公开了采用Uni-Mol深度学习方法进行基于配体3D相似性虚拟筛选的性能,这些数据汇总于表1。
表1. ROCS_TverskyComboScore与其它3D相似性方法在DUD-E数据集上的虚拟筛选性能比较
| 方法 | ROC AUC | EF1% | EF5% | EF10% |
|---|---|---|---|---|
| ROCS_TverskyComboScorea | 0.756 | 25.3 | 8.4 | 5.0 |
| ROCS_ComboScoreb | 0.598 | 12.44 | 4.33 | 2.80 |
| ROCS_ColorScoreb | 0.620 | 13.20 | 4.76 | 3.09 |
| ROCS_ShapeTanimotob | 0.547 | 6.89 | 2.93 | 2.07 |
| Phase Shape_Mmodb | 0.677 | 13.73 | 5.06 | 3.34 |
| Phase Shape_Eleb | 0.674 | 14.57 | 5.50 | 3.57 |
| Phase Shape_Pharmb | 0.692 | 16.03 | 6.00 | 3.84 |
| Shape-itb | 0.541 | 4.73 | 2.50 | 1.91 |
| Align-itb | 0.659 | 12.44 | 4.87 | 3.21 |
| ShaEP_bestb | 0.658 | 10.28 | 4.32 | 2.99 |
| ShaEP_shapeb | 0.625 | 8.86 | 3.82 | 2.76 |
| ShaEP_ESPb | 0.606 | 6.36 | 3.03 | 2.25 |
| SHAFTSb | 0.733 | 19.13 | 6.97 | 4.37 |
| WEGAb | 0.645 | 12.14 | 4.67 | 3.16 |
| LIGSIFTb | 0.718 | 16.22 | 5.79 | 3.78 |
| LS-alignb | 0.699 | 15.88 | 5.74 | 3.66 |
| Uni-Molc | – | 20.40 | 8.48 | 5.59 |
a:肖高铿7;b:Jiang等人5;c:郑行6
可以发现,采用Tversky( α=0.95)相似性方法的ROCS TverskyComboScore的虚拟筛选总体性能最佳,在DUD-E 102个靶标上的平均ROC AUC=0.756,优于其它所有的方法,如图1所示。
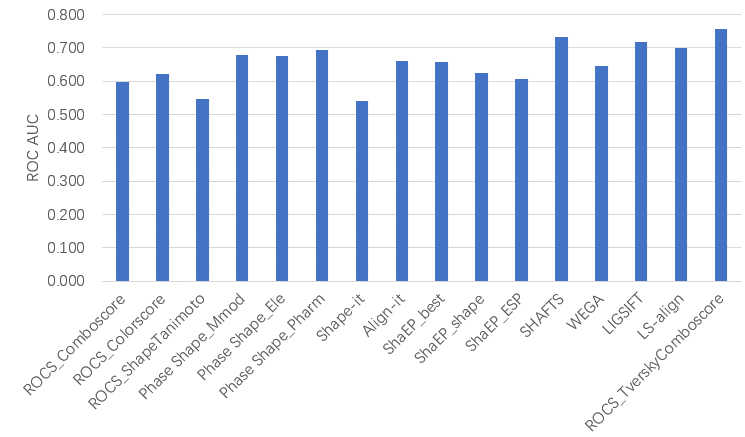
图1. ROCS TverskyComboScore与其它9种3D相似性方法在DUD-E数据集上虚拟筛选总体性能(ROC AUC)比较
同样,采用Tversky(α=0.95)相似性方法的ROCS TverskyComboScore的虚拟筛选方法表现出了最强的早期富集能力。在DUD-E 102个靶标上,富集因子EF1%均值=25.3,优于所有的其它方法,并且大幅领先于第二名Uni-Mol(EF1%=20)与第三名SHAFTS(EF1%=19.13),如图2所示。
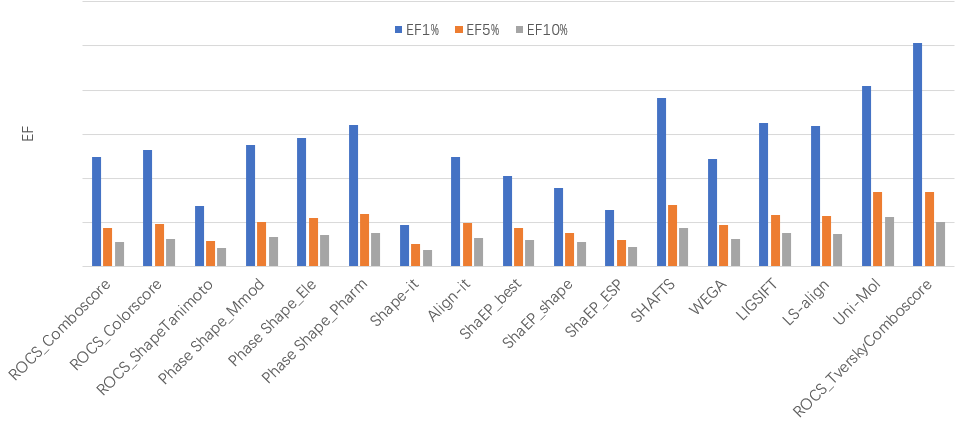
图2. ROCS TverskyComboScore与其它9种3D相似性方法在DUD-E数据集上虚拟筛选富集因子EF(Top 1%,5%,10%)的比较
总的来说,采用Tversky(α=0.95)相似性方法的ROCS TverskyComboScore不仅在虚拟筛选总体性能上优于其它基于配体3D相似性方法与深度学习方法,而且在早期富集能力上也优于其它方法。
方法
详细的ROCS RefTversky相似性计算方法请参见前文7。
结论
总的来说,采用Tversky(α=0.95,即RefTversky)相似性方法的ROCS,不仅在虚拟筛选总体性能(ROC AUC)上优于其它基于配体3D相似性方法与深度学习方法,而且在早期富集能力(EF1%)上也优于其它方法,敬请大胆地在基于配体的3D相似性虚拟筛选中使用Tversky相似性打分。
文献
- ROCS. https://docs.eyesopen.com/applications/rocs/theory/measure_similarity.html
- Horvath, D.; Marcou, G.; Varnek, A. Do Not Hesitate to Use Tversky—and Other Hints for Successful Active Analogue Searches with Feature Count Descriptors. J. Chem. Inf. Model. 2013, 53 (7), 1543–1562. https://doi.org/10.1021/ci400106g.
- Kearnes, S.; Pande, V. ROCS-Derived Features for Virtual Screening. J. Comput. Aided. Mol. Des. 2016, 30 (8), 609–617. https://doi.org/10.1007/s10822-016-9959-3.
- Koes, D. R.; Camacho, C. J. Shape-Based Virtual Screening with Volumetric Aligned Molecular Shapes. J. Comput. Chem. 2014, 35 (25), 1824–1834. https://doi.org/10.1002/jcc.23690.
- Jiang, Z.; Xu, J.; Yan, A.; Wang, L. A Comprehensive Comparative Assessment of 3D Molecular Similarity Tools in Ligand-Based Virtual Screening. Brief. Bioinform. 2021, 22 (6), 1–17. https://doi.org/10.1093/bib/bbab231.
- 郑行. 分子3D预训练模型Uni-Mol在药物研发中的应用|CADD领袖论坛第3期. https://www.bilibili.com/video/BV1Wd4y137q1
- 肖高铿. ROCS——用形状技术进行高效的虚拟筛选. 墨灵格的博客. 2020-03-01. http://blog.molcalx.com.cn/2022/03/15/rocs.html
联系我们,安排在线会议介绍、商务合作与软件试用
您有两种方法可以用上ROCS:1)采购软件,安装在自己的电脑上;2)使用我们的虚拟筛选计算服务。了解更多,请联系我们。



