摘要:本文用DUDE数据集对基于配体的ROCS虚拟筛选方法进行了性能评估,评估指标包括总体性能ROC AUC、早期富集能力logAUC与BEDROC。并在AUC表征的总体性能上与其它同类方法进行性能比较;在logAUC与BEDROC指标上与最流行的分子对接方法GLIDE、GOLD等进行了比较。结果表明,ROCS与同类基于分子形状方法相比,具有最好的总体性能;与流行的分子对接方法相比,在更多的算例上显示出早期富集能力,并与分子对接方法互补;ROCS非常快速、高效,每CPU核心每秒可完成50多个化合物的虚拟筛选。
肖高铿/2020-03-01
2022-03-28:添加钰沐菡公开课的视频录像
2022-04-03: 添加部分图表、并对部分数据进行了修订
2023-03-19: 补充了Jiang等人对9种基于配体3D相似性虚拟筛选的性能数据
1.ROCS与分子形状

图1. 分子的形状(左)与静电(右)是与结合直接相关的两个物理性质
形状与静电是化合物两个基本的物理性质(见图1),也是与蛋白-配体相互识别直接相关的两个基本性质:如果一个化合物与蛋白结合位点在形状与静电上互补,那么这个化合物就自然而然地成为该蛋白的配体(见图2);如果两个化合物具有相似的形状与静电,那么这两个化合物就可能结合到同一蛋白位点、具相似的生物学活性。除此之外的其它性质都是这两个性质的近似:1)2D结构相似的两个化合物可能形状与静电相似;2)药效团相似的两个化合物可能具有相似的静电。因为静电信息量太大而导致计算得很慢,药效团是静电的近似,所以常常采用形状与药效团来快速地比较化合物的3D相似性。
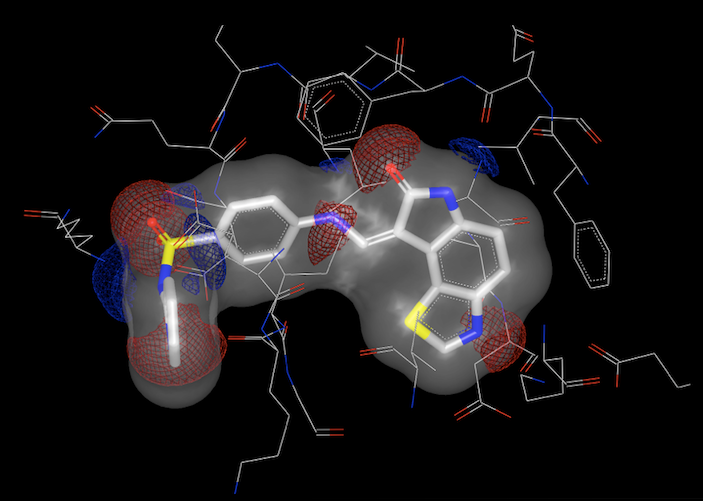
图2. 配体-蛋白的形状与静电互补是分子识别的基础
ROCS是OpenEye Applications软件包中的一个强大的基于配体的虚拟筛选工具[1],它将分子描述为内嵌了药效团特征的形状、然后通过形状比较来快速地识别潜在的活性化合物[2],流程如图3所示。
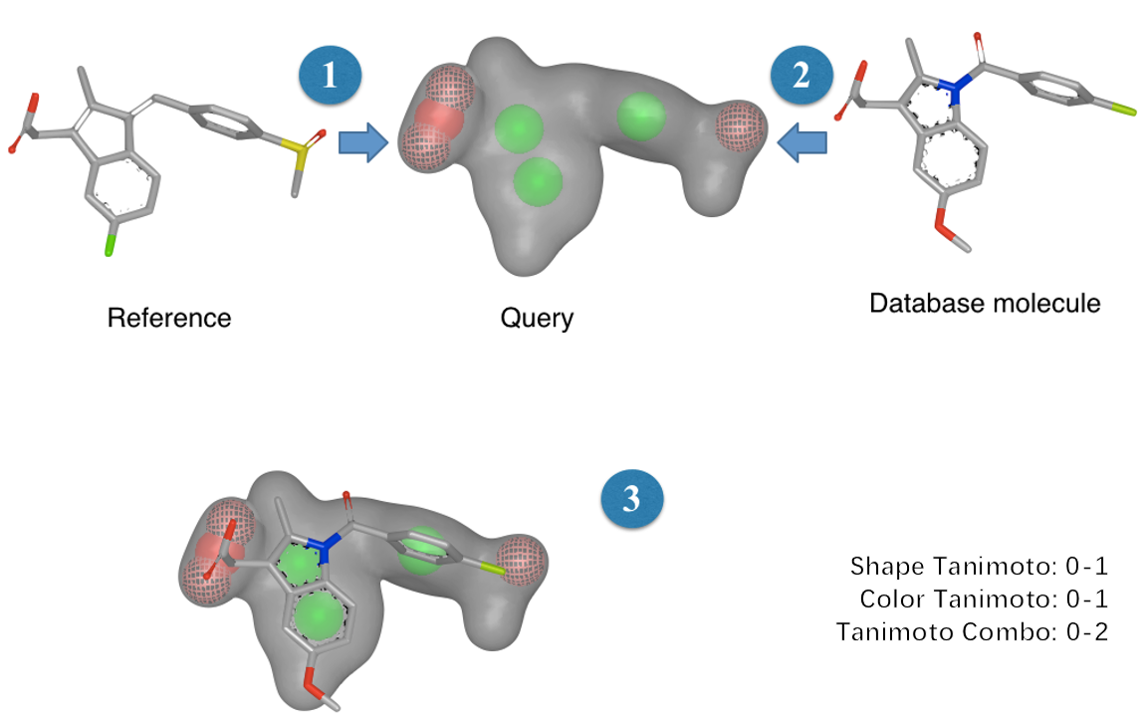
图3. 用ROCS进行虚拟筛选的流程。1)query准备:将参比化合物表征为内嵌药效团特征的形状;2)数据库化合物的叠合:将数据库化合物的形状与药效团与query的进行比较、叠合,搜索出所有的与query匹配的化合物;3)打分、排序:每个数据库化合物依匹配程度进行打分、排序。
如图3所示,ROCS对化合物进行叠合与比较之后,给出三种打分值:形状相似性(Shape similarity),颜色相似性(Color Similarity,即化学特征相似性或药效团相似性)以及形状与颜色相似性的和(Combo)。ROCS的相似性计算方法包括Tanimoto与Tversky,以Tanimoto相似性为例,有:
TanimotoCombo = Shape Tanimoto + Color Tanimoto
其中Shape Tanimoto与Color Tanimoto的取值区间为[0,1],0代表的没有相似性,1代表完美一致。以Shape Tanimoto为例,通常如果达到0.7或以上,以化学家的角度可以肉眼看到分子存在显著的形状相似性。而ComboTanimoto为Shape与Color Tanimoto之和,因此取值范围为[0,2],0代表着两个化合物之间没有关联,而2代表的完美一致。
已有的研究表明,ROCS在总体性能和稳健性方面都可与基于结构的虚拟筛选方法相媲美,并且通常优于基于结构的方法[3-5];针对特定靶标寻找有意思的新骨架通常被认为是计算技术难以解决的,ROCS是此类问题的一种高效解决方法[6]。
此前Koes等人[7]已经在DUDE数据集[8]上对ROCS等形状方法的虚拟筛选性能进行了评估,Hawkins等人[3]以及McGaughey等人[9]还系统地将分子形状技术与分子对接技术的虚拟筛选性能进行了比较。本文的目的是将最近报道的分子形状技术、分子对接方法与深度学习方法在DUDE上测试得到性能指标进行汇总,然后用同样的性能指标来评估ROCS,以便读者可以从不同的角度对不同方法进行性能比较。
2. 方法
2.1 DUDE数据集的准备
测试用的actives与decoys来自DUDE,直接下载sdf格式文件,然后用OpenEye Applications软件包中OMEGA[10]的pose方法在默认参数下进行结构准备,将结果保存为oeb.gz格式。
acitves化合物的结构准备:
1 2 3 4 5 6 7 | $OE_DIR/bin/oeomega pose -in actives_final.sdf \ -out actives.oeb.gz \ -flipper true \ -enumNitrogen true \ -enumRing true \ -prefix actives \ -useGPU |
decoys化合物的结构准备:
1 2 3 4 5 6 7 | $OE_DIR/bin/oeomega pose -in decoys_final.sdf \ -out decoys.oeb.gz \ -flipper true \ -enumNitrogen true \ -enumRing true \ -prefix decoys \ -useGPU |
构象搜索之后分别得到actives与decoys数据集的构象系综数据库:actives.oeb.gz与decoys.oeb.gz。
需要注意的是:一般情况下ROCS虚拟筛选用omega的rocs方法就可以,分子对接目的用pose方法。我在这里用pose方法是因为我很久之前已经用pose方法准备好了。
2.2 query的准备
ROCS是形状技术快速进行分子叠合的方法,在虚拟筛选过程中需要每个分子与query进行形状与药效的比较。在本文中,所有的query都根据DUDE受体的PDB ID从结合位点里提取的配体。在提取之前用spruce[11]进行了蛋白的结果准备,但是没有进行任何的检查、电荷或质子化状态的修改。虽然这种默认模式显然不完全合理,但是可以得到“默认”模式下的性能。
2.3 虚拟筛选
除了将mpi_np设置为24以便使用24核心进行虚拟筛选、将besthits设置为0以确保ROCS输出所有的化合物计算结果之外,其余的参数均采用默认值。
acitves化合物的虚拟筛选:
1 2 3 4 5 | $OE_DIR/bin/rocs -mpi_np 24 \ -query ligand.mol2 \ -dbase actives.oeb.gz \ -prefix actives \ -besthits 0 |
decoys化合物的虚拟筛选:
1 2 3 4 5 | $OE_DIR/bin/rocs -mpi_np 24 \ -query ligand.mol2 \ -dbase decoys.oeb.gz \ -prefix decoys \ -besthits 0 |
2.4 性能评估
鉴于DUDE下载的actives与decoys数据集因为互变异构体与质子化状态的枚举,导致一个化合物会以不同的形式多次出现、并可能被虚拟筛选多次命中,因此对虚拟筛选结果按化合物名称进行去重,同名化合物仅以其中打分最高那个来代表。所有的性能评估指标都在去重的基础上进行计算。此外,对接失败的化合物给予一个很低的打分值0。
本文用的评价指标包括AUC、logAUCλ=0.001、α=321.9、80.5、20.0时的BEDROC等等。这些指标的具体计算方法见如何进行虚拟筛选的方法学验证[12]。
在本文中,分别对ROCS的RefTverskCombo与TanimotoCombo两种打分函数进行了性能评估,分别表示为ROCS-RefTverskCombo与ROCS-TanimotoCombo。
3. 结果
3.1 以AUC表征的总体虚拟筛选性能
如前文所述[12],ROC AUC常用来定量比较不同方法的性能。已有很多文献报道了ROCS的虚拟筛选性能,其中Koes等人[7]还用DUDE数据集对ROCS进行了性能评测。与Koes等人[7]的评测结果相比,就TanimotoCombo为打分函数评价的AUC而言,如图4的散点图所示,两者的AUC在统计学上具有极其显著的相关性:Pearsion相关性系数r=0.953,Spearman秩相关性系数ρ=0.945,均方差MSD=0.00319。这也说明,本文计算与文献计算的结果基本一致。
图4、在DUDE数据集的102个靶标上,Koes等人[7]计算的与本文计算的AUC比较。
如图5所示,本文根据TanimotoCombo计算的AUC均值与中值分别为0.69与0.72,略高于Koes等人报道的0.66与0.69,但是两者之间并不具有统计学意义上的显著性差异(p-value=0.147,计算见5.1)。

图5、在DUDE数据集的102个靶标上,Koes等人[7]计算的与本文计算的AUC中值与均值比较。
ROCS提供了两种相似性指数算法:Tanimoto与Tversky。除了上面已经讨论过的TanimotoCombo,我们还鼓励使用Tversky相似性。如图5所示,可以发现RefTverskyCombo相似性(灰色)计算的AUC均值与中值都为0.76,比TanimotoCombo相似性的AUC均值(0.69)与中值(0.72)都要高,Nemenyi post-hoc test表明具有统计学意义的显著性差异(p=0.001,计算见5.1),因此RefTverskyCombo相似性提升了TanimotoCombo的虚拟筛选性能。
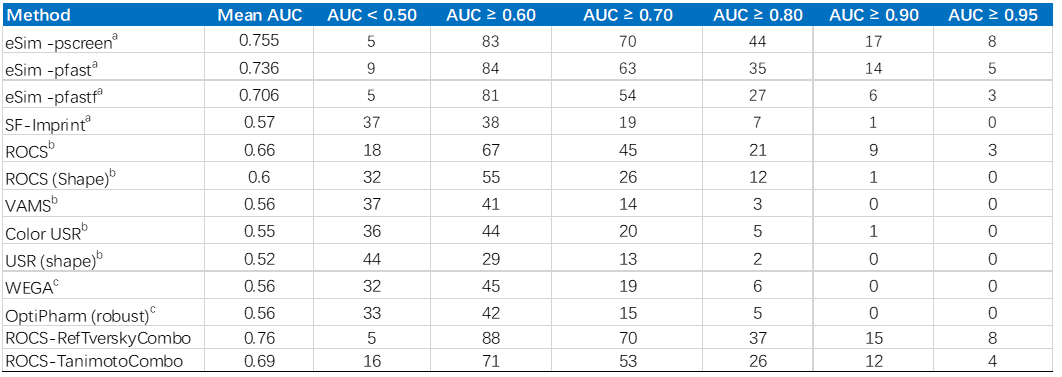
表1、多种基于配体的虚拟筛选方法在DUDE数据集上的AUC分布(数值表示靶标数量)
其中a:文献[13];b:文献[7];c:文献[14]
表1汇总了文献报道的各种3D相似性虚拟筛选方法在DUDE上的性能表现,数值为在102个DUDE靶标中AUC落在特定范围的靶标数。比如AUC小于0.5列表示各种方法失败的虚拟筛选靶标数量。就AUC值的分布而言,与其它的基于配体的虚拟筛选方法相比,采用RefTversky为相似性指数的ROCS与eSim-pscreen相当,而优于其它方法。就ROCS而言,同时考虑了分子形状与药效团相似的TanimotoCombo比仅考虑了分子形状的打分方法(ROCS shape)性能要更好。
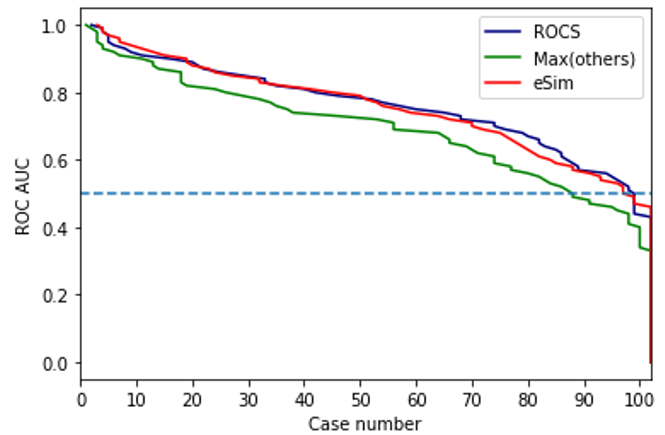
图6、ROCS与其它基于分子形状形式性方法在DUDE数据集102个靶标上的AUC值比较。横坐标:算例数;纵坐标:AUC值。其中ROCS是指RefTverskyCombo打分;eSim为pscreen模式;max(others)指其它所有形状相似性方法AUC最大值。
绘制ROCS(RefTverskyCombo),eSim (pscreen)与其它形状相似方法(取最大值)的AUC在DUDE 102个靶标上的算例数曲线,如图6所示,可以清楚地看到ROCS(蓝色)与eSim(红色)的AUC曲线基本重合在一起,说明这两种方法从AUC角度看总体性能基本一致;两者明显地优于其它方法,因为其它方法取AUC最大值的曲线(绿色)位于ROCS(蓝色)与eSim(红色)曲线的下方。
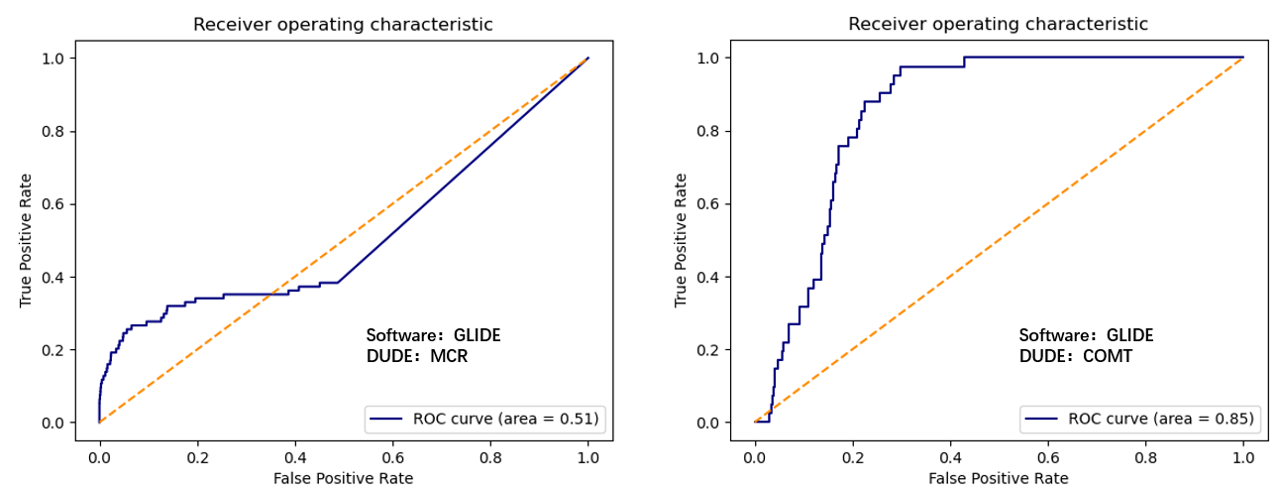
虽然AUC被广泛用于评估虚拟筛选性能,然而AUC不能正确反映ROC曲线的形态:AUC高的不代表在早期也可以富集到活性化合物,AUC低的未必不能在早期富集到活性化合物,更具体的讨论见前文[12]。如图S1所示,在MCR算例上,虽然GLIDE虚拟筛选的AUC=0.51,但是ROC曲线的在非常早期时(横坐标很小)的时候就开始紧贴着纵坐标,因此GLIDE在这个靶标上表现出良好的早期富集能力。而在COMT算例上,虽然AUC=0.85,但是在非常早期的时候,ROC曲线位于随机方法对角线的下方,因此GLIDE在COMT这个靶标上没有表现出好的早期富集能力。这说明,AUC小不一定没有富集能力,AUC大不一定有好的早期富集能力。
早期富集性能的评估很重要,因为在对数万、数百万、甚至数亿个分子的虚拟筛选中,通常只有排名靠前的0.1%化合物才会受到重视而下一步的实验评估。如果在回溯性虚拟筛选计算中显示已知活性化合物仅在10%左右开始被识别出来,则预期在虚拟筛选中可能会遗漏新的活性化合物。因此早期富集能力在实践中具有更重要的指导意义,接下来主要讨论ROCS的早期富集能力。
3.2 logAUC表征的早期富集能力
logAUC并不是专门为评估早期富集能力而设计,仅仅只是相对ROC AUC而言,突出了早期富集的性能。在不同的文献中,有不同的logAUC表述,其中一种是减扣掉随机曲线下面积(0.14462)后的logAUC,也就是adjusted-logAUC。adjusted-logAUC小于等于0,表示该方法不如随机筛选;而adjusted-logAUC大于0,表示优于随机筛选。为了方便比较,在接下来的部分里,提到logAUC的时候是指adjusted-logAUCλ=0.001。
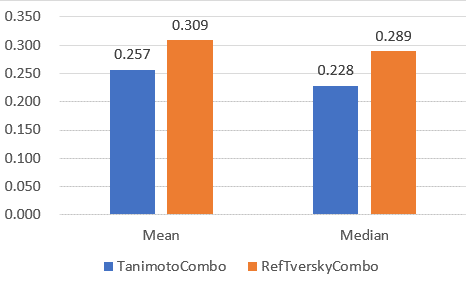
图7、ROCS的TanimotoCombo与RefTverskyCombo两种打分函数在DUDE数据集102个靶标上的logAUC均值与中位数比较
如图7所示,在DUDE 102个靶标上测试的结果表明,ROCS基于Tversky相似性指数的虚拟筛选比基于Tanimoto相似性指数的虚拟筛选有着更好的早期富集能力,主要体现在RefTverskyCombo打分比TanimotoCombo打分的具有更高的logAUC均值与中值。如表2所示,在DUDE数据集上,ROCS的TanmotoCombo打分函数虚拟筛选在102个靶标里有10个logAUC小于0没有表现出富集能力;而在采用RefTverskyCombo为打分函数时,仅有两个靶标的logAUC小于0而没表现出富集能力。鉴于此,再一次鼓励大家在虚拟筛选的时候尝试使用Tversky相似性指数方法。
表2、ROCS与分子对接方法在logAUC表征的虚拟筛选性能比较

其中,f:文献[15];e:文献[16];h:文献[17],并将原文的logAUC变换为adjusted-logAUC;
尚未有文献报道过其它基于分子形状虚拟筛选方法的logAUC性能,因此本文汇集了基于分子对接虚拟筛选方法在DUDE上的logAUC性能表现,如表2与图8所示。
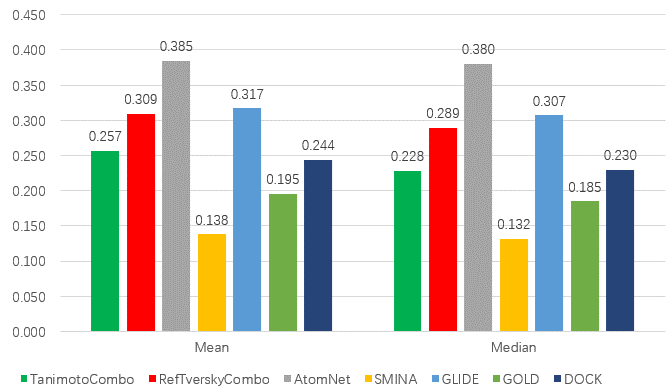
图8、ROCS以及5种基于结构的虚拟筛选方法在DUDE数据集上logAUC均值与中位数的比较
可以发现,从logAUC的中值与均值、以及各个范围的分布上看,AtomNet有着最高的均值与中值,明显地优于其它方法。而分子对接方法Smina的均值与中值最低,是所有方法中性能最差的。紧接AtomNet之后的是ROCS RefTverskyCombo与GLIDE,它们的均值非常接近,分别为0.309与0.317,Nemenyi post-hoc Test结果表明(见5.2小节)两者之间没有统计学意义上的显著差异(p-value=0.702)。这说明,ROCS RefTverskyCombo与GLIDE分子对接方法以logAUC表征的富集能力相当。再接下来是ROCS TanimotoCombo与DOCK,两者的logAUC均值分别为0.257与0.244,Nemenyi post-hoc Test结果表明(见5.2小节)两者之间没有统计学意义上的显著差异(p-value=0.118)。而GOLD的logAUC均值在ROCS TanimotoCombo与DOCK之后。因此以logAUC表征虚拟筛选性能排序从高到低依次是:AtomNet,ROCS RefTverskyCombo~GLIDE,ROCS TanimotoCombo ~ DOCK,GOLD,Smina。
3.3 BEDROC表征的早期富集能力
BEDROC是另一个常用于评估虚拟筛选早期富集能力的指标。然而,BEDROC并不是一个直观的量,在Chaput的研究中[18],发现α=321.9、80.5与20.0的BEDROC对方法的性能评估与EF0.5%、EF2%与EF8%相当,这使得我们大体对BEDROC有个间接理解。
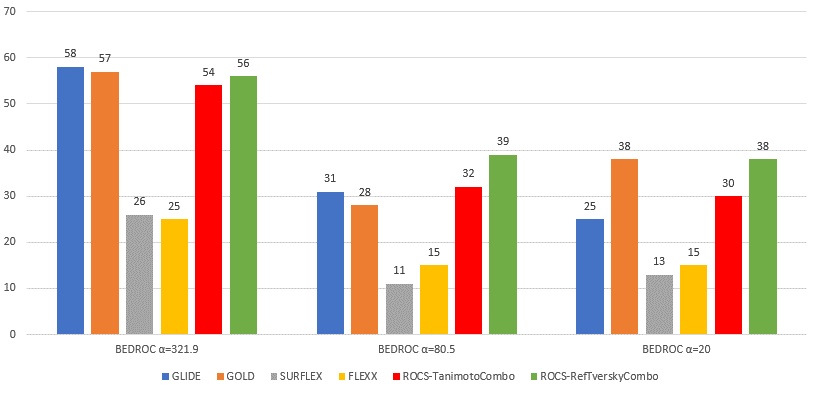
图9. 不同方法在DUDE数据集上三种α参数下的BEDROC表征的早期富集性能比较,数值为BEDROC大于0.5的靶标数量
Chaput等人[18]提出的虚拟筛选性能的评估方法是:将BEDROC大于0.5视为成功的虚拟筛选,计算每个方法成功虚拟筛选的算例数,算例数越多的方法虚拟筛选性能越好。为了方便比较,将Chaput的结果[18]与本文的ROCS结果汇集在图9中,其中GLIDE、GOLD、SURFLEX与FLEXX的数据是根据Chaput等人[18]的图表Figure S1识别而来。
如图9所示,当α=321.9时,BEDROC表征的虚拟筛选性能排序依次是:Glide, GOLD, ROCS,Surflex~FLEXX。这是因为Glide有最多的算例数(58)其BEDROC不低于0.5,接着是GOLD(57)与ROCS RefTverskyCombo(56)以及ROCS TanimotoCombo(54),最后是SURFLEX(26)与FLEXX(25)。在BEDROCα=80.5表征的早期富集能力上,ROCS RefTverskyCombo有39个算例数的BEDROCα=80.5不低于0.5而居首;ROCS TanimoCombo次之(32个算例数);接下来是GLIDE(31算例)与GOLD(28算例);Surflex(21算例)与FLEXX(15算例)则排在最后。在BEDROCα=20.0表征的早期富集能力上,ROCS RefTverskyCombo与GOLD的性能相当且表现最优,BEDROCα=20.0大于0.5的算例数均为38个;ROCS TanimotoCombo与GLIDE性能表现次优,算例数分别为30与25个;最后是FLEXX与SURFLEX,算例数分别为13与15个。总的来说,就BEDROC在三种α参数大于0.5的算例数而言,ROCS性能优于大多数的分子对接方法,这与之前Hawkins[3]与McGaughey[9]关于基于配体的方法ROCS优于基于结构的方法结论一致。
需要强调的是,BEDROC大于0.5的算例数是“总数”的比较,并不代表其中一个方法在各个靶标上都优于另一个方法。因此有必要进一步了解:是否不同的方法有各自擅长的靶标?由于Chaput等人[18]的研究仅公开了BEDROCα=80.5的数据,因此接下来以该指标对不同的虚拟筛选方法进行考察,以确定不同方法之间的真正性能关系。
鉴于GLIDE是性能表现最好的分子对接方法,下面以GLIDE作为分子对接方法的代表与ROCS进行比较。首先统计ROCS RefTverskyCombo与GLIDE在BEDROCα=80.5不低于0.5的算例数。如图10所示,在DUDE的102个算例中,ROCS RefTverskyCombo在39个算例上虚拟筛选成功(BEDROCα=80.5不低于0.5);Glide在31个算例上虚拟筛选成功。总体看起来ROCS比GLIDE有更多的算例获得成功,有19个算例两者都获得成功;有22个算例仅ROCS虚拟筛选成功而glide失败;14个算例仅GLIDE虚拟筛选成功而ROCS失败。这充分说明两种方法是互补的,ROCS与GLIDE合起来总共在53个算例上获得成功。

图10. 在DUDE数据集上,ROCS RefTverskyCombo与GLIDE的BEDROCα=80.5不低于0.5的算例数
GLIDE在DUDE数据集上的性能表现有多篇文献报道:根据Wang等人[19]的研究为33,根据Shen等人[18]的研究为29;而Chaput等人[17]报道为31。这说明不同文献统计出来BEDROCα=80.5大于0.5的算例数略有不同,但是基本一致,不管用哪个值,对性能的评估结果是一样的。
ROCS与GLIDE的这种互补关系使得通过数据融合策略来提高虚拟筛选性能成为可能,Svensson等人[20]的研究证明了这一点,因此鼓励大家在有条件的情况下尽量同时使用不同方法进行虚拟筛选,然后用数据融合策略合并结果。
从BEDROCα=80.5均值上看,如图11所示,GOLD与ROCS RefTverskyCombo的性能最优,接着是GLIDE与ROCS TanimotoCombo,而Surflex与FlexX排在最后。此排序只是大体的性能排序,得到的结论与根据BEDROCα=80.5大于0.5算例数评估的结果也不同,也不像前面那样可以分析出不同方法之间的互补关系。

图11. 不同方法在DUDE数据集上BEDROCα=80.5的均值与中值
3.4 计算速度
根据Koes等人的报道[7],计算速度为每秒50个分子。在本次的计算中,以PDE5A的数据集的decoy为例,统计信息如下:
1 2 3 4 5 6 | Number of Molecules Processed = 27969 Number of Molecules Failed = 20 Average time/molecule = 0.201044 Average Number of Rotors = 7.034599 Average Number of Conformers = 356.125458 Elapsed Time = 5623.000000 |
也就是说,PDE5A的decoy数据集包含27969个分子,OMEGA成功处理了27949个分子,每个分子平均356个构象。ROCS进行虚拟筛选的统计信息如下:
1 2 3 4 5 6 7 8 9 10 11 | Run time(sec) : 305.0 Percent complete : 100.0 Molecules read : 27949 Molecules/sec : 91.6 Hitlist size : 0 Current worst score : -1.00 Average conf/sec : 32633.9 Molecule read failures: 0 #warnings : 0 #errors : 0 |
在本文测试中,共使用了24个Intel(R) Xeon(R) CPU E5-2683 v3 @ 2.00GHz核心进行计算,则每核心的计算速度为:1360构象/秒。如果每个化合物25个构象,则每核心的计算速度为54分子/秒,这与Koes等人的报道[7]每秒50个分子基本一致,这是一个非常快速、高效的虚拟筛选方法。
3.5 ROCS虚拟筛选性能对数据库化合物的构象数不敏感
ROCS的虚拟筛选性能对数据库化合物的构象数并不敏感。DUDE数据集的平均AUC与化合物的最大构象数关系如图12所示,只有当最大构象数为1时,ROCS的虚拟筛选性能才显著下降。这为ROCS虚拟筛选的化合物库准备提供了依据:在默认条件下,OMEGA ROCS使用50个构象、FASTROCS使用25个构象。
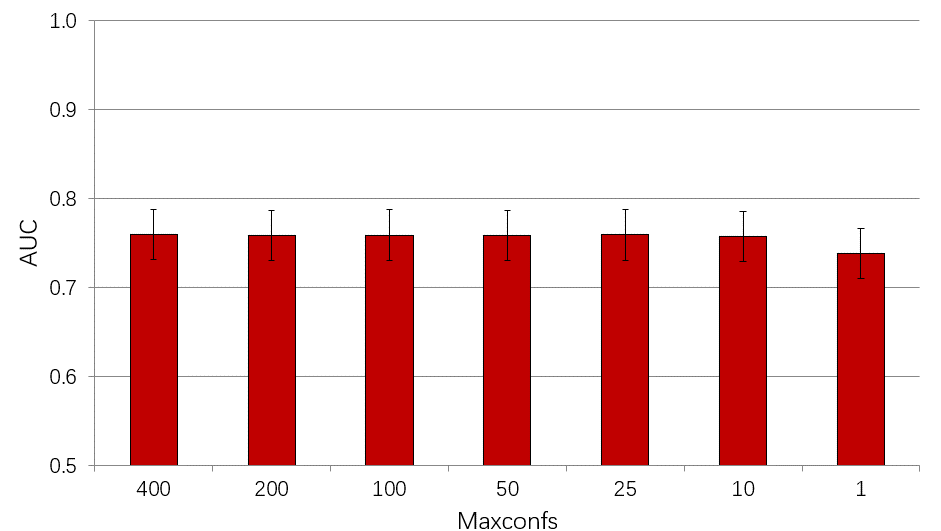
图12. ROCS对化合物库的构象数不是很敏感
在本文的测试中个,omega用的pose参数是根据化合物可旋转键数量动态调整每分子200-800个构象;而在Koes的研究中,每个分子设定最大构象数为25[7]。如图4所示,在DUDE的102个靶标上,两种不同构象数的虚拟筛选AUC基本成线性关系(Pearsion相关性系数r=0.890);如图5所示,AUC的中值与均值也没有显著的统计学差异(p=0.147)。这进一步证明了ROCS的虚拟筛选性能对化合物的构象数并不敏感。
3.6 Query(参比分子):生物活性构象与计算构象相比优势不大
在上面的测试中,作为query的参比分子是从蛋白-配体复合物结构里提取的,其构象为x-ray测得的生物活性构象。如果query的生物活性构象未知,而是一个低能构象则虚拟筛选性能如何?为了回答这个问题,用OMEGA对共晶结构的配体进行了构象搜索,将能量最低的那个构象作为query进行ROCS虚拟筛选。比较生物活性构象(X-Ray)与计算构像(OMEGA)query在ROCS虚拟筛选后的AUC(以RefTverskyCombo为打分函数),结果如图13所示:生物活性构象query的AUC比计算构象query的AUC略高,中值分别为0.765(CI95%=[0.733,0.787])与0.747(CI95%=[0.707,0.765])。这说明比起生物活性构象的query,计算构象query的虚拟筛选性能略有降低,但是这种降低不具备统计学意义。
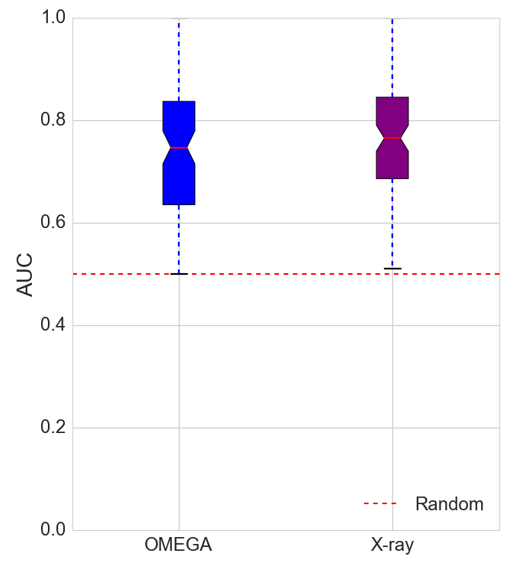
图13. 生物活性构象(X-Ray)与计算构像(OMEGA)query在ROCS虚拟筛选后的AUC比较
在Brickelmaier等人[21]的一项研究中,在没有参比化合物活性构象、不了解靶点的情况下进行ROCS虚拟筛选从已经进入临床研究、上市的化合物中发现新的JC病毒抑制剂,这进一步验证了ROCS对参比化合物活性构象不敏感这个结论。
4. 结论
在本文中,用DUDE数据集对ROCS虚拟筛选性能进行了评估,评估指标包括总体性能ROC AUC、早期富集能力logAUC与BEDROC。并在ROC AUC表征的总体性能上与其它同类方法进行性能比较;在logAUC与BEDROC上与最流行的分子对接方法进行了比较。
结果表明,ROCS使用分子形状与药效团作为打分函数比仅使用形状作为打分函数的虚拟筛选性能要好;同样使用了形状与药效团作为打分函数,RefTerverskyCombo比TanimotoCombo的性能更好。因此,推荐在用ROCS进行虚拟筛选时,不妨试试Tversky相似性指数。
从AUC表征的总体虚拟筛选性能角度上看,ROCS与Surflex eSim的性能相当并大幅优于其它基于分子形状相似性虚拟筛选方法,比如WEGA、OptiPharm、USR与VAMS。
从logAUC表征的早期富集能力上看,AtomNet性能最优;ROCS RefTverskyCombo与GLIDE性能相当,排在第二;ROCS TanomotoCombo与DOCK相当,排在第三;接着是GOLD;最后是Smina。
根据不同α参数的BEDROC大于等于0.5的算例数对不同虚拟筛选性能进行了比较。当α=321.9时,ROCS的虚拟筛选性能最佳,RefTverskyCombo与TanimotoCombo成功的算例数分别为61与62;GLIDE与GOLD相当,成功算例数分别为58与57;而SURFLEX与FLEXX成功的算例数为26与25,排名最后。当α=80.5时,ROCS RefTverskyCombo性能最佳,成功的算例数为42;TanimotoCombo次之,成功的算例数为35;之后为GLIDE与GOLD相当,成功算例数分别为31与28;最后为SURFLEX与FLEXX,成功的算例数为11与15。当α=20.0时,ROCS RefTverskyCombo与GOLD性能最佳,成功算例数均为38;TanimotoCombo与GLIDE次之,成功算例数均为28与25;最后为SURFLEX与FLEXX,成功的算例数为13与15。
根据不同α参数BEDROC大于等于0.5的算例数上看,ROCS的性能比分子对接方法有优势。进一步分析发现,基于配体的ROCS与GLIDE为代表的分子对接相比是互补的,主要体现在:在DUDE的102个靶标中23个上,ROCS成功虚拟筛选而GLIDE失败;在12个靶标上,GLIDE成功虚拟筛选而ROCS失败。
从BEDROCα=80.5均值与中值上看,ROCS RefTverskyCombo的性能最优,接着是GOLD,再接着是ROCS TanimotoCombo与Glide,而Surflex与FlexX排在最后。此排序只是大体的性能排序,也不像前面那样可以得出不同方法之间的互补关系。
ROCS是一种高效的虚拟筛选方法,每CPU核心每秒可完成1360的构象比较,如果一个化合物25个构象,则每秒可完成50多个化合物的虚拟筛选。
总的来说,ROCS与同类基于分子形状方法相比,具有最好的总体性能;与流行的分子对接方法相比,在更多的算例上显示出早期富集能力,并与分子对接方法互补; ROCS非常高效,每CPU核心每秒可完成50多个化合物的虚拟筛选。因此ROCS是一个高效的虚拟筛选工具,推荐大家在有确切起始活性化合物的情况下使用ROCS进行虚拟筛选。
5. 支持信息
5.1 ROCS打分函数计算的AUC均值比较
5.2 几种虚拟筛选方法logAUC的均值比较
5.3 GLIDE虚拟筛选两个AUC与早期富集能力背离的算例
根据Shen等人[17]公开的GLIDE打分数据(见5.4),绘制DUDE的COMT与MCR两个靶标的ROC曲线,结果如图S1所示。
图S1. GLIDE虚拟筛选两个AUC与早期富集能力背离的算例。左:MCR算例;右:COMT算例
在MCR算例上,虽然GLIDE虚拟筛选的AUC=0.51,但是ROC曲线的在非常早期时(横坐标很小)的时候就开始紧贴着纵坐标,因此GLIDE在这个靶标上表现出良好的早期富集能力。而在COMT算例上,虽然AUC=0.85,但是在非常早期的时候(在假阳性率3%之前),ROC曲线位于随机方法对角线的下方,因此GLIDE在COMT这个靶标上没有表现出好的早期富集能力。这说明,AUC小不一定没有富集能力,AUC大不一定有好的早期富集能力。
表S1、GLIDE在DUDE MCR与COMT两个靶标上的性能指标
| 指标 | MCR | COMT |
|---|---|---|
| AUC | 0.51 | 0.85 |
| CI 95%(1000 resample) | 0.4482 – 0.5775 | 0.8238 – 0.8785 |
| ER1% | 12.8 | 0 |
| logAUCλ=0.1% | 24.7 | 30.8 |
| adjusted-logAUCλ=0.1% | 10.3 | 16.3 |
| BEDROC α=321.9 | 0.40 | 0 |
| BEDROC α=80.5 | 0.22 | 0.01 |
| BEDROC α=20.0 | 0.23 | 0.15 |
| EF0.5% | 20.6 | 0 |
| EF1% | 11.6 | 0 |
| EF5% | 4.5 | 3.4 |
| EF10% | 2.8 | 3.2 |
性能指标是根据Shen等人[17]的数据统计而来。
如表S1所示,在logAUC的数值上并没有体现出MCR比COMT靶标具有更好的早期富集性能,但是ER1%、BEDROC与EF却正确反映了早期富集能力差异。比如,表示早期富集能力的ER1%、BEDROCα=321.9与EF0.5%,MCR比COMT具有更高的值,实际上COMT对应的值均为0。
5.4 数据集下载
ROCS数据集下载:https://pan.baidu.com/s/1B-Vi2IOFeCWtaB8fvi9obw
- dude_rocs.tar.gz:DUDE数据集虚拟筛选结果文件。
- rocs_TanimotoCombo.csv: ROCS TanimotoCombo的性能参数。
- rocs_RefTverskyCombo.csv: ROCS RefTverskyCombo的性能参数。
- glide_shen2021.tar.gz:根据shen等人[17]公开数据统计的GLIDE性能参数。
- gold_shen2021.tar.gz:根据shen等人[17]公开数据统计的GOLD性能参数。
6. 与其它基于配体3D相似性方法比较
最近,Jiang等人22对9种基于配体的3D相似性方法在DUD-E数据集上进行了比较,郑行23等人则公开了采用Uni-Mol深度学习方法进行基于配体3D相似性虚拟筛选的性能,这些数据汇总于表3。
表3. ROCS_TverskyComboScore与其它3D相似性方法在DUD-E数据集上的虚拟筛选性能比较
| 方法 | ROC AUC | EF1% | EF5% | EF10% |
|---|---|---|---|---|
| ROCS_TverskyComboScorea | 0.756 | 25.3 | 8.4 | 5.0 |
| ROCS_ComboScoreb | 0.598 | 12.44 | 4.33 | 2.80 |
| ROCS_ColorScoreb | 0.620 | 13.20 | 4.76 | 3.09 |
| ROCS_ShapeTanimotob | 0.547 | 6.89 | 2.93 | 2.07 |
| Phase Shape_Mmodb | 0.677 | 13.73 | 5.06 | 3.34 |
| Phase Shape_Eleb | 0.674 | 14.57 | 5.50 | 3.57 |
| Phase Shape_Pharmb | 0.692 | 16.03 | 6.00 | 3.84 |
| Shape-itb | 0.541 | 4.73 | 2.50 | 1.91 |
| Align-itb | 0.659 | 12.44 | 4.87 | 3.21 |
| ShaEP_bestb | 0.658 | 10.28 | 4.32 | 2.99 |
| ShaEP_shapeb | 0.625 | 8.86 | 3.82 | 2.76 |
| ShaEP_ESPb | 0.606 | 6.36 | 3.03 | 2.25 |
| SHAFTSb | 0.733 | 19.13 | 6.97 | 4.37 |
| WEGAb | 0.645 | 12.14 | 4.67 | 3.16 |
| LIGSIFTb | 0.718 | 16.22 | 5.79 | 3.78 |
| LS-alignb | 0.699 | 15.88 | 5.74 | 3.66 |
| Uni-Molc | – | 20.40 | 8.48 | 5.59 |
a:本文;b:Jiang等人22;c:郑行23
我们可以发现,采用Tversky( α=0.95)相似性方法的ROCS ComboScore的虚拟筛选总体性能最佳,在DUD-E 102个靶标上的平均ROC AUC=0.756,优于所有的其它方法,如图14所示。
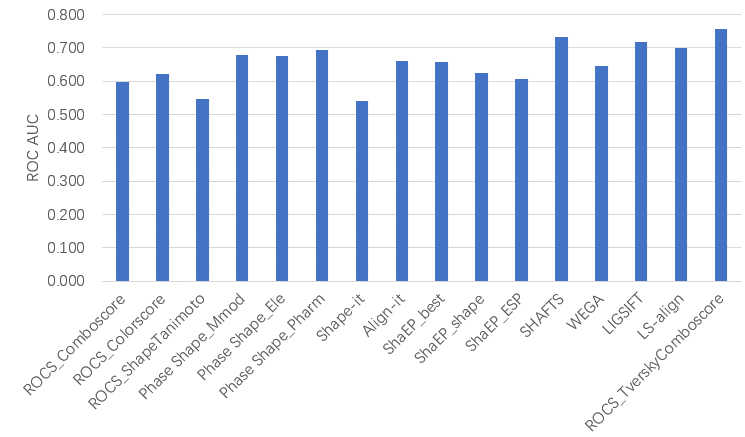
图14. ROCS TverskyComboScore与其它9种3D相似性方法在DUD-E数据集上虚拟筛选总体性能(ROC AUC)比较
同样,采用Tversky( α=0.95)相似性方法的ROCS ComboScore的虚拟筛选方法表现出了最强的早期富集能力。在DUD-E 102个靶标上,富集因子EF1%均值=25.3,优于所有的其它方法,并且大幅领先于第二名Uni-Mol(EF1%=20)与第三名SHAFTS(EF1%=19.13),如图15所示。
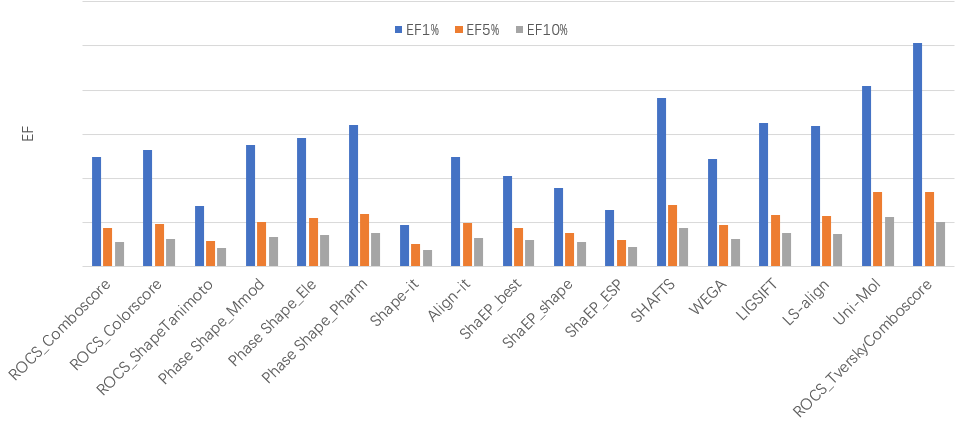
图15. ROCS TverskyComboScore与其它9种3D相似性方法在DUD-E数据集上虚拟筛选富集因子EF(Top 1%,5%,10%)的比较
总的来说,采用Tversky相似性方法的ROCS ComboScore不仅在总体虚拟筛选性能优于其它基于配体3D相似性方法与深度学习方法,而且在早期富集能力上也优于其它方法。
7. 文献
- ROCS. https://www.eyesopen.com/rocs
- Grant, J. A.; Gallardo, M. A.; Pickup, B. T. A Fast Method of Molecular Shape Comparison. J. Comput. Chem. 1996, 17 (14), 1653–1666.
- Hawkins, P. C. D.; Skillman, A. G.; Nicholls, A. Comparison of Shape-Matching and Docking as Virtual Screening Tools. J. Med. Chem. 2007, 50 (1), 74–82. https://doi.org/10.1021/jm0603365.
- Venhorst, J.; Núñez, S.; Terpstra, J. W.; Kruse, C. G. Assessment of Scaffold Hopping Efficiency by Use of Molecular Interaction Fingerprints. J. Med. Chem. 2008, 51 (11), 3222–3229. https://doi.org/10.1021/jm8001058.
- Sheridan, R. P.; McGaughey, G. B.; Cornell, W. D. Multiple Protein Structures and Multiple Ligands: Effects on the Apparent Goodness of Virtual Screening Results. J. Comput. Aided. Mol. Des. 2008, 22 (3), 257–265. https://doi.org/10.1007/s10822-008-9168-9.
- Rush, T. S.; Grant, J. A.; Mosyak, L.; Nicholls, A. A Shape-Based 3-D Scaffold Hopping Method and Its Application to a Bacterial Protein−Protein Interaction. J. Med. Chem. 2005, 48 (5), 1489–1495. https://doi.org/10.1021/jm040163o.
- Koes, D. R.; Camacho, C. J. Shape-Based Virtual Screening with Volumetric Aligned Molecular Shapes. J. Comput. Chem. 2014, 35 (25), 1824–1834. https://doi.org/10.1002/jcc.23690.
- DUDE. http://dude.docking.org
- McGaughey, G. B.; Sheridan, R. P.; Bayly, C. I.; Culberson, J. C.; Kreatsoulas, C.; Lindsley, S.; Maiorov, V.; Truchon, J.-F. F.; Cornell, W. D. Comparison of Topological, Shape, and Docking Methods in Virtual Screening. J. Chem. Inf. Model. 2007, 47 (4), 1504–1519. https://doi.org/10.1021/ci700052x.
- OMEGA. https://www.eyesopen.com/omega
- SPRUCE. https://www.eyesopen.com/spruce
- 肖高铿. 如何进行虚拟筛选的方法学验证. 墨灵格的博客. 2016-09-22. http://blog.molcalx.com.cn/2016/09/22/virtual-screening-methodology-validation.html
- Cleves, A. E.; Johnson, S. R.; Jain, A. N. Electrostatic-Field and Surface-Shape Similarity for Virtual Screening and Pose Prediction. J. Comput. Aided. Mol. Des. 2019, 33 (10), 865–886. https://doi.org/10.1007/s10822-019-00236-6.
- Puertas-Martín, S.; Redondo, J. L.; Ortigosa, P. M.; Pérez-Sánchez, H. OptiPharm: An Evolutionary Algorithm to Compare Shape Similarity. Sci. Rep. 2019, 9 (1), 1–24. https://doi.org/10.1038/s41598-018-37908-6.
- Izhar, W.; Michael, D.; Abraham, H. AtomNet: A Deep Convolutional Neural Network for Bioactivity Prediction in Structure-Based Drug Discovery. arXiv:1510.02855 2015.
- Mysinger, M. M.; Carchia, M.; Irwin, J. J.; Shoichet, B. K. Directory of Useful Decoys, Enhanced (DUD-E): Better Ligands and Decoys for Better Benchmarking. J. Med. Chem. 2012, 55 (14), 6582–6594. https://doi.org/10.1021/jm300687e.
- Shen, C.; Hu, Y.; Wang, Z.; Zhang, X.; Pang, J.; Wang, G.; Zhong, H.; Xu, L.; Cao, D.; Hou, T. Beware of the Generic Machine Learning-Based Scoring Functions in Structure-Based Virtual Screening. Brief. Bioinform. 2021, 22 (3), 1–22. https://doi.org/10.1093/bib/bbaa070.
- Chaput, L.; Martinez-Sanz, J.; Saettel, N.; Mouawad, L. Benchmark of Four Popular Virtual Screening Programs: Construction of the Active/Decoy Dataset Remains a Major Determinant of Measured Performance. J. Cheminform. 2016, 8 (1), 56. https://doi.org/10.1186/s13321-016-0167-x.
- Wang, D.; Cui, C.; Ding, X.; Xiong, Z.; Zheng, M.; Luo, X.; Jiang, H.; Chen, K. Improving the Virtual Screening Ability of Target-Specific Scoring Functions Using Deep Learning Methods. Front. Pharmacol. 2019, 10 (August), 1–11. https://doi.org/10.3389/fphar.2019.00924.
- Svensson, F.; Karlén, A.; Sköld, C. Virtual Screening Data Fusion Using Both Structure- and Ligand-Based Methods. J. Chem. Inf. Model. 2012, 52 (1), 225–232. https://doi.org/10.1021/ci2004835.
- Brickelmaier, M.; Lugovskoy, A.; Kartikeyan, R.; Reviriego-Mendoza, M. M.; Allaire, N.; Simon, K.; Frisque, R. J.; Gorelik, L. Identification and Characterization of Mefloquine Efficacy against JC Virus In Vitro. Antimicrob. Agents Chemother. 2009, 53 (5), 1840–1849. https://doi.org/10.1128/AAC.01614-08.
- Jiang, Z.; Xu, J.; Yan, A.; Wang, L. A Comprehensive Comparative Assessment of 3D Molecular Similarity Tools in Ligand-Based Virtual Screening. Brief. Bioinform. 2021, 22 (6), 1–17. https://doi.org/10.1093/bib/bbab231.
- 郑行. 分子3D预训练模型Uni-Mol在药物研发中的应用|CADD领袖论坛第3期. https://www.bilibili.com/video/BV1Wd4y137q1

![在DUDE数据集的102个靶标上,Koes等人<sup>[7]</sup>计算的与本文计算的AUC比较](http://blog.molcalx.com.cn/wp-content/uploads/2022/03/2022040307254456.png)