摘要:本文以SARS-CoV-2的3CL Mpro非共价抑制剂为例,演示了如何协同使用Flare FEP与Spark以加速新的非共价抑制剂的识别。具体地讲,先用一组活性已知的3CL Mpro非共价抑制剂进行Benchmark模式FEP计算,验证方法的可靠性;再用Spark生成新的化合物;最后在产品模式下用FEP预测新化合物的活性,从中发现预测可靠且具有高结合亲和力预测值的新化合物。
原文:https://www.cresset-group.com/media/uploads/files/COVID-Mpro_FEP_bioisost_repl_FINAL_Hqx0oIb.pdf
作者:Sofia Bariami, Giorgia Zaetta, Mark Mackey
单位:Cresset, Cambridgeshire, UK
前言
SARS-CoV-2冠状病毒利用各种酶进行高效地复制。两种半胱氨酸蛋白酶,即3CLpro/Mpro和PLpro负责剪切多种蛋白以产生构建新病毒颗粒所需的病毒蛋白。这些蛋白已被确定为发现治疗药的潜在靶点,在药物发现领域引起了极大的关注,重点是了解它们的结构生物学并开发抑制剂。
Flare™ FEP是Cresset开发的可靠的结合亲和力预测工具,在本文中用于研究一组实验活性已知的SARS-CoV-2冠状病毒抑制剂数据集1-4。我们旨在阐明这些化合物对Mpro的结合亲和力,并确定对活性有益的关键相互作用。
方法
Flare FEP通过将水溶液中的蛋白-配体复合物中的结合型配体与非结合型配体的中间态进行突变来获得相对结合自由能(ΔΔG)。主要参数如下:
- 小分子力场:OpenFF 2.0.0
- 蛋白力场: AMBER FF14SB
- 电荷计算方法: AM1-BCC
- 溶剂: Explicit TIP3P water
- 初始每λ窗口模拟时长: 4ns
图1. 用户友好的Flare界面使得FEP计算与故障排除非常便利
数据集
选择了一系列蛋白晶体结构与共晶配体进行了评估,选择的依据是配体结构相似性。
Table 1.与本研究相关的晶体学数据信息,红框表示我们计算的起点。
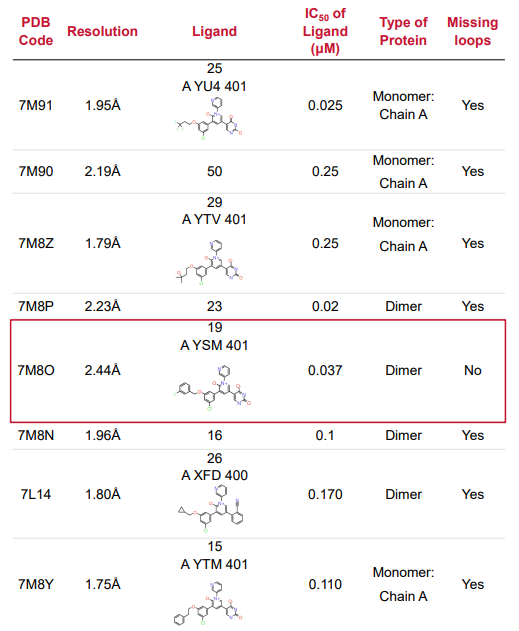
最后选择具有共晶配体A YSM 401的PDB 7M8O作为起点:
- 没有缺失的loop
- 检测到单体与二聚体分子动力学模拟稳定
蛋白-配体相互作用
如图2所示,共晶配体以吡啶酮环为中心呈三叶草形:
- Pyridinyl: Pocket S1
- Uracil: Pocket S1’
- Phenyl: Pocket S2
在本研究中,我们将探索朝向S4子口袋取代基的贡献。
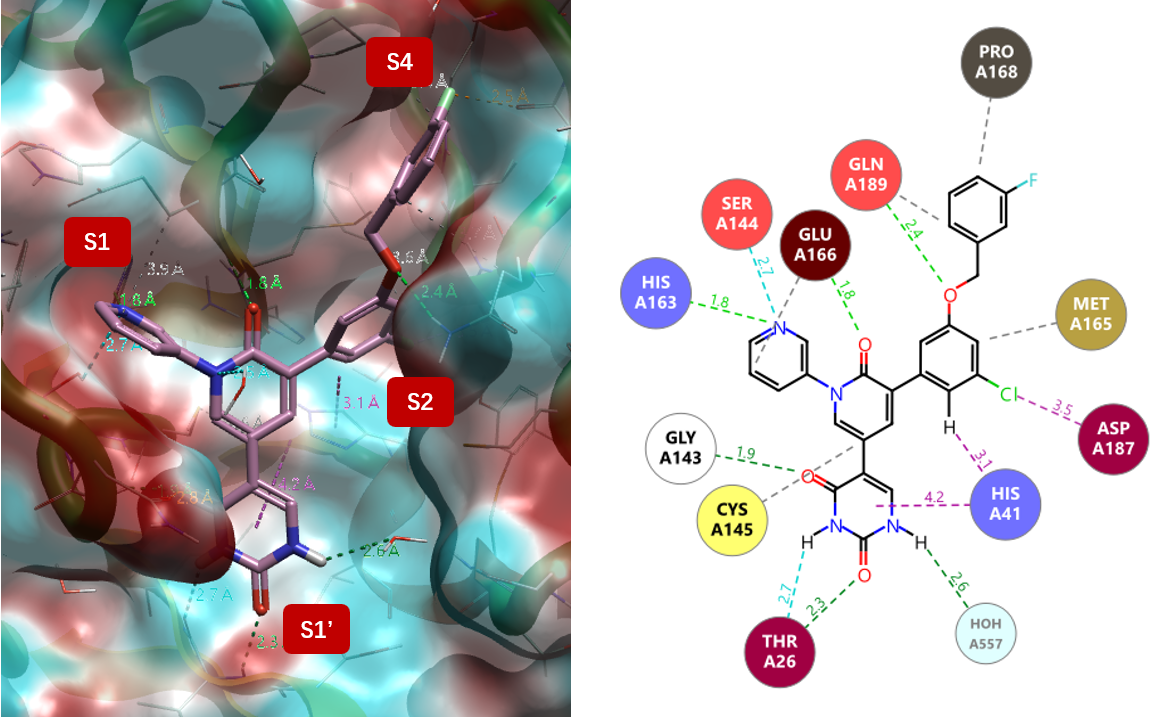
图2. 左:共晶配体伸向4个子口袋。右:蛋白-配体相互作用。紫色:芳香边对面;淡紫色:卤键;淡绿色/深绿色:强氢键;淡蓝色:弱氢键。
基准模式FEP计算
对54个有文献报道IC50的化合物进行评估以选出适合用Flare进行FEP计算的数据集。结果发现有36个衍生物适合用来生成数据集用于FEP计算,组成如下:
- 尿嘧啶衍生物(28)
- 五元环(8)
使用Flare的Conformation Hunt and Alignment方法将36个化合物叠合到PDB 7M80的共晶配体A YSM 401上。
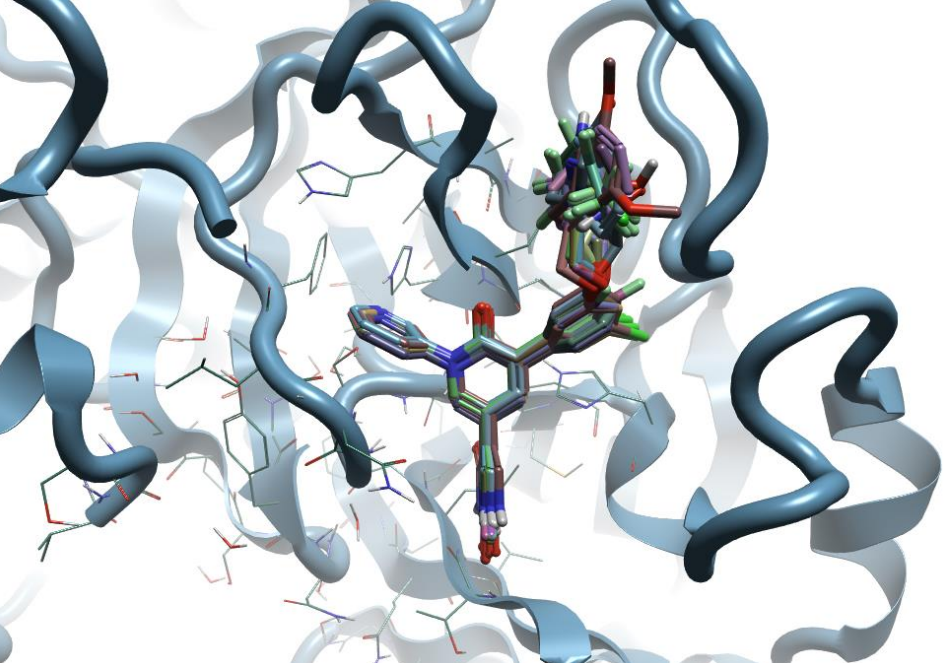
图3. 衍生物必须在结合口袋中叠合好,这可以用Flare的最大公共子结构 (MCS)叠合方轻松实现。
为基准FEP计算生成了一个良好连接的微扰网络,其中包含三个中间分子。如图4所示,FEP微扰图是用LOMAP5生成的。
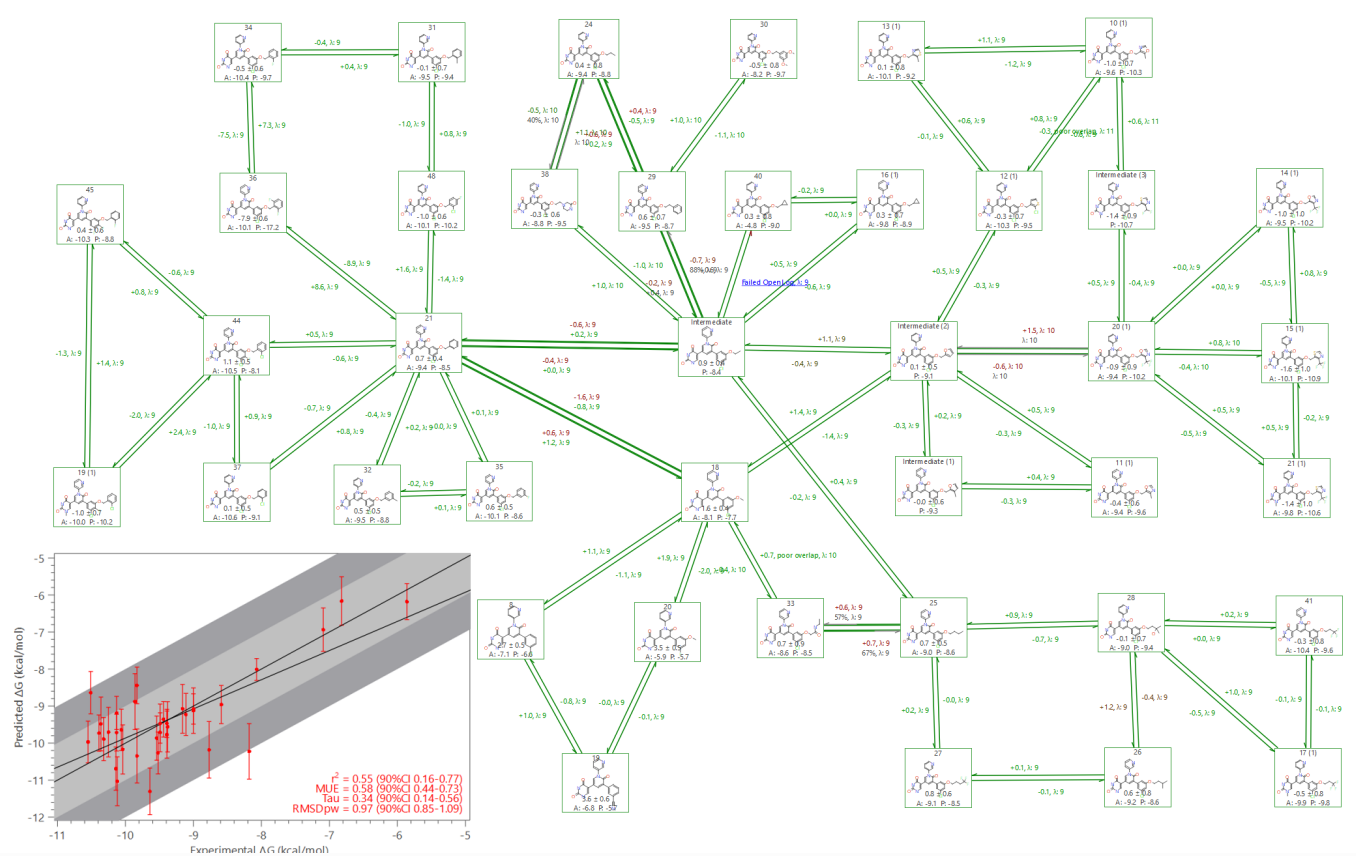
图4. 基准数据集的FEP微扰图。左下的活性图表明实验值与预测值之间具有良好的相关性。
理想情况下,配体之间的计算ΔΔG应该与实验ΔG值的差值相似,并且计算的ΔG值应与实验确定的ΔG值相差不超过1kcal/mol。在本算例中,我们发现实验数据与计算值之间具有良好的相关性,并且误差低于1kcal/mol的科学共识:
- R2 = 0.55
- Tau = 0.34
- MUE = 0.58
- RMSD = 0.97
生物等排体替换
Cresset的生物等排体替换软件Spark™用于生成新的设计,然后用于FEP产品模式计算。Spark使用3D静电和形状特性来探索给定骨架上的R基团。用3D相似性打分对结果进行优先级排序,并可选地使用蛋白作为排除体积。
本次实验采用化合物21作为起点:
- 具有中等活性,IC50 = 120 nM
- 对苄基片段进行从头设计设计的探索
图5. 化合物21的SPARK生物等排体R-基团替换实验结果
Spark总共生成了500个化合物,从中选择了27个用于产品模式的FEP计算。这27个化合物选择的标准是:形式电荷为零,与化合物21的2D相似性低、具有均衡的物理化学性质以及对FEP研究有意义的化学修饰。
产品模式FEP计算
用27个新化合物和4个已知活性的化合物进行了生产模式的FEP计算。从生产模式FEP计算结果来看,预测到13个化合物比参比化合物21(ΔG小于9.4 kcal/mol)更具活性(结合自由能高10倍),并且与整个数据集中活性最强衍生物(化合物37)具有相似的活性(ΔG=10.6 kcal/mol)。
图6. FEP production模式计算,其中27个化合物是Spark生成的,4个化合物来自基准测试集(蓝色链接),另有4个中间体分子
观察到两个有意思的现象:
- 与O连接臂相比,氨基连接臂具有良好的耐受性
- S4口袋优选卤素取代的苯基
结论
- Flare FEP准确地预测了小分子与Mpro的结合亲和力,可用于新设计分子的结合研究
- Spark可以设计出高预测活性值的新分子
- Cresset工具协同应用是加速新药发现的一种很有前途的方法
文献
- Chun-Hui Zhang, et al., ACS Cent. Sci. 2021, 7, 467−475
- Chun-Hui Zhang, et al., ACS Med. Chem. Lett. 2021, 12, 1325−1332
- Maya G. Deshmukh, et al., Structure 29, 823–833, August 5, 2021
- William L. Jorgensen, WO 2022/150584 A1
- David L. Mobley, et al., J. Comput. Aided Mol. Des., 2013, 27, 9, 755-770





