摘要:鉴于计算方法在活性预测方面相比实验室方法的效率优势日益凸显,加之由于计算机硬件的进步,高性能结构赋能方法的可及性不断提高,我们认为,ABFE计算将很快成为计算机虚拟筛选工作流程中标准优先级排序方法的一部分。在本文中,我们使用ABFE对基于片段的生物等排体替换实验结果进行了优先级排序。Spark结合Cresset独特的场和形状打分方法,能够以最小的计算成本生成一组结构多样的生物等排体。通过ABFE,我们发现在Spark生成的分子中约有70%预测的pIC50值达到6以上,达到了对靶标强结合活性的阈值。
Thomas Potter et al. (2025) Available at: https://www.cresset-group.com.
前言
生物等排体替换实验是虚拟筛选实验中的一种极具价值的工具,它能够在保留参比配体重要特性的同时快速生成新的分子。Spark是一款领先的计算机模拟工具,用于快速、高效地进行片段生物等排体替换实验,这得益于其直观的用户界面。Spark利用电荷和形状相似性(这对蛋白质-配体结合至关重要)来生成分子并进行排序。
绝对结合自由能(Absolute binding free energy,ABFE)计算在药物发现领域中越来越受欢迎,它是一种精确定确结合亲和力的方法,并且在对诸如Spark之类方法生成的结果进行优先级排序时非常有用。在本文中,我们对ABFE计算预测BTK靶点已知结合自由能的能力进行了基准测试。然后,我们证明了该方法可以用于根据结合亲和力对Spark生成的分子进行排序。最后,我们利用分子动力学(MD)模拟获得了这些分子结合行为的详细见解。
用SPARK进行生物等排体替换
Spark是一款用于片段生物等排体替换的桌面应用程序。它能够执行多种实验,包括骨架跃迁和R-基团替换。Spark是Cresset的旗舰计算机辅助药物设计(CADD)建模套件Flare中的一个模块。
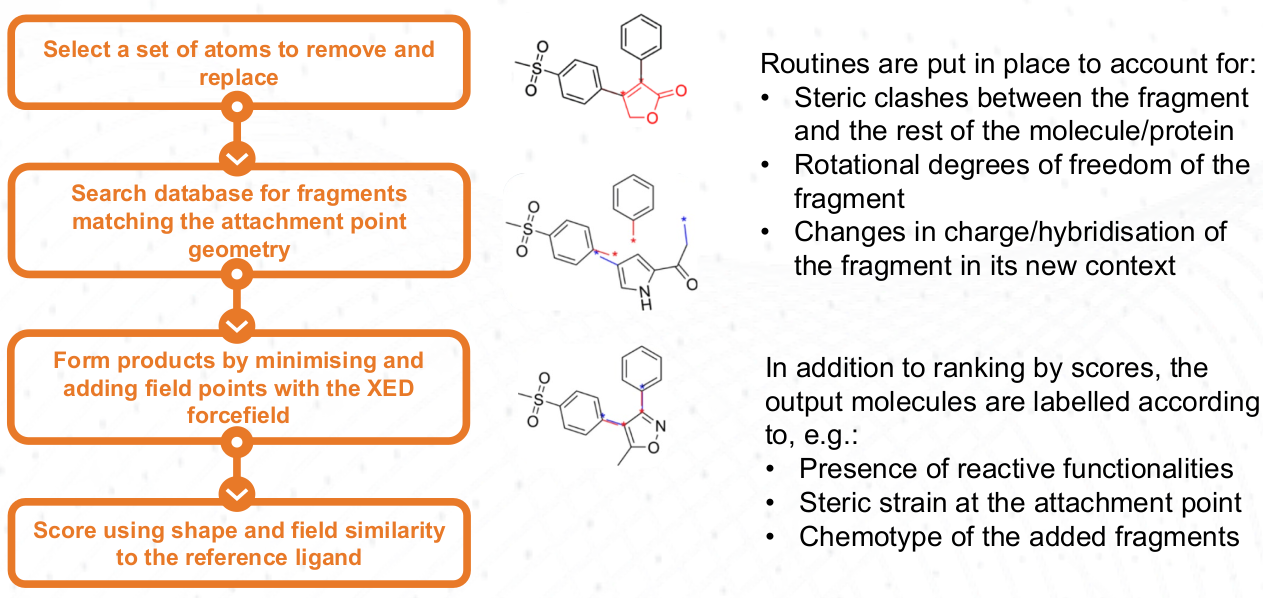
图1. Spark的骨架跃迁工作流
在Spark中,分子的打分是通过场点1来进行的,这些场点表示使用XED力场计算出的静电场的正极值(红色)和负极值(蓝色),以及范德华表面(黄色)和疏水表面(橙色)。这些场点被用来计算每个试验分子与输入的参比配体的相似性。关键的是,打分是基于整个分子的静电特性计算的,而不仅仅是测试片段。
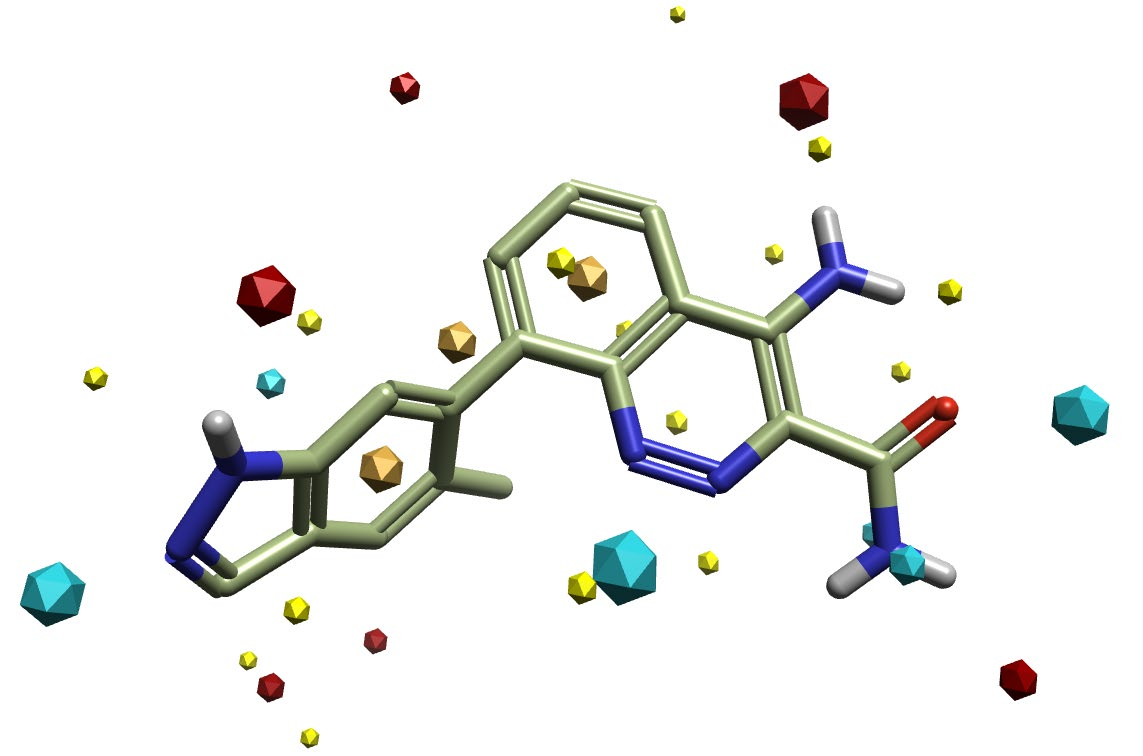
图2. 在Spark计算中用来打分的场点
Flare : 绝对结合自由能(Absolute binding free energy ,ABFE)计算
ABFE(绝对结合自由能)是一种炼金术自由能方法,作为Flare FEP2的一部分,通过分子动力学直接计算配体的结合自由能。与相对结合自由能(relative binding free energy,RBFE)计算不同,ABFE不需要一系列同系物配体。该计算涉及一组模拟,在自由状态和结合状态中逐步关闭相互作用。Flare的实现使用Boresch约束来确保配体在整个过程中保持在结合位点内。
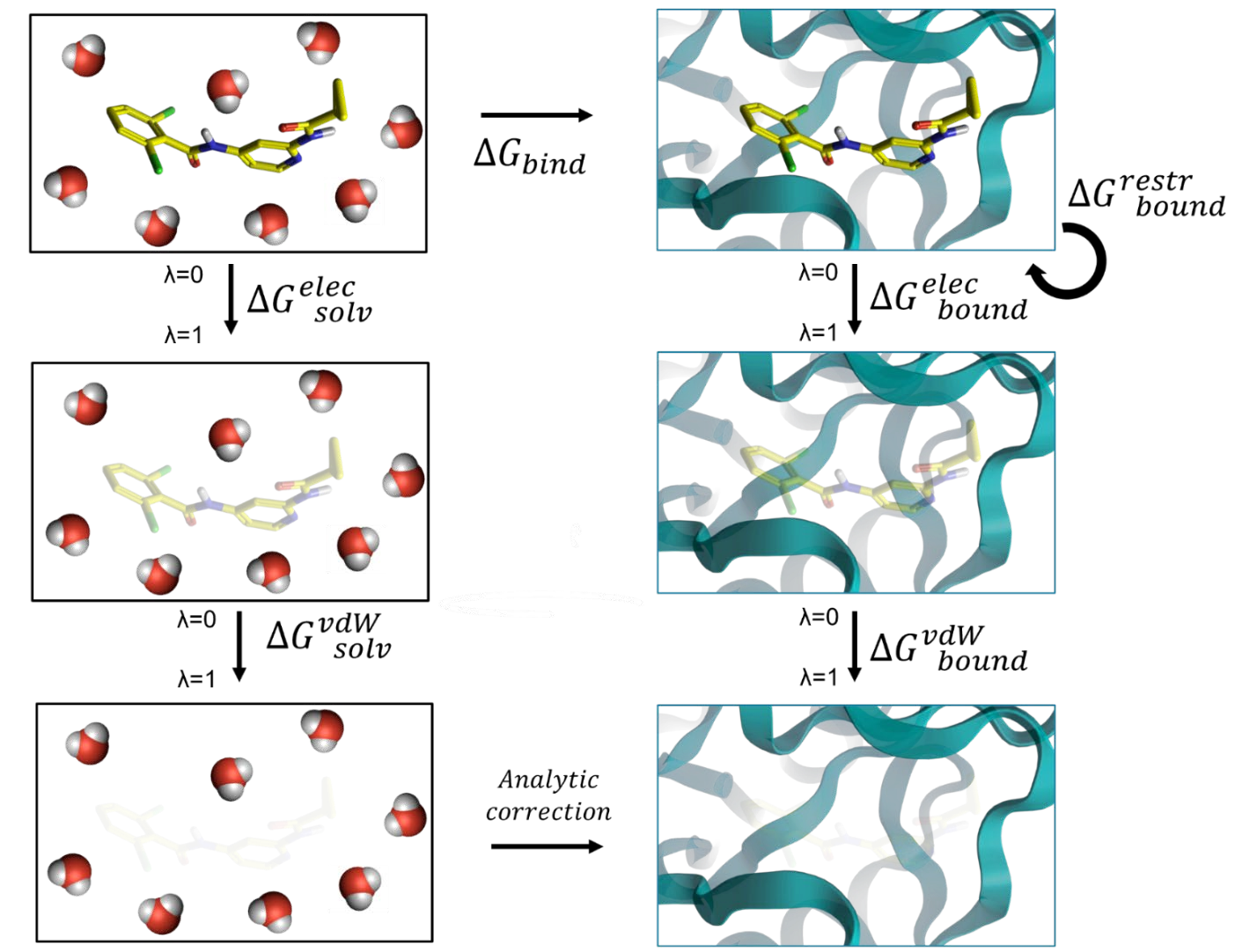
图3. ABFE计算的热力学循环
我们进行了基准测试,以确定Flare ABFE方法在布鲁顿酪氨酸激酶(BTK)靶点上的适用性。pIC50阈值设为6.0,并在调整结果以考虑平均 ABFE偏移后,ABFE能够准确对18个已知活性的配体进行分类:
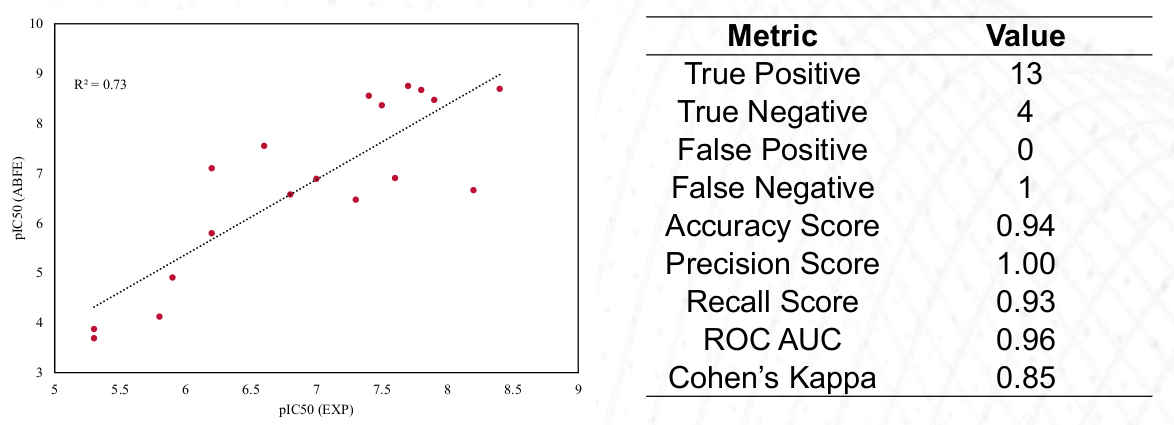
图4. 在BTK数据集上ABFE基准测试的相关性图(左)和分类性能指标(右)
Spark: R基团替换实验
对图5所示的BTK抑制剂进行了Spark R基团替换实验,靶标和参比配体结构取自PDB 4Z3V3:
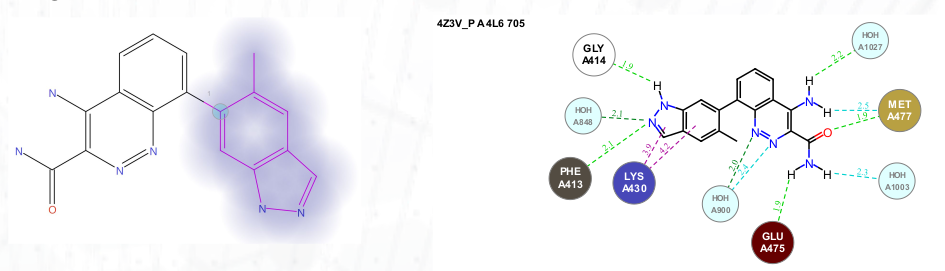
图5. Spark参比分子及其R基团替换区域(左),以及显示关键结合相互作用的相互作用图(右)
用经过训练的QSAR模型对排名前1000的Spark分子进行了排序,该模型用于预测化合物对BTK的结合活性。选取预测活性最高的30个分子进行进一步的ABFE和分子动力学分析。
Flare:更多ABFE和Spark结果分析
ABFE用于计算Spark结果中前30个分子的活性。大多数(70%)分子的pIC50 \(>\) 6.0,表明其具有强结合能力。
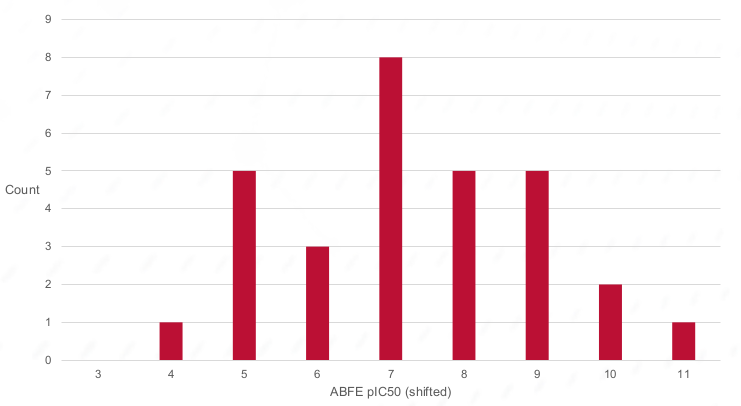
图6. 在Spark结果中ABFE计算靠前的活性分布
通过GCNCMC方法4进行的分子动力学模拟显示,排名高和低的配体在相互作用和行为上存在显著差异。排名最高的配体形成了强烈的氢键相互作用,与参比分子类似,而排名第二低的配体则部分被挤出结合位点。
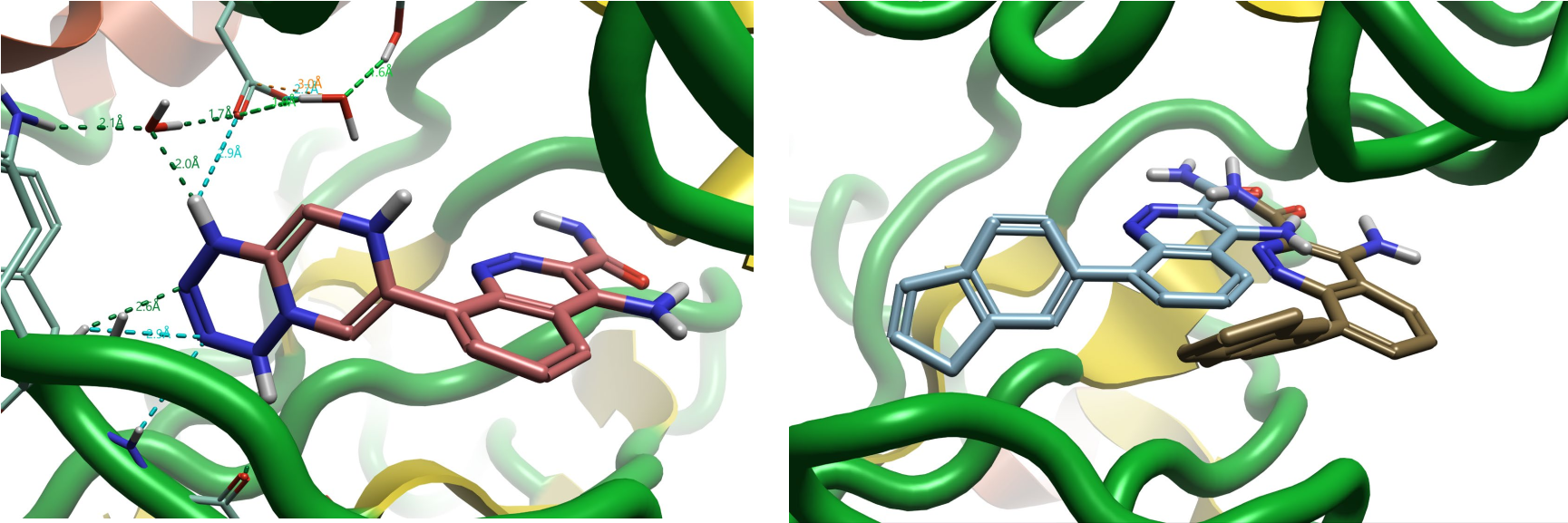
图7. 排名最高(左)和排名第二低(右)配体的分子动力学模拟快照
结论
鉴于计算方法在活性预测方面相比实验室方法的效率优势日益凸显,加之由于计算机硬件的进步,高性能结构赋能方法的可及性不断提高,我们认为,ABFE计算将很快成为计算机虚拟筛选工作流程中标准优先级排序方法的一部分。
在本文中,我们使用ABFE对基于片段的生物等排体替换实验结果进行了优先级排序。Spark结合Cresset独特的场和形状打分方法,能够以最小的计算成本生成一组结构多样的生物等排体。通过ABFE,我们发现在Spark生成的分子中约有70%预测的pIC50值达到6以上,达到了对靶标强结合活性的阈值。
文献
- Cheeseright, T. et al. (2006) “Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation,” Journal of Chemical Information and Modeling, 46(2), pp. 665–676. Available at: https://doi.org/10.1021/ci050357s.
- Kuhn, M. et al. (2020) “Assessment of Binding Affinity via Alchemical Free-Energy Calculations,” Journal of Chemical Information and Modeling, 60(6), pp. 3120–3130. Available at: https://doi.org/10.1021/acs.jcim.0c00165.
- Smith, C.R. et al. (2015) “Fragment-Based Discovery of a Small Molecule Inhibitor of Bruton’s Tyrosine Kinase,” Journal of Medicinal Chemistry, 58(14), pp. 5437–5444. Available at: https://doi.org/10.1021/acs.jmedchem.5b00734.
- Melling, O.J. et al. (2023) “Enhanced Grand Canonical Sampling of Occluded Water Sites Using Nonequilibrium Candidate Monte Carlo,” Journal of Chemical Theory and Computation, 19(3). Available at: https://doi.org/10.1021/acs.jctc.2c00823.
联系我们
Flare V10是Flare最新版本,交付先进的科学方法、分析工具和直观、易用的增强功能,洞察您的配体 – 蛋白质复合物结构。
想要尝试Flare信息丰富、用户友好界面,发现它如何帮助您自信地推动潜在先导化合物优化?请现在就联系我们安排试用,快速访问Flare的广泛功能。我们的专业团队随时准备通过安装和设置为您提供支持,而我们全面的教程库——涵盖从常见工作流程到高级方法和功能的所有内容将帮助您开始使用。我们在这里帮助您更快地实现目标,让您设计出感兴趣的分子。
电邮:info@molcalx.com
电话:020 – 38261356



