摘要:如果一个分子具有带电原子,那么从真空DFT计算获得的MESP将完全被该电荷主导,这使得计算的MESP不能于指导分子设计的目的。在本文中,以一个带正电荷的大环PDGFRα抑制剂L2为例,演示如何用设计中性模型分子,然后用Flare QM进行电子密度与ESP计算,最后通过基于QM-场点的可视化验证了蛋白与配体之间发生非经典的Glu675/Cys677-C=O···H—C氢键相互作用的假设。
肖高铿/2025-04-14
前言
根据Derewenda等人统计1,到2020年为止,FDA大约批准了48个激酶抑制剂用于治疗各种疾病。对其中29个激酶抑制剂与其靶标的共晶结构进行了系统分析,结果发现在抑制剂与铰链羰基之间广泛存在着C─H···O=C氢键相互作用。此外,作者还注意到在FDA批准的激酶抑制剂中发现的骨架类型非常有限,在利用激酶铰链区内的羰基上是一致的:不仅作为典型氢键的受体,而且还是弱氢键的受体,其中C-H基团通常来自芳环或与电负性氮原子相邻。
鉴于这种C─H···O=C氢键相互作用的重要性,此前介绍了如何利用Flare的XED力场来定性分析激酶抑制剂的此类相互作用2。在另外一个例子里3,还介绍了如何用Flare的场技术来定量分析Tyk2抑制剂的SAR,并用来对不同铰链区结合片段进行优先级排序,与QM方法达到了同样的骨架优选效果。在Tyk2铰链结合片段优选的算例里3,还用Flare QM4计算了配体的分子表面静电势(MESP),创造性地从蛋白“视角”观察配体的MESP,这使得可以定性、定量地分析激酶铰链区与配体之间的C─H···O=C氢键相互作用以及静电互补性。
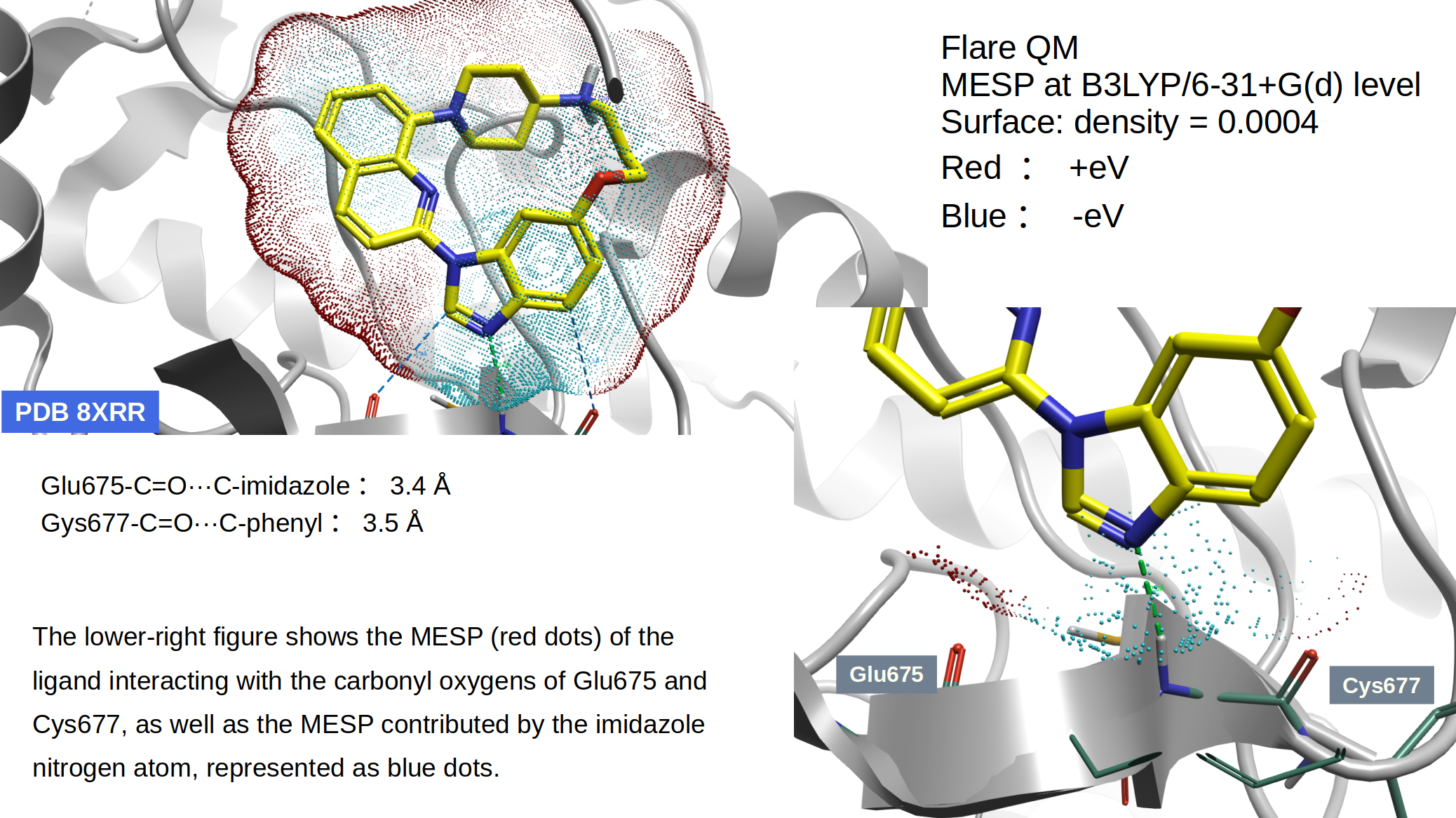
最近Liang等人5公开了一个大环PDGFRα抑制剂L2的共晶结构(PDB 8XRR),如图1所示,L2的咪唑氮原子与Cys677酰胺-NH发生经典的氢键相互作用,此外咪唑环与苯环的C─H也铰链区Glu675与Cys的羰基氧也发生非经典的C─H···O=C氢键相互作用。
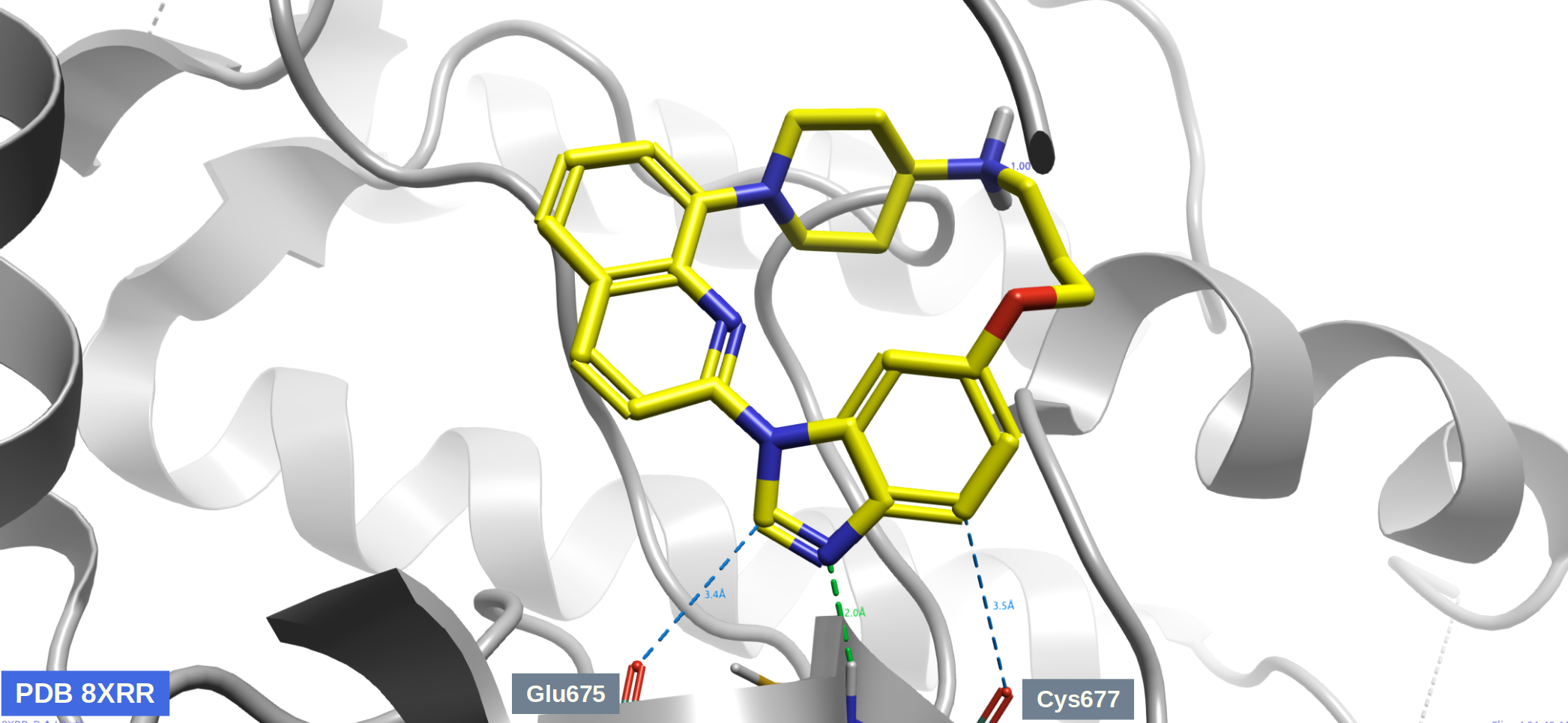
图1. 大环抑制剂L2与PDGFRα的共晶结构(PDB 8XRR)。黄色配体:L2;飘带:PDGFRα;图片用Flare V10生成。
本文的主要目的是演示如何用Flare QM来计算分析PDGFRα铰链区Glu675与Cys677-C=O与L2之间的静电相互作用,特别是从Glu675与Cys677-C=O视角分析配体的MESP。
方法与结果
计算方法与Tyk2算例3中的Flare QM分析部分一致,简述如下:
- 几何优化:在xTB理论水平进行位置约束的几何优化,然后叠合到共晶配体上
- 电子密度:在B3LYP/6-31+G(d)理论水平单点能计算
- ESP计算:在B3LYP/6-31+G(d)理论水平单点能计算
- MESP计算:将diensity = 0.0004的点定义为分子表面,并给出该点的静电势(EPS)
- MESP可视化:将MESP作为XED场点(QM-based filed point),在Flare里可视化
众所周知,如果一个分子具有带电原子,那么从真空DFT计算获得的ESP表面将完全被该电荷主导。这不是DFT计算的错误,这是ESP表面的精确表示。但是这种单调的ESP分子表面缺乏区分特征,意味着不能用来精确解释配体-蛋白相互作用;也不能用来精确地叠合、比较两个分子的静电相似性。与Tyk2算例的中性模型分子所不同的是,L2的净形式电荷为+1。因此,有必要设计一个中性的模型分子来代替L2进行MESP计算。
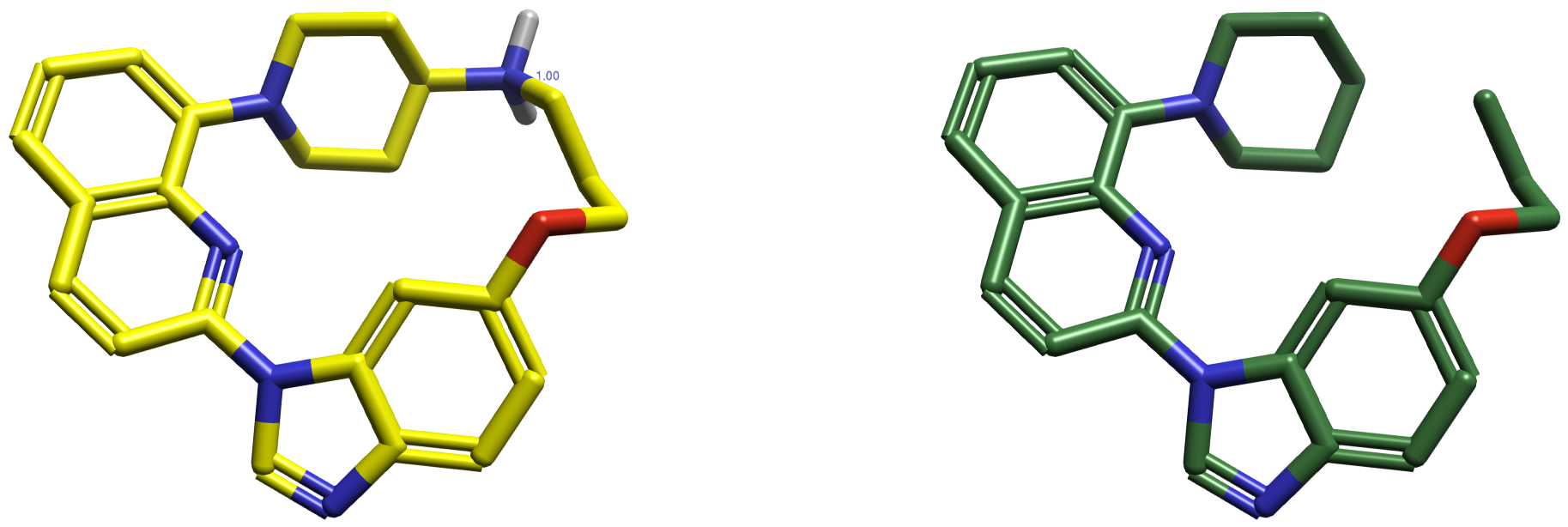
图2. PDGFRα大环抑制剂L2(左)与用来计算MESP的中心模型分子L2’(右)
有多种模型分子可供选择,比如将氮原子中性化处理,或者仅保留最小的铰链结合区结构。在这里,我选择将L2(图2-左)的正电荷氮原子删除后的开环分子作为计算电荷的模型分子L2’(图2-右)。
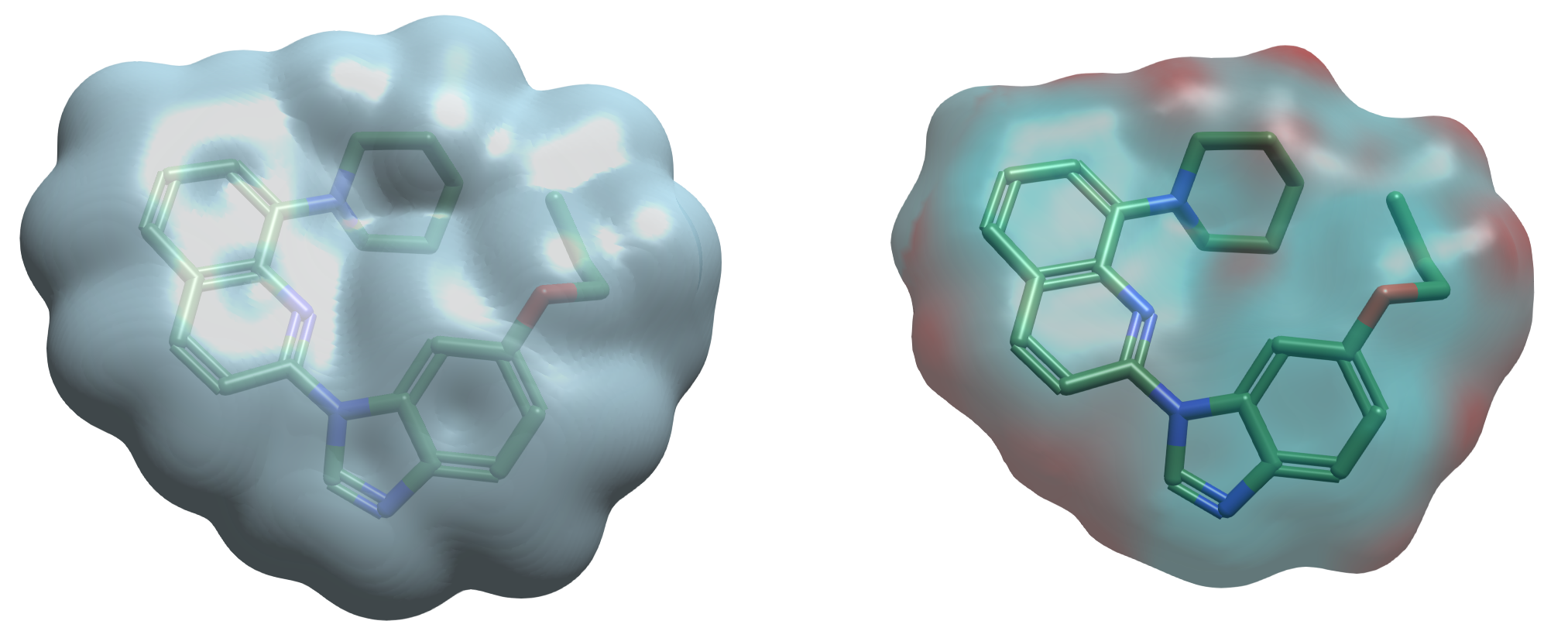
图3. 在B3LYP/6-31+G(d)理论水平计算的模型分子L2‘的分子表面(density=0.0004)与MESP
将PDB 8XRR下载到Flare里,用Protein Prep进行结构准备,然后将L2提取到配体表单(Ligand Table),同时用Flare Edit将一个L2副本编辑为L2‘。在xTB理论水平对模型分子L2‘进行位置约束的几何优化,然后使用Flare| 3D Pose | Superpose将优化后的L2‘叠合到共晶配体L2上。最后在用Flare QM在B3LYP/6-31+G(d)理论水平进行单点计算,得到电子密度=0.0004分子表面与MESP,结果如图3所示。
在Flare | Surfaces里,将计算的DFT电子密度(density)与EPS格点导出为dx格式文件,分别保存为L2_den.dx与L2_esp.dx。
在这里我用pyflare脚本,将电子密度=0.0004格点上静电势导出为csv格式:
1 2 3 | flare_qm_mesp.py --den L2_den.dx \ --esp L2_esp.dx \ --output L2_mesp.csv |
然后,将MESP不为0的分子表面作为场点追加到SDF文件:
1 2 3 | flare_qm_field_point_to_sdf.py \ --isdf L2_xtal.sdf \ --osdf L2_mesp.sdf L2_mesp.csv |
现在,已经将计算的QM场点追加到SDF文件L2_mesp.sdf里。然后将该SDF读入Flare,通过Flare | home | Field来激活3D可视化,结果如图4所示。
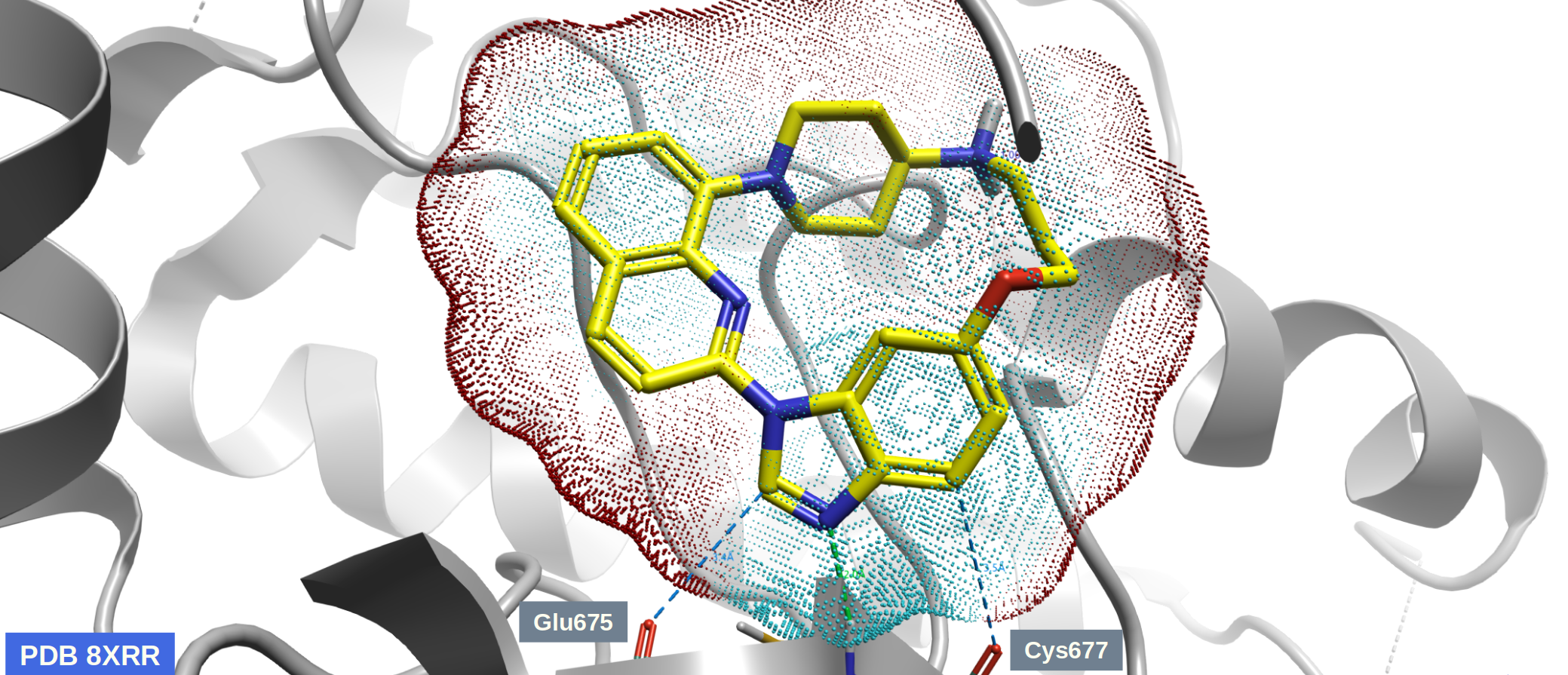
图4. 在B3LYP/6-31+G(d)理论水平计算的模型分子L2‘的MESP。灰色飘带:PDGFRα;黄色棍棒:L2;蓝色场点:负ESP;红色场点:正ESP;场点大小:点越大则EPS绝对值越大。
从图4可以看到,铰链区Glu675与Cys677-C=O附近的配体QM场点为红色,这意味羰基氧与配体的在空间上静电上互补;铰链区Cys677-NH与配体的蓝色QM场点重合,这与Cys677-NH···N-咪唑之间的经典氢键相互作用一致。
聚焦于铰链区Glu675与Cys677-C=O能够“摸”到的配体场点可以让我们进一步了解C-H···O=C之间的相互作用。因此有必要从Glu675与Cys677-C=O视角考察看到的配体场点,这可通过可视化羰基氧原子半径范围内的配体表面ESP来实现。这里以Glu675-C=O为例,用一个pyflare脚本将碳基氧原子半径范围内的配体场点提取出来,附加到SDF里:
1 2 3 4 5 6 | flare_qm_add_near_point_to_SDF.py --atom O \ --xyz 25.361 -1.781 -11.240 \ --den L2_den.dx \ --esp L2_esp.dx \ --isdf L2_xtal.sdf \ --osdf L2_Glu675_CO_mesp.sdf |
这里我用bondi原子半径来计算接触的距离(d),半径参考了Manjeera(2019)等人的文章并会汇编为字典,通过atom选项告诉脚本查寻原子半径,并计算距离(d):
$$
d = r_{atom} + 0.5
$$
其中xyz选项是Glu675-C=O的氧原子的坐标,并将配体表面上距离该氧原子d范围内的QM场点追加到SDF文件。同理,还可以将Cys677-C=O可视范围的配体场点,以及配体咪唑环上与Cys酰胺NH发生氢键相互作用的氮贡献的配体场追加到SDF。然后读入Flare,在3D视窗进行可视化分析,结果如图5所示。
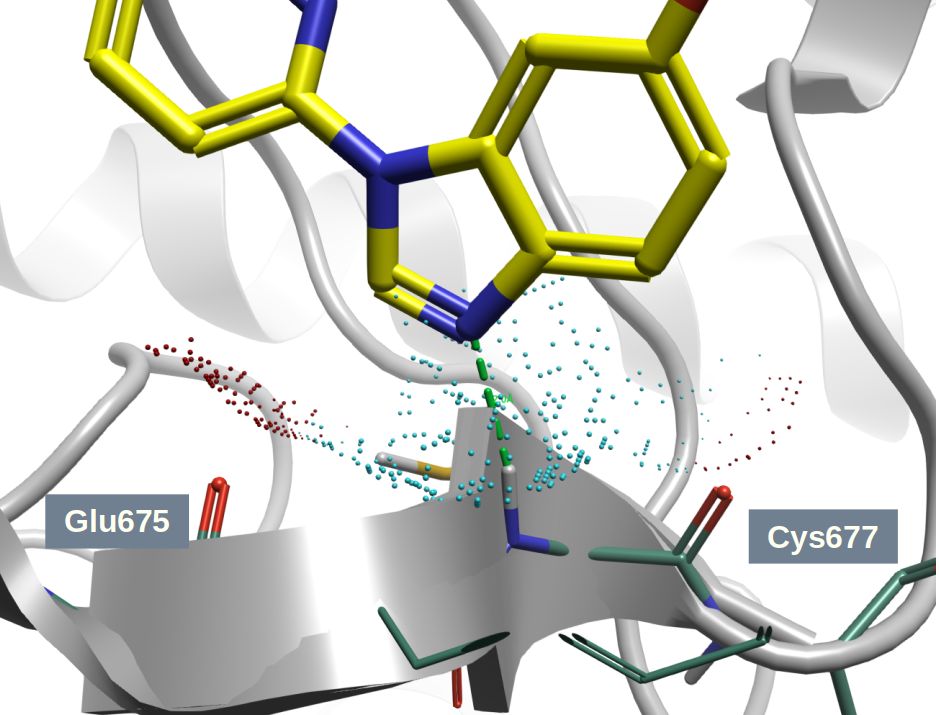
图5. 从铰链区Glu675与Cys677-C=O以及Cys677酰胺-NH视角出发“看到”的配体QM场点。灰色飘带:PDGFRα;黄色棍棒:L2;蓝色场点:负ESP;红色场点:正ESP;场点大小:点越大则EPS绝对值越大。
图5展示了铰链区Glu675/Cys677-C=O、Cys677酰胺NH能够“触摸到/看到”配体的形状与静电。可以清晰地发现:Glu675与Cys677-C=O与配体的红色场点靠近,而Cys677酰胺-NH与配体的蓝色场点靠近。这与我们假设在蛋白与配体之间发生非经典的Glu675/Cys677-C=O···H—C氢键相互作用是一致的。
结论
如果一个分子具有带电原子,那么从真空DFT计算获得的MESP将完全被该电荷主导,这使得计算的MESP不能于指导分子设计的目的。在本文中,以一个带正电荷的大环PDGFRα抑制剂L2为例,演示如何用设计中性模型分子,然后用Flare QM进行电子密度与ESP计算,最后通过基于QM-场点的可视化验证了蛋白与配体之间发生非经典的Glu675/Cys677-C=O···H—C氢键相互作用的假设。
接下来可以作什么?
- 根据QM计算结果,优化场点模型,为BLAZE虚拟筛选提供更加完美的query
- 根据QM计算结果,为Spark骨架跃迁提供更加完美的query,用来发现新型铰链区结合片段
在药物设计过程中Flare全程陪你
Flare交付先进的科学方法、分析工具和直观、易用的增强功能,洞察您的配体-蛋白质复合物结构。
想要尝试Flare信息丰富、用户友好界面,发现它如何帮助您自信地推动潜在先导化合物优化?请现在就联系我们安排试用,快速访问Flare的广泛功能。我们的专业团队随时准备通过安装和设置为您提供支持,而我们全面的教程库——涵盖从常见工作流程到高级方法和功能的所有内容将帮助您开始使用。我们在这里帮助您更快地实现目标,让您设计出重要的分子。
- 电邮:info@molcalx.com
- 电话:020-38261356
文献
- Derewenda, Z.S., Hawro, I. and Derewenda, U. (2020) “C─H···O hydrogen bonds in kinase-inhibitor interfaces,” IUBMB Life, 72(6), pp. 1233–1242. Available at: https://doi.org/10.1002/iub.2282.
- 用XED力场分析激酶抑制剂的C─H···O氢键. 墨灵格的博客. Available at: http://blog.molcalx.com.cn/2022/01/31/c-h-o-hydrogen-bonds-in-kinase-inhibitor.html
- 用静电分析TYK2 JH1抑制剂铰链结合片段的SAR. 墨灵格的博客. Available at: http://blog.molcalx.com.cn/2023/02/20/tyk2-inhibitor-pf-06826647.html
- Flare QM. https://cresset-group.com/software/flare-qm
- Liang, H. et al. (2025) “Designing Macrocyclic Kinase Inhibitors Using Macrocycle Scaffold Hopping with Reinforced Learning (Macro-Hop),” Journal of Medicinal Chemistry, 68(6), pp. 6698–6717. Available at: https://doi.org/10.1021/acs.jmedchem.5c00087.



