摘要:本文提供了一个Flare Python拓展,根据被选择的原子,创建芳香环药效团特征描述,可用来直接替换CDPKit药效团模型里特征或添加新的特征。本文以SOS1抑制剂8共晶结构(PDB 9MJL)的药效团模型为例,演示了如何通过Flare Extension,根据被选中的6个苯环碳原子创建芳香环特征,用来替换两个疏水特征,并新增一个芳香环特征。
简介
CDPKit提供了基于结构的药效团模型生成方法1,2,这使得可以非常便利地根据蛋白与配体的结合模式生成药效团模型。然而,在实践中发现有时不能捕捉住关键的芳香药效团特征,或者有时候需要根据自己的见解来添加新芳香药效团特征。
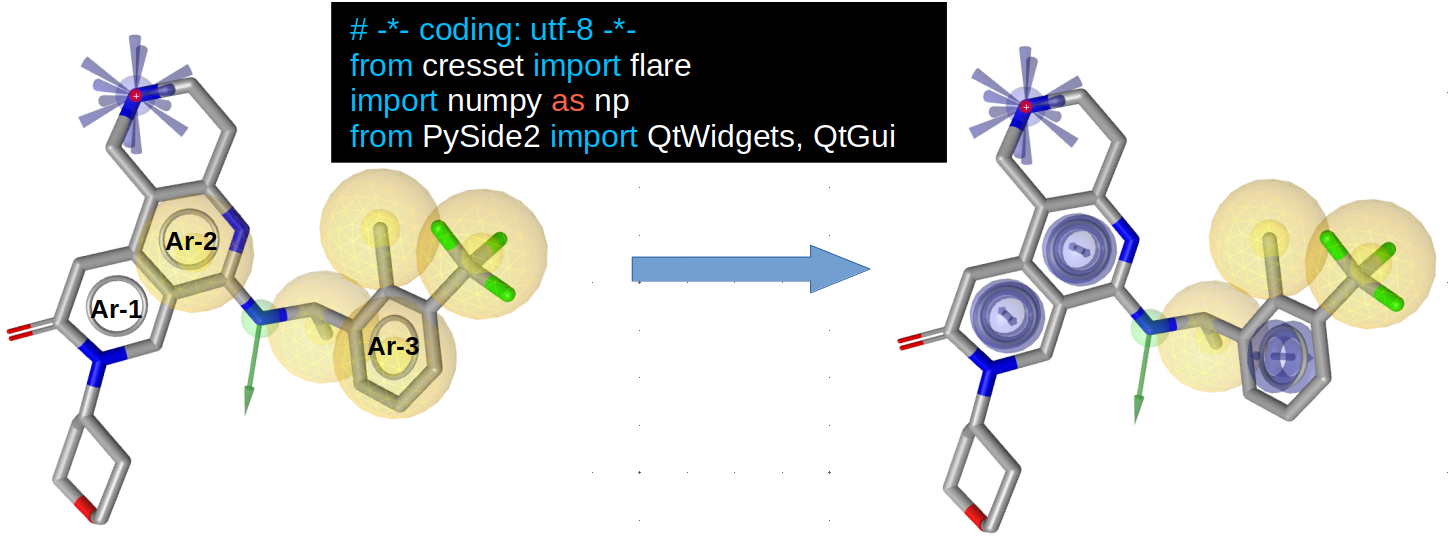
以SOS1抑制剂8的共晶结构PDB 9MJL为例,在Flare Contact分析中,如图1-中与右图所示,三个芳香环(Ar-1、2、3)参与了π-π相互作用;而在CDPKit识别的药效团中,如图1-左所示,Ar-1没有参与相互作用,而Ar-2、3仅参与疏水接触。很明显,CDPKit基于结构的药效团模型不能反映实际相互作用模式,因此有必要根据Flare Contact分析对CDPKit生成的药效团模型进行修正。
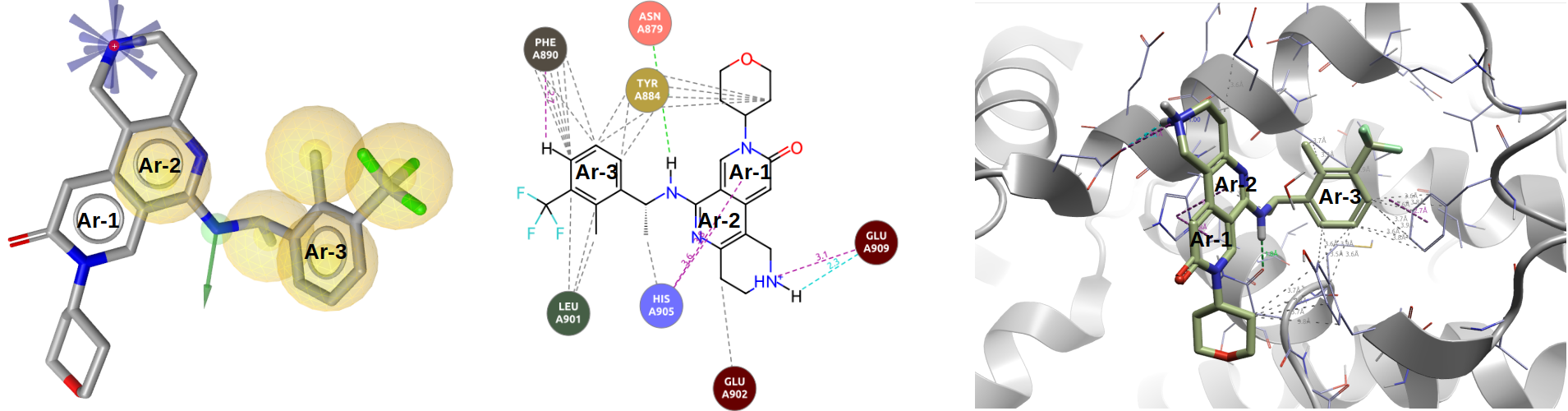
图1. SOS1抑制剂8的共晶结构PDB 9MJL的药效团模型。左:CDPKit生成的基于结构的模型;中间与右边:Flare Contact分析。
在CDPKit中,芳香环药效团特征表示为一个圆形的平面。这个平面通过点法式方程来确定,即利用一个点 P(x0,y0,z0)和一个法向量n=(A,B,C)来定义平面。由于手工计算点P的坐标与法向量比较繁琐,因此有必要写一个脚本自动完成。因此本文的主要目的是,演示了如何通过Flare Extension,根据被选中的原子,自动创建芳香环药效团特征,可直接用来替换CDPKit药效团模型里的特征或添加新的特征。
操作步骤
生成基于结构的药效团
将化合物8与SOS1的共晶结构PDB 9MJL下载到Flare,然后用Protein Prep小心地进行结构准备以添加氢原子、优化氢键、消除原子冲突并将最佳质子化状态分配给蛋白质结构。任何截短的蛋白质链被封端作为蛋白质准备的一部分。
准备完毕,将配体从蛋白表单里提取到配体表单,分别将蛋白与配体结构导出为:
- 9mjl_prot.pdb
- 9mjl_ligand.sdf
然后,使用CDPKit的python脚本2生成基于结构的药效团模型:
1 2 3 | pharm_gen_ia_ph4s.py -r 9mjl_prot.pdb \
-l 9mjl_ligand.sdf \
-o 9mjl_ph4.pml |
在LigandScout或3Dmol.js里可视化,结果如图1-左所示。注意到,芳香环Ar-1、2、3在这个模型里没有参与芳香相互作用,接下来要手动生成它们的芳香环特征表示。
选中用来定义芳香环特征的原子
在Flare的3D视窗中,按住Shift键,用鼠标左键依次单击用来定义芳香特征的原子,以便选中这些原子,完毕如图2所示。
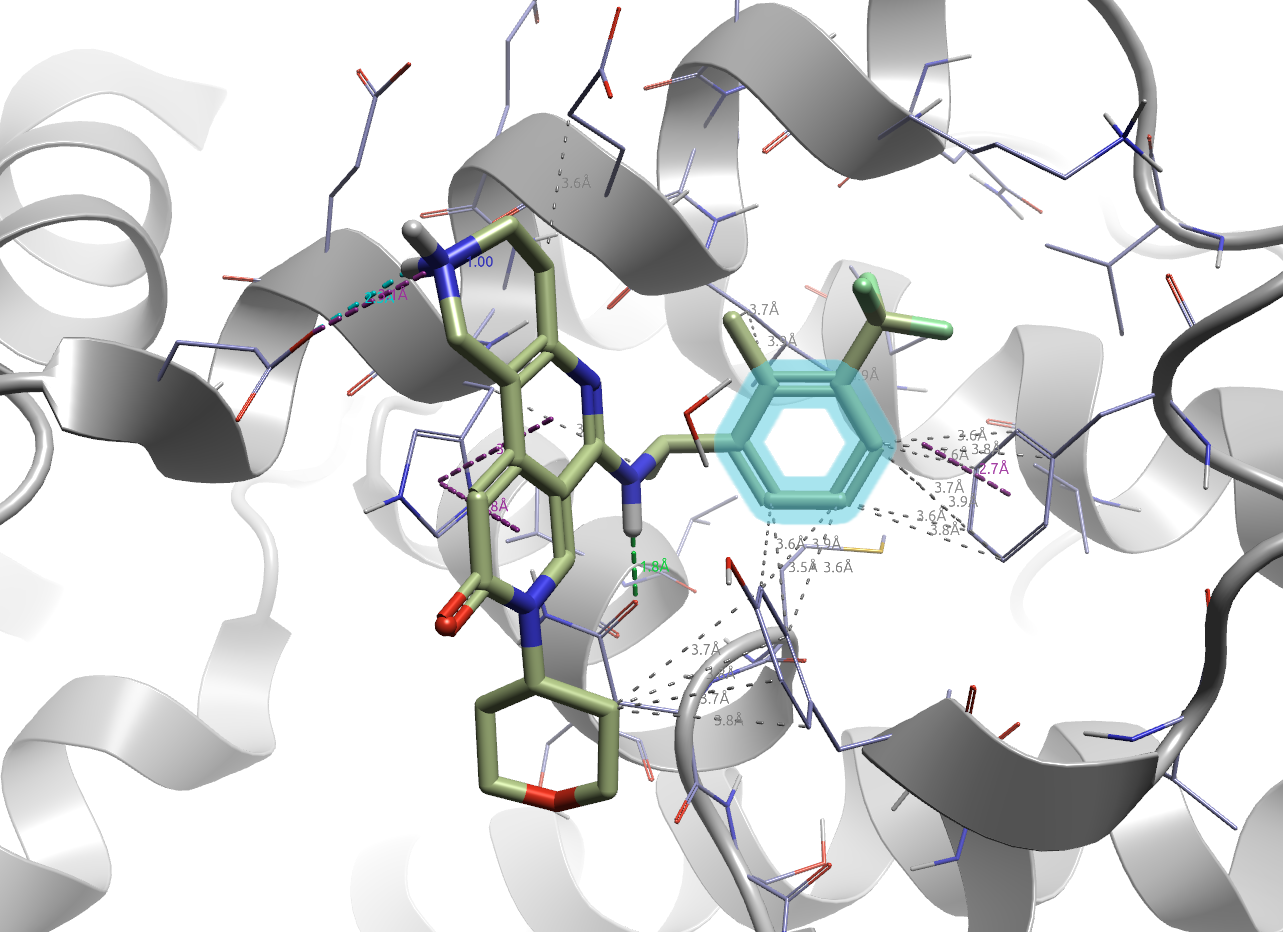
图2. 选中用来定义芳香环的原子
使用Python拓展生成Ar特征
然后,在Flare的Extensions里,Ligand标签下,点击Create AromaticFeat按钮,如图3所示。
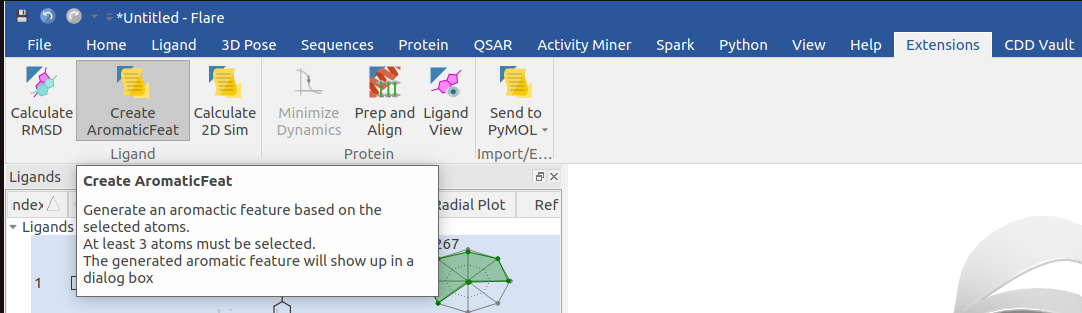
图3. 生成Ar特征的Python拓展
然后会弹出“Define Aromatic Feature”对话框,如图4所示,你可以看到根据选中原子生成的药效团特征。
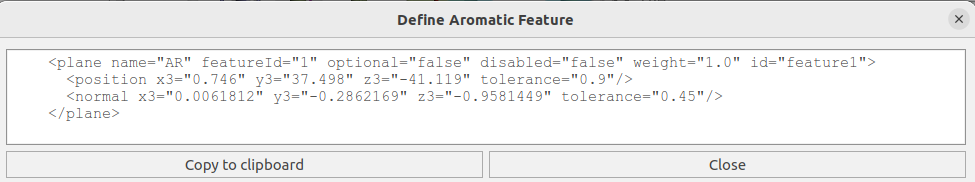
图4. Define Aromatic Feature
药效团特征以XML格式描述,包含一个plane元素,由定义平面的点与法线组成。点击“Copy to clipboard“,然后可以添加到前面生成的药效团模型文件9mjl_ph4.pml里。
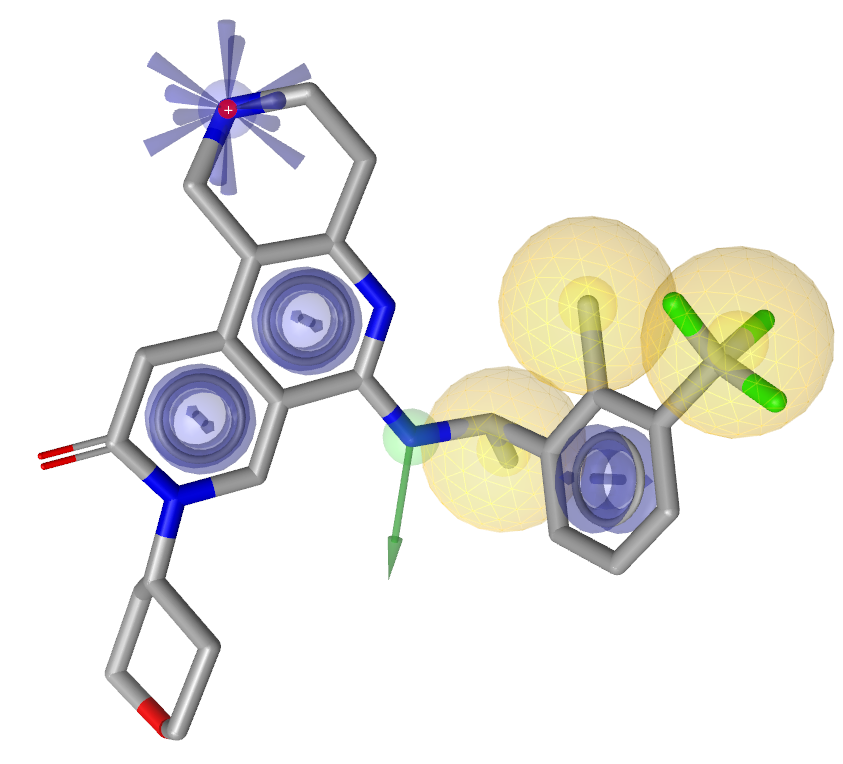
图5. 修正后的药效团模型
依次生成三个芳香环特征。修正后的模型如图5所示,可以看到少了两个疏水特征,而新增加了3个芳香环特征,这个模型与Flare Contact分析的结果一致。其中,芳香环表示为深蓝色圆以及与之垂直的法线,圆的大小通过tolerance来控制。
接下来可以作什么?
- 虚拟筛选大型化合物库
- 虚拟筛选片段库
附件
Python脚本create_cdpkit_ar_feats_ext.py可从github仓库下载3,放在cresset/Flare/python-extensions目录,重启Flare。
文献
- CDPKit Version 1.2.3. Available at: https://github.com/molinfo-vienna/CDPKit
- Generating ligand-receptor interaction pharmacophores.
Available at: https://cdpkit.org/cdpl_python_tutorial/cdpl_python_tutorial.html#generating-ligand-receptor-interaction-pharmacophores - create_cdpkit_ar_feats_ext.py. https://github.com/gkxiao/pyflare-extension
联系我们
Flare V10是Flare最新版本,交付先进的科学方法、分析工具和直观、易用的增强功能,洞察您的配体 – 蛋白质复合物结构。还可以通过Flare Extension无限软件的功能。
想要尝试Flare信息丰富、用户友好界面,以及拓展药效团的功能?请现在就联系我们安排试用,快速访问Flare的广泛功能。我们的专业团队随时准备通过安装和设置为您提供支持,而我们全面的教程库——涵盖从常见工作流程到高级方法和功能的所有内容将帮助您开始使用。我们在这里帮助您更快地实现目标,让您设计出感兴趣的分子。
电邮:info@molcalx.com
电话:020 – 38261356



