摘要:SPARK水分子替换 在之前的博文《SPARK教程|水分子替换》一文中的中描述了如何进行水分子替换优化化合物的活性,在当时的版本中,还没有Water Replacement Wizard。本文专门针对Water Replacement Wizard而写,描述SPARK如何与Flare结合,完美地实现水分子替换。
SPARK水分子替换简介
Spark是一款基于配体的基团替换软件,采用Cresset专有的XED场技术将水与起始配体一起作为参比分子,进行数据库搜索可以找到代替水分子的分子片段。在《SPARK教程 | SPARK替换结晶水分子》一文已经演示了如何用Forge将起始分子与水分子合并,然后用SAPRK的生物等排替换功能, 以一个选择性BTK(Bruton’s tyrosine kinase)抑制剂的X-衍射晶体结构为起始分子(IC50=100nM)进行水分子的替换获得更强的化合物,最后将IC50提高到12nM与4nM水平(Figure 1)。新版的SPARK专门提供了Water replacement模块,让水分子的替换变得非常简单、便利。本文主要演示了如何联用SPARK与Flare,便捷地实现水分子替换。
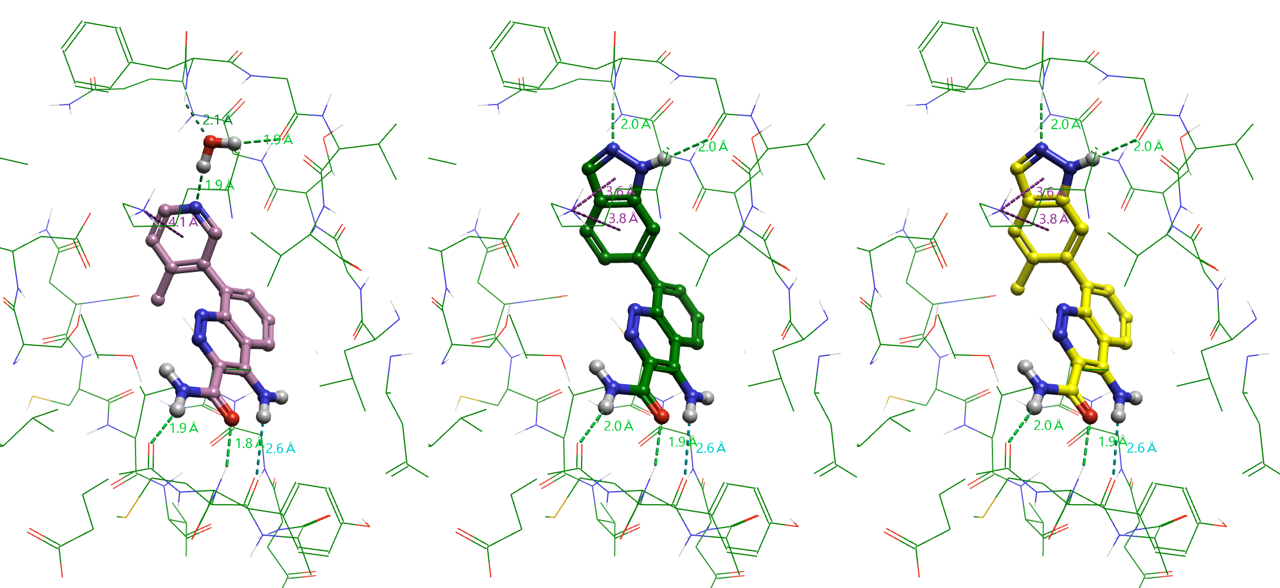
Figure 1. 左:起始的化合物与识别出来的可替换结晶水(PDB ID: 4ZLZ);中、右:SPARK生成的两个被文献报道过活性的化合物,IC50分别为12、4nM。
操作步骤
1.复合物晶体的下载
打开Flare,Flare | File | PDB Downloader, 弹出PDB Downloader对话框,输入PDB 代码:4ZLZ,点击OK按钮。
2. 打开蛋白
此时弹出Open Molecule对话框,确保protonation state下拉菜单为Do full preparation on protein and ligand,其它为默认设置,然后点击OK。
3. 结构准备
如Figure 2所示,在弹出的Flare Protein Preparation对话框中,将Cap trains与Auto Extract Ligand选项框打钩,而去掉Copy protein选项卡的打钩;将active site size设置为6 Å;其它用默认值,点击Start开始准备结构。
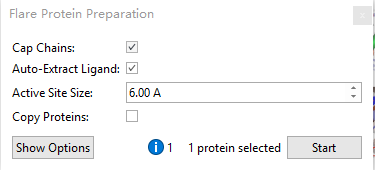
Figure 2. 蛋白结构准备
4. 合并准备好的配体与蛋白结构
准备完毕,在Flare图形界面上的配体表单会出现一个新的配体4VR,在蛋白表单则出现一个蛋白4zlz,用鼠标点击配体表单的4VR配体,按住鼠标左键,将配体拖放到蛋白表单的4ZLZ蛋白里,松开右键,即得到复合物结构。此时,配体表单里的配体也不见了,而蛋白表单里则多了一个配体4VR。
5. 导出复合物结构备用
在蛋白表单里右键点击4zlz,点击export,导出复合物结构,保存为4zlz.pdb,备用。
步骤4,5视频演示
6. SPARK Water Replace实验
双击SPARK图标打开SPARK,选择New Project;或者SPARK | File | New Project,在New Project Wizard中点击Water replacement。
在Load Starter and water to replace对话框中,点击Broswer,读入上一步准备好的复合物结构4zlz.pdb。在弹出的Open Molecules对话框中选择Protonation state为Use input protonation states,因为Flare已经准备好质子化状态;然后点击Open。
7. 选择起始的配体与要替换的水分子
在Protein editor对话框,用鼠标点击名称为4VR配体,再单击use as reference按钮,则4VR被设置为我们的初始配体。
在Residue那一列,找到残基号为954的行,为一个水分子,用鼠标单击它,再单击use as water,现在我们设置好了要被替换的水分子,新设计的分子将有片段代替水与蛋白发生氢键相互作用。
点击Import as protein按钮,导入起始配体与要被替换的水分子。
8. 选择配体被替换的片段
在New project wizard对话框点击Next按钮,弹出Select the region of the starter molecule to replare对话框。按住用鼠标右键,选择甲基吡啶部分。点击next 按钮。
9. 编辑附着点的原子类型
SPARK需要设定附着点的原子类型,以便控制化合物的合理性与合成可行性。这里我的设定如下Figure 3所示。
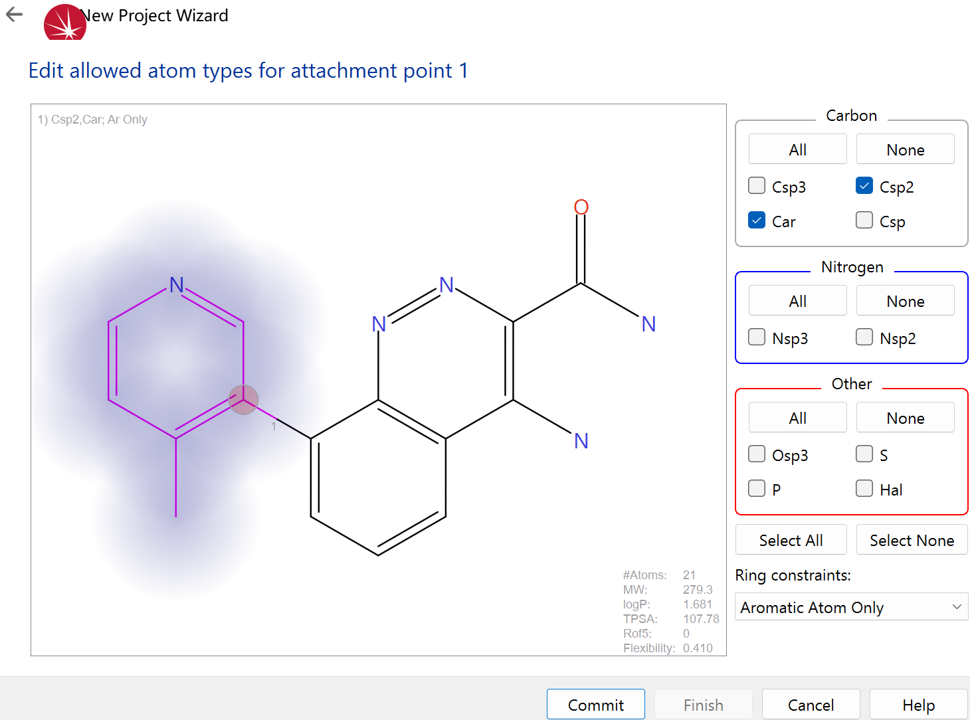
Figure 3. 附着点原子类型的设定
点击Commit,弹出Load additional ligand(s) to guide water displacement,在这里我们直接点Next按钮就可以。
10.设置约束条件:添加场约束
在弹出的Set Constrains对话框里,单击Field Constrains下的Add按钮,然后分别单击水分子的蓝色与红色场点,会出现Constrain 7.00,Constrain 3.85字样(见Figure 4)。说明这两个场点被约束,需要在搜索的过程中被尽量匹配。
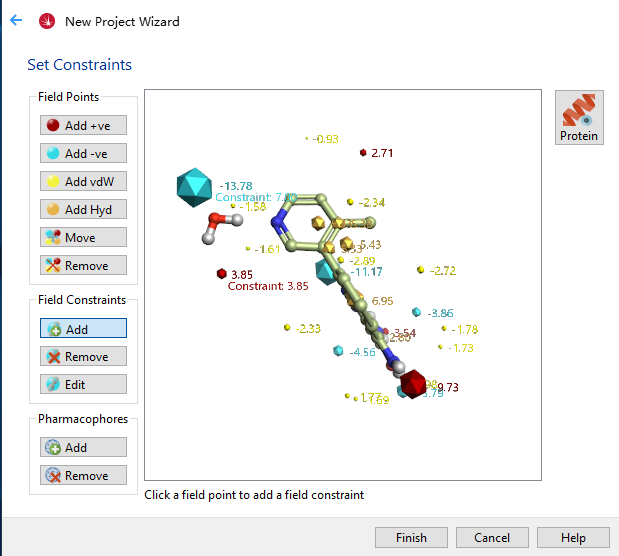
Figure 4. 添加场约束
点击Finish按钮,进入下一步。
11. 选择数据库,开始虚拟筛选
点击Finish按钮后,弹出Spark Search对话框,我们选择合适的数据库即可,具体参照2017年6月15日的《SPARK教程 | SPARK替换结晶水分子》。我的设置如下Figure 5:
Figure 5. 数据库选择
步骤6-11视频演示
结果分析
详见2017年6月15日的《SPARK教程 | SPARK替换结晶水分子》。



