摘要:Wang等人提出的卤键理性设计策略强调通过卤素原子替换与骨架羰基形成氢键的结合位点水分子。hCatL抑制剂SAR为实践这一策略提供了理想案例:甲基(CH₃)取代提高活性,氟(F)取代活性下降,卤素系列呈现梯度提升。本文使用Flare软件整合场技术与GIST水分析技术,对这一案例进行系统分析。蛋白场(PIP)以负静电势可视化表征Gly61羰基氧作为卤键受体的特征;GIST水密度以氧密度与氢密度可视化表征被替换水分子的位置与取向,作为卤键供体的设计模板;配体场可视化揭示氟取代的静电复杂性及卤素σ-空穴的梯度特征;静电互补性分析定量评估取代基的静电贡献(R² = 0.803)。本研究展示了Flare提供的完整技术框架可用于实践卤键理性设计策略,为药物化学家提供可落地的定量分析工具。
Gaokeng Xiao. 2026-02-15. Guiding Rational Halogen Bond Design Using Field and Hydration Analysis: A Case Study of hCatL Inhibitors. Available at: http://blog.molcalx.com.cn/2026/02/27/guiding-rational-halogen-bond-design-using-field-and-hydration-analysis.html
1. 前言
1.1 利用卤键进行理性药物设计的策略
Wang等人1的研究揭示了一个重要现象:在蛋白质-配体复合物中,形成卤键(halogen bond, XB)的卤素原子往往占据着结合位点中原本由水分子占据的位置。值得注意的是,这些被替换的水分子与骨架羰基形成的氢键(O–H···O=C)取向,与配体中卤素原子形成的卤键(C–X···O=C)取向高度一致。基于这一发现,作者提出了一种利用卤键进行理性药物设计的策略:通过引入卤素原子替换与骨架羰基形成氢键的结合位点水分子,从而形成方向性匹配的卤键相互作用(图1)1。该策略强调,为确保替换过程带来有利的熵增,需满足两个关键几何条件:(i)C–X键与O–H键之间的夹角应处于160°–180°的最佳范围内;(ii)卤素原子与被替换水分子的氧原子之间的距离应小于1.5 Å。满足这些条件有望提高母体化合物的结合亲和力。

图1. 通过替换结合位点水分子利用卤键进行理性药物设计的策略。图片改编自文献1。
1.2 配体场:XED力场与静电特征描述
Cresset开发的XED力场采用原子偏心电荷模型,能够更精细地描述分子的静电特征2,3。该力场的独特之处在于将π和σ电子体系的偏电荷贡献进行分离,从而能够准确再现分子周围静电势的各向异性分布。这一特性使XED力场特别适用于探究卤素取代对芳香体系(如苯环)静电势的影响(图2),为理解卤素取代基的电子效应提供了重要的计算工具。
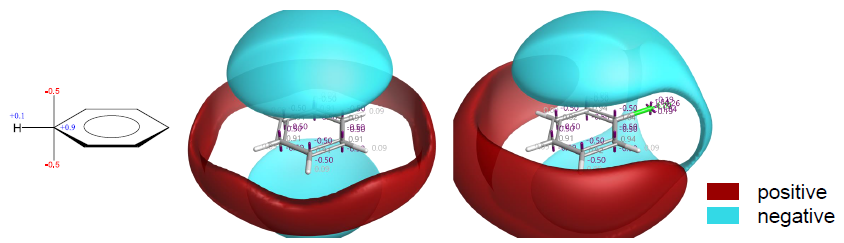
图2. 苯的XED模型:点电荷分布与静电势
1.3 蛋白相互作用势:从配体场到蛋白场
将XED力场拓展至蛋白活性位点的静电环境计算,这对于深入理解配体结合机制、阐释构效关系(SAR)以及设计新型靶向分子具有重要价值4。蛋白相互作用势(protein interaction potentials,PIP)又称为蛋白场,其计算原则上类似于配体场的计算:在蛋白活性位点中填充探针原子,采用距离依赖的复杂介电模型计算每个格点的相互作用势,并将静电势等值化为三维表面。其中,正负静电势表面通过携带氧原子范德华参数的带电探针原子生成。
1.4 静电互补性分析
静电互补性(Electrostatic Complementarity, EC)打分用于量化配体与蛋白结合口袋之间的静电势匹配程度。其核心原理为:当配体与受体的静电势在空间上呈现等量异号(即配体带负电区域对应蛋白带正电区域,反之亦然)时,二者可实现最大化的静电亲和力。Bauer 等人4的研究已表明:当系列类似物间的活性差异主要源于静电性质变化时,EC 打分与实验测得的结合亲和力高度相关。
1.5 GIST水分析:水合位点的预测与热力学表征
由Nguyen等人5提出的GIST(Grid Inhomogeneous Solvent Theory)使用显式水对蛋白质进行约束的分子动力学模拟,并使用非均匀溶剂化理论 (inhomogeneous solvation theory,IST) 计算预定义体积(通常是结合位点)的水分子分布和热力学性质。对蛋白结合口袋进行无配体GIST(apo-GIST)分析,可以获得与羰基氧相互作用的水合位点及其热力学性质、优势氢键取向,用来评估卤键设计取向是否满足Wang等人1提出的设计策略。
1.6 本文目的
hCatL抑制剂系列的完整SAR数据(图3)呈现丰富的趋势1:除Cl、Br、I等可形成卤键的取代基外,甲基(CH₃)与三氟甲基(CF₃)取代同样显著提高活性,而氟(F)取代反而导致活性下降。深入理解这些现象,需要对结合位点的静电特性、立体匹配程度以及被替换水分子的真实能量状态进行综合评估——这正是Flare场技术与GIST水分析技术的优势所在。
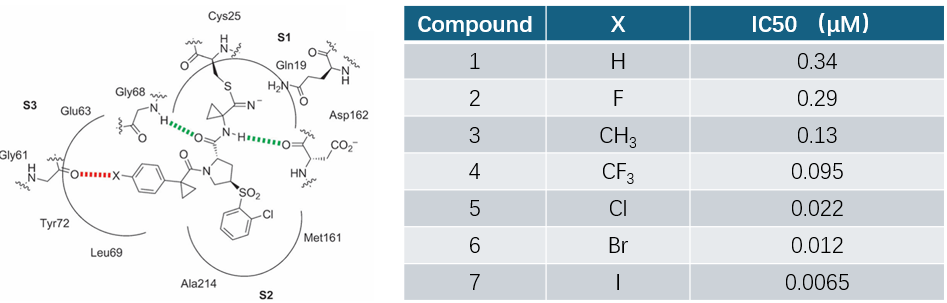
图3. 人组织蛋白酶L(hCatL)抑制剂的SAR。红色虚线:卤素原子与hCatL的Gly61羰基氧形成卤键相互作用。
因此,本文旨在使用Flare软件整合场技术(配体场、蛋白场与EC)与GIST水分析技术,对hCatL抑制剂的SAR进行系统分析,从而实践Wang等人1提出的卤键理性设计策略。通过这一案例研究,我们期望为药物化学家提供一个可落地的卤键设计工具集,使得Wang等人1提出的理论策略能够在实际药物设计项目中得到有效实施。
2. 结果与讨论
2.1 结合位点的场分析
首先使用Flare对hCatL共晶结构(PDB code: 2XU1)进行结构准备,随后在排除HOH2087结晶水(因为在后面GIST计算中为Happy水,ΔG = -1.89 kcal/mol,因此予以保留作为蛋白的一部分)的“干”蛋白的情况下对蛋白进行PIP计算。图4展示了结合口袋中共晶配体氯原子附近5 Å范围内的蛋白相互作用势(PIP)。在C–Cl键的延长方向上,蛋白PIP呈现出强烈的负静电势,表现为蓝色网格等值面(负静电势,PIP \(-ve \ge 6.0 \))。该负静电势区域恰好对应于Gly61羰基氧的位置,表明该羰基氧作为卤键受体,与C–Cl键的σ-空穴(卤键供体)在电荷符号和空间方向上均形成高度互补——这与预期的卤键相互作用特征高度一致。
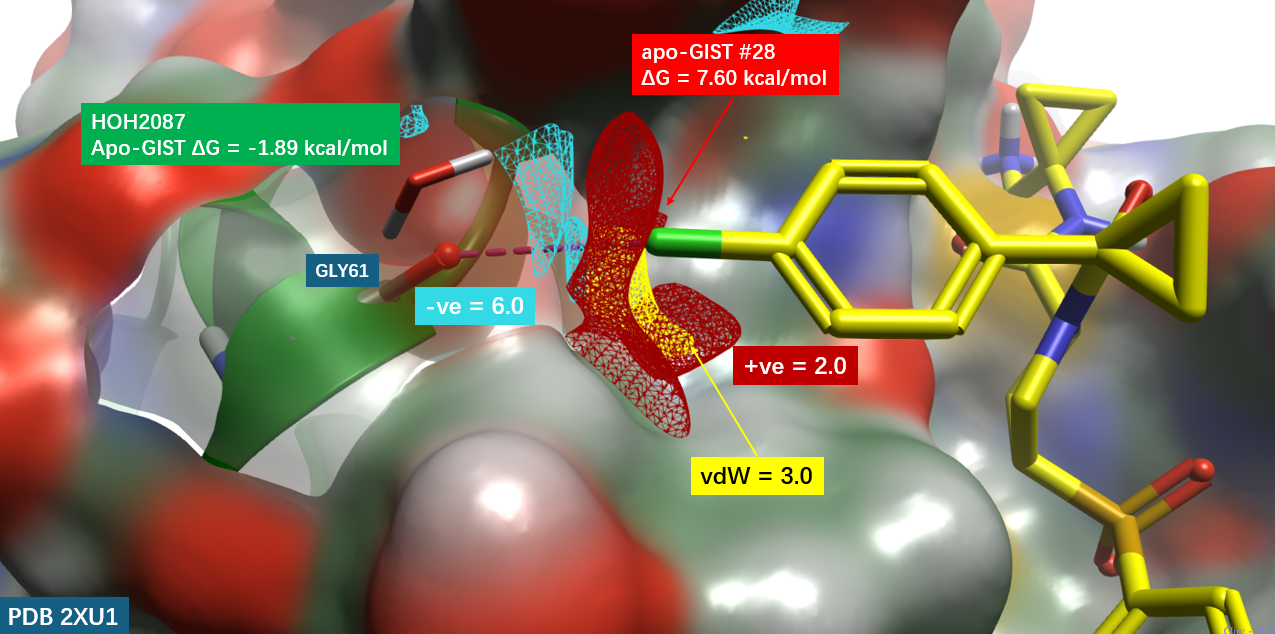
图4. hCatL 结合口袋GLY61处蛋白相互作用势(PIP)分析。分子表面:hCatL,按元素着色;黄色棍棒:hCatL抑制剂,X=Cl。红色网格PIP:\(+ve \ge 2.0 \); 蓝色网格PIP:\(-ve \ge 6.0 \);黄色网格PIP:\(vdW \ge 3.0 \)
在C–Cl键的同一方向上,氯原子近旁还观察到黄色网格等值面(范德华贡献,PIP\(vdW \ge 3.0\)),提示该区域存在强烈的范德华相互作用信号。这些有利的静电与范德华贡献共同支撑了卤键的形成,其强度随卤素原子序数增加而增强的趋势(\(I > Br > Cl\))与本系列化合物的活性数据高度吻合:IC50值分别为I(0.0065 μM)、Br(0.012 μM)、Cl(0.022 μM)。此外,在C–Cl键的绕轴方向上还观察到红色网格等值面(正静电势,PIP \(+ve \ge 2.0 \)),表明蛋白静电场与卤键供体在绕轴方向上也存在互补性,进一步丰富了卤键相互作用的静电基础。
上述分析表明,利用Flare的PIP技术可以有效表征蛋白结合口袋的静电特征,并以负静电势(-ve PIP)作为卤键受体的可视化代理,快速评估目标口袋是否具备开展卤键设计的潜力。
2.2 静电互补性分析
hCatL抑制剂SAR系列化合物的区别仅在于苯环对位的取代基(H, F, CH₃, CF₃, Cl, Br, I),这使得该系列化合物在形状上高度相似,而主要差异集中于静电性质——这正是应用静电互补性(Electrostatic Complementarity, EC)打分进行定量分析的理想场景4。
| 取代基(X) | pIC50 | EC Score | ΔGwatdisp |
|---|---|---|---|
| H | 6.54 | 0.284 | -26.168 |
| F | 6.47 | 0.298 | -32.278 |
| CH3 | 6.89 | 0.29 | -32.602 |
| CF3 | 7.02 | 0.308 | -33.393 |
| Cl | 7.66 | 0.307 | -32.602 |
| Br | 7.92 | 0.321 | -32.602 |
| I | 8.19 | 0.319 | -32.602 |
ΔGwatdisp单位:kcal/mol
在Flare中,我们以hCatL共晶结构(PDB ID: 2XU1)的配体为模板,通过结构编辑将氯原子依次替换为其他取代基,并使用XED力场对各取代基进行几何优化以获得合理的构象。随后,将优化后的系列化合物与去除结晶水(仅保留“Happy” HOH 2087作为受体的一部分 )的蛋白模型(“干”蛋白)关联,计算每个化合物与蛋白的EC打分值,并将其与实验测得的pIC50进行相关性分析。EC打分与pIC50数据汇总于表1。
从表1数据可以看出,除氟取代外,其余取代基的EC打分与pIC50呈现出良好的协同趋势:CH₃ (0.29/6.89)、CF₃ (0.308/7.02)、Cl (0.307/7.66)、Br (0.321/7.92)、I (0.319/8.19),EC打分随原子序数递增,pIC50同步提升。这一趋势与卤素原子的σ-空穴强度(\(I > Br > Cl\))及其所形成的卤键强度顺序高度一致,表明EC打分所捕捉的静电贡献是卤键形成能力的重要组成部分,并与卤素取代基对活性的提升作用密切相关。氢取代的EC打分(0.284/6.54)作为基线,与上述趋势一致。
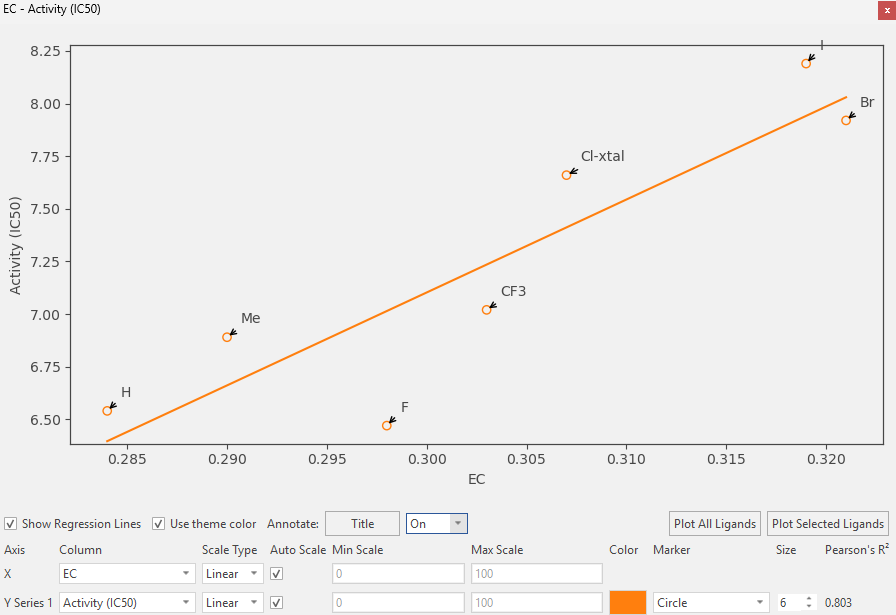
图5. hCatL抑制剂SAR系列的EC打分与pIC50的相关性分析,Pearson R2 = 0.803。
氟取代的EC打分(0.298)高于氢(0.284)与甲基(0.29),但其pIC50(6.47)却略低于氢(6.54)与甲基(6.80),成为该系列中唯一偏离整体趋势的数据点。EC打分与pIC50的相关性分析显示(图5),二者总体呈现良好的线性关系(Pearson R2 = 0.803),氟的偏离恰好落在回归线下方,提示其可能存在除静电贡献之外的去稳定因素。
EC打分与pIC50的良好相关性(Pearson R2 = 0.803)表明,该方法能够有效捕捉取代基静电效应对结合亲和力的贡献,与Bauer等人4的观察一致——在静电主导的SAR系列中,EC打分与实验活性高度相关。氟取代的例外情况提示,除静电互补性外,取代基的立体匹配程度、溶剂置换自由能可能对结合贡献同样重要。为深入理解氟取代的静电行为,我们进一步对不同取代基的配体场进行了分析。
2.3 配体场分析:氟取代的静电复杂性
为了深入理解氟取代为何成为EC打分趋势中的唯一异常点,我们分析了不同取代基的配体场(图6)。配体场可视化基于XED力场计算,展示了分子周围静电势的空间分布特征。
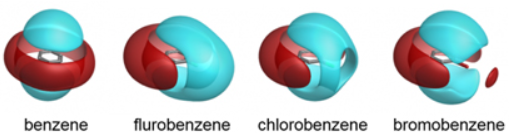
图6. 苯环不同取代的配体场。从左至右:苯、氟苯、氯苯、溴苯。红色等值面:正静电势;蓝色等值面:负静电势。图片改编自文献6。
从图6可以看出,苯环对位取代基的改变对配体静电场产生显著影响。与苯(X=H)相比,氟苯(X=F)的配体场呈现出独特的各向异性特征:沿C–F键的延长方向上,氟原子周围出现更强的负静电势区域(红色等值面),这与氟原子高电负性导致的强吸电子效应一致;然而,在垂直于C–F键的绕轴方向上,氟苯同样表现出比苯更负的静电势分布。
这一观察为理解氟取代的EC打分提供了关键线索。如2.1节所述,hCatL结合口袋中Gly61羰基氧处呈现强烈的负静电势(图4中蓝色PIP等值面,PIP \(-ve \ge 6.0\))。沿C–X轴向的负配体场(氟苯 \(>\) 苯)理论上会与蛋白的负PIP产生更强的静电排斥,不利于结合。然而,EC打分是配体与蛋白在整个接触界面上静电势点乘的加权和,而非仅取决于单一方向。氟苯在绕轴方向上更负的静电势分布,使其与蛋白其他区域的正静电势形成更优的静电互补,从而在整体EC打分上略高于苯。
这一现象揭示了氟取代静电效应的复杂性:取代基不仅改变局部的静电势,还通过共轭效应影响整个芳香体系的电子分布。对于氟原子而言,其强吸电子效应通过诱导效应和共振效应共同作用于苯环,导致静电势的各向异性重分布。这使得氟取代在轴向引入不利的静电排斥(与Gly61羰基氧的负PIP),但在绕轴方向贡献有利的静电互补——两者竞争的结果是EC打分略高于氢,但活性却因轴向不利排斥而略低于氢。
卤素系列(Cl, Br, I)的配体场则呈现出典型的各向异性特征:沿C–X键延长方向出现明显的正静电势区域(σ-空穴),且强度随原子序数增加而增强(\(I > Br > Cl\))。这一特征与图2中蛋白Gly61羰基氧的负PIP形成完美的静电互补,是卤键形成的静电基础。尽管I取代的EC打分比Br的略低,这可能与刚性的蛋白模型有关系,尤其是HOH2087位置固定与实际会略微移动以适应不同的卤素存在区别。
综上,配体场分析揭示了氟取代静电行为的复杂性,解释了其EC打分略高于氢的原因,同时明确了氟的活性劣势源于轴向静电排斥。这一分析也提示,除静电互补性外,取代基的立体匹配程度可能对结合贡献同样重要——氟原子的范德华半径(1.47 Å)显著小于水合位点的空腔尺寸(~2.0 Å),可能导致其无法获得充分的范德华接触。这一问题将在2.4节结合GIST水分析进一步探讨。
2.4 apo-GIST水分析:水合位点作为卤键供体的代理
GIST(Grid Inhomogeneous Solvation Theory)将水的热力学性质表示为空间位置的连续函数,这与传统IST方法仅聚焦于离散水合位点的思路不同。在GIST中,水合位点由水分子氧原子密度的高值区域自然显现,而水分子的取向则由氧密度与氢密度分布共同决定。因此,通过分析氧密度与氢密度的空间关系,可以获得水合位点中水分子的优势取向信息。
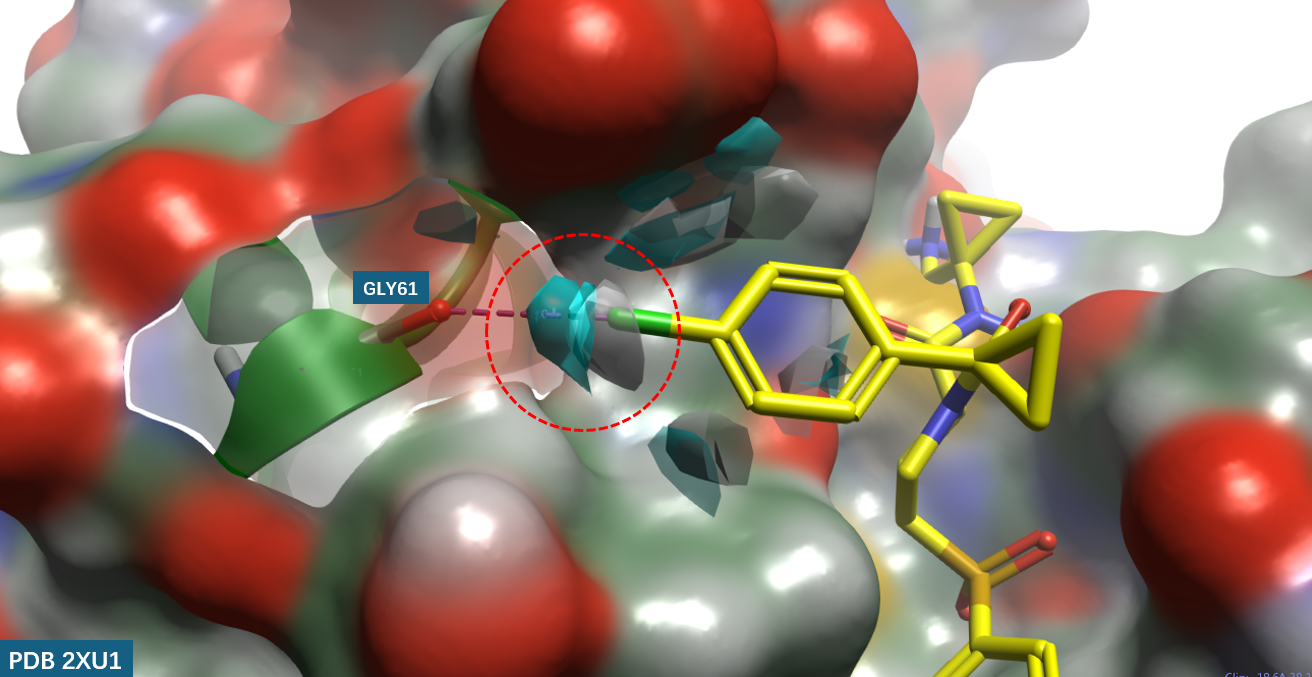
图7. hCatL结合口袋Gly61处的apo-GIST分析结果。分子表面:hCatL,按元素着色;黄色棍棒:hCatL抑制剂(X = Cl);灰色等值面:GIST水氧密度(密度阈值 = 4);青蓝色等值面:GIST水氢密度(密度阈值 = 4)。红色圆圈高亮处为氯原子、感兴趣的水合位点。
图7展示了hCatL结合口袋中氯原子附近4 Å范围内的GIST分析结果。灰色等值面表示水分子氧密度(density = 4),青蓝色等值面表示水分子氢密度(density = 4)。值得注意的是,氯原子的位置恰好位于灰色氧密度等值面的中心(红色圆圈高亮处),表明该氯原子物理上占据了一个原本由水分子占据的位置——即替换了一个水合位点。更为关键的是,沿着灰色氧密度中心与青蓝色氢密度中心的连线方向,恰好与C–Cl键的延长方向一致,并指向Gly61羰基氧。这一几何关系完美复现了该水合位点与Gly61羰基氧之间形成的O–H···O=C氢键取向。换言之,GIST所揭示的水分子取向与卤键形成所需的方向性高度吻合。
上述观察具有重要的方法论意义:GIST水氧密度与水氢密度可以作为与骨架羰基形成氢键的水合位点的可视化代理。通过考察配体C–X键取向与GIST所揭示的O–H···O=C取向的一致性,可以快速判断该位点是否满足Wang等人1提出的卤键形成条件——即卤素原子是否以相似的取向替换了原有的水分子。
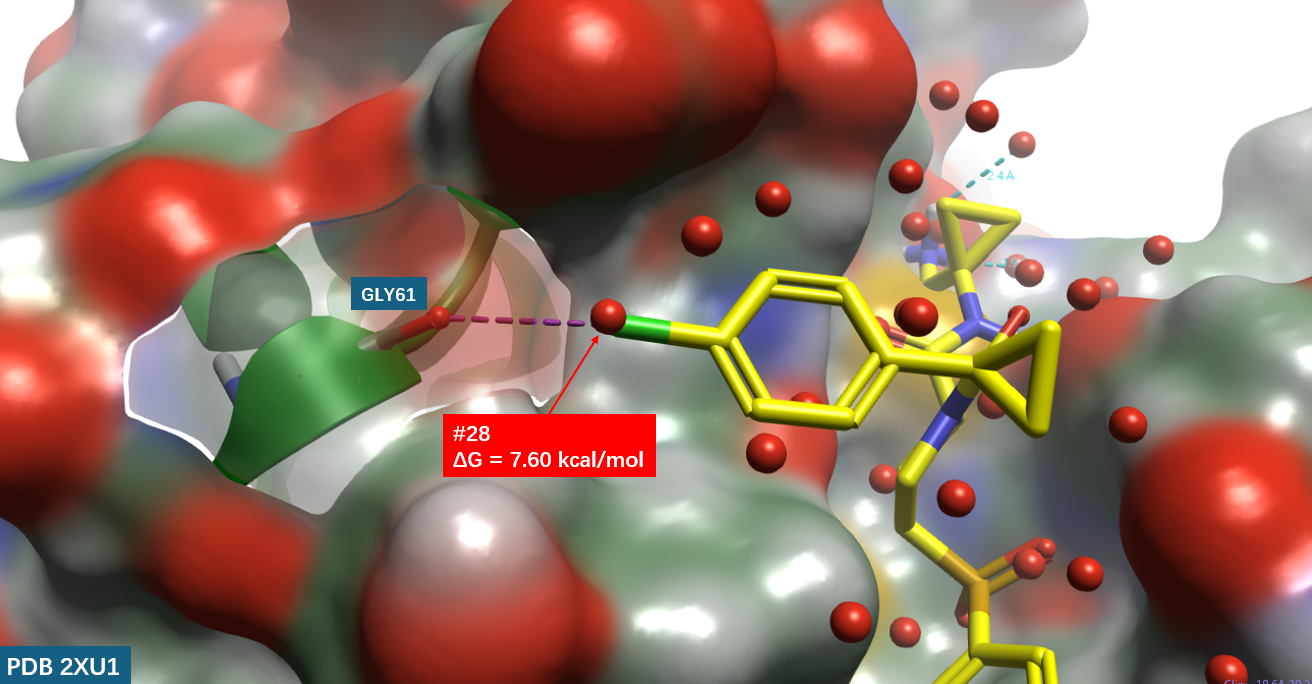
图8. hCatL结合口袋Gly61处的apo-GIST水合位点。分子表面:hCatL,按元素着色;黄色棍棒:hCatL抑制剂(X = Cl);红色球体:apo-GIST水合位点。
图8进一步展示了通过峰值拾取法从GIST密度场中识别出的离散水合位点(仅显示距离配体4 Å范围内的位点)。其中编号为#28的水合位点恰好与氯原子位置重合——这与Wang等人1报道的W10位点一致。该水合位点的溶剂化自由能(ΔGsolv)经GIST计算为+7.60 kcal/mol。这是一个典型的高能(不稳定)水合位点,意味着将其从结合位点移除并释放至体相水中将带来显著的自由能收益。
基于这一发现,我们可以量化各取代基因置换#28所获得的自由能增益。以氢取代为基准(其ΔGwatdisp = -26.168 kcal/mol,反映的是未发生#28水置换时的基准状态),其他取代基因占据#28位点而获得的去溶剂化增益(ΔΔGwatdisp)可通过公式1计算:
$$
\Delta \Delta G_{watdisp} = \Delta G_{watdisp}(X) – \Delta G_{watdisp}(H)
$$
计算结果已列于表1的ΔGwatdisp列。氟取代的ΔΔGwatdisp为-6.11 kcal/mol;而甲基及所有卤素取代(CH₃、Cl、Br、I)的ΔΔGwatdisp均为-6.43 kcal/mol,三氟甲基取代的ΔΔGwatdisp最大,为-7.23 kcal/mol。这一结果表明:所有能够有效占据#28水合位点的取代基(无论是否形成卤键)均可从水置换中获得相近的自由能增益。这为两个关键现象提供了统一解释:
- 甲基为何能提高活性:尽管甲基不能形成卤键,但其尺寸恰好能够填满#28空腔,获得与卤素相当的水置换收益(ΔΔG ≈ -6.43 kcal/mol),从而显著提升活性。
- 三氟甲基为何提高活性:三氟甲基具有最大的置换自由能收益,比甲基多了-0.8kcal/mol,再加上更好的静电互补性,因此比甲基取代活性更高。
- 氟为何活性下降:氟的ΔΔGwatdisp(-6.11 kcal/mol)与其他取代基相近,但其活性却低于氢——这进一步印证了2.3节的推论:氟的活性劣势源于轴向静电排斥(与Gly61羰基氧的负PIP不利相互作用)及立体不匹配(范德华半径过小,无法获得充分的范德华接触)。
卤素系列(Cl、Br、I)在获得相同水置换收益的基础上,通过卤键的额外贡献(静电互补与σ-空穴相互作用)实现了活性的进一步梯度提升,其趋势与EC打分及配体场观察完全一致。
2.5 讨论:水合位点自由能的计算方法差异与数据解释力
本研究中,GIST分析显示#28(W10)水合位点的溶剂化自由能ΔGsolv = +7.60 kcal/mol。这一数值与Wang等人1报道的卤键数据集平均置换自由能(-1.68 kcal/mol)在符号上存在差异。由于两种方法均以体相水为参考态(即将水分子从结合位点移至体相的自由能变化),这一差异并非源于参考系选择,而是反映了不同计算方法在力场参数、熵估算策略、采样方式等方面的本质区别——这正是水合位点热力学计算中常见的方法依赖性现象。
Wang等人1采用的SPA方法与本文使用的GIST方法虽同属基于分子动力学模拟和IST理论的计算框架,但在具体实现上存在显著差异:力场选择(AMBER99SB vs. AMBER14SB)、熵估算的近似处理、水合位点识别策略(轨迹聚类 vs. 网格密度积分)等均可能影响最终计算结果。因此,对同一水合位点给出不同的绝对自由能值并不意外——SPA将其归入卤键数据集统计平均值(-1.68 kcal/mol),而GIST识别为+7.60 kcal/mol的不稳定水。
更为关键的是,一个计算方法的价值最终取决于其能否自洽地解释实验现象。在本研究中,GIST所揭示的#28(W10)高能状态为hCatL抑制剂系列的全部SAR现象提供了简洁而统一的解释:
- 甲基(CH₃)的活性提高:若W10为高能水(ΔGsolv = +7.60 kcal/mol),则任何能够有效占据该空腔的基团置换都将带来显著的自由能收益。甲基虽不能形成卤键,但其尺寸恰好填满W10空腔,获得了与卤素相当的水置换收益(ΔΔGwatdisp ≈ -6.4 kcal/mol),从而显著提高活性(pIC50从6.54提升至6.89)。这一现象在“稳定水”框架下难以解释——若W10为稳定水,无卤键的甲基应导致活性下降,与实验观察相悖。
- 氟(F)的活性下降:氟的ΔΔGwatdisp(-6.11 kcal/mol)与其他取代基相近,但其pIC50(6.47)却低于氢(6.54)——这提示氟的劣势来源于水置换收益之外的因素。结合2.3节配体场分析,氟在C–F轴向上更强的负静电势与蛋白Gly61羰基氧的负PIP产生静电排斥;加之氟的范德华半径(1.47 Å)显著小于W10空腔尺寸(~2.0 Å),无法获得充分的范德华接触。这两方面因素共同抵消了部分水置换收益,导致活性下降。
- 卤素系列(Cl、Br、I)的梯度活性:在获得相同水置换收益(ΔΔGwatdisp ≈ -6.4 kcal/mol)的基础上,卤素取代基通过σ-空穴与蛋白负PIP的静电互补形成卤键,实现了活性的额外梯度提升(pIC50:7.66 → 7.92 → 8.19)。尽管EC打分(0.307 → 0.321 → 0.319)及配体场观察的σ-空穴强度(\(Cl > Br > I\))并不完全一致,但是Br与I的EC打分还是最高一类。I与EC打分出现差异,主要是因为HOH2087作为氢键供体与C-X绕轴负静电场相互作用时蛋白刚性的处理所致,还包括忽略了其它水分子的贡献。
值得指出的是,本研究聚焦于配体结合后对结合位点水分子的置换收益,尚未系统考虑配体从水相转移至结合位点过程中所涉及的去溶剂化自由能损耗。配体在进入结合口袋前,其自身周围的水化层被破坏同样伴随着自由能变化——这一贡献对结合亲和力的影响不可忽视,尤其是在取代基极性差异较大的系列中。对这一因素的定量考察将是后续研究的重要方向。
然而,无论是否考虑配体去溶剂化,本研究的核心结论已获得充分支持:#28(W10)是一个高能水合位点,其置换贡献是活性提升的基础,而静电互补性与立体匹配程度在此基础上进行调节。GIST分析所提供的连续密度场与热力学数据,为实践Wang等人1提出的卤键理性设计策略提供了更为精细的定量工具——通过识别高能水合位点、评估其被置换的收益、并结合场技术分析取代基的静电与立体适配性,药物化学家可以在引入卤素前做出更为理性的设计决策。
3. 方法
3.1 蛋白的结构准备
从蛋白质数据库(PDB)下载配体 – 蛋白质复合物结构(PDB code: 2XU1)至 FlareTM (Version 11)软件7中。使用 Protein Prep 工具进行标准化预处理,包括添加氢原子、优化氢键网络、消除原子冲突及分配最佳质子化状态。截短的蛋白质链末端进行了封端处理。
3.2 apo-GIST分析
GIST5 是一种使用显式水对蛋白质进行约束的分子动力学模拟,并使用非均匀溶剂化理论 (inhomogeneous solvation theory,IST) 计算预定义体积(通常是结合位点)的水分子分布和热力学性质。在本文中,用Flare 实现的GIST方法8分别对 PDB 2XU1 结合位点进行了无配体的GIST分析,使用了如下条件:
- Calculation method: Normal
- Ligand: None
- Grid spacing:0.5 Å
- Grid Definition:Ligand
- Chains: A, Water
- Simulation length: 20ns
- Solvent Model: explicit TIP4Pew Water
计算完毕,采用文献 9 的贪心峰值拾取(greedy peak picking)法对水氧密度网格进行聚类获得水合位点,并用公式(5)方法计算水合位点溶剂化自由能,最后用公式(8)的方法计算配体的溶剂置换自由能ΔGwatdisp。
标准的Flare GIST计算并不直接给出水氢密度,这里我使用GIST的Dynamics轨迹使用Pyflare脚本生成。
3.3 蛋白相互作用势(PIP)分析
蛋白相互作用势(Protein Interaction Potential, PIP)是 Cresset 分子相互作用势在蛋白体系上的延伸,均基于 XED 力场计算。该方法原理类似于配体场计算:用探针原子填充蛋白活性位点,计算每个格点上的相互作用势3,4。在本文中,PIP 通过 Flare | Protein | Field Surface 对 PDB 2XU1 的Chain A 进行计算,主要参数如下:
- Surface Type: +ve, -ve, vdW
- Quality: Very High
- Region: Near select ligand
- Protein: 2XU1
- Chains: A + HOH2087
3.4 配体场分析
基于PDB 2XU1共晶化合物,在Flare中对氯原子进行编辑,然后在Very Accurate XED模式下对新增的基团进行几何优化,随后计算化合物的配体场
4. 结论
本研究以hCatL抑制剂系列为案例,展示了如何使用Flare软件整合场技术与GIST水分析技术,实践Wang等人提出的卤键理性设计策略。通过这一案例分析,我们得出以下结论:
第一,GIST水密度可作为被替换水分子的可视化代理。 apo-GIST分析显示,#28水合位点(W10)的氧密度与氢密度分布清晰揭示了该水分子与Gly61羰基氧形成氢键的取向。氯原子位置恰好与氧密度中心重合,且C–Cl键方向与氧密度-氢密度连线方向一致——这一可视化证据直观验证了卤素原子以相似取向替换原有水分子的设计原则。#28(W10)的溶剂化自由能ΔGsolv = +7.60 kcal/mol表明其为高能水合位点,所有能够有效占据该位点的取代基均可获得相近的水置换收益(ΔΔGwatdisp ≈ -6.4 kcal/mol),解释了甲基(CH₃)虽不能形成卤键但仍显著提高活性的现象。
第二,蛋白场(PIP)可作为卤键受体的可视化代理。 PIP分析以负静电势等值面(PIP\( -ve \ge 8.0\))清晰表征了Gly61羰基氧作为卤键受体的空间位置与电荷特征。该负静电势区域与卤素原子的σ-空穴在方向上形成高度互补,为卤键的形成提供了直观的静电依据。
第三,配体场可视化揭示了取代基的静电特征及其与蛋白场的匹配关系。 氟取代在C–F轴向上呈现更强的负静电势,与蛋白负PIP产生静电排斥;而在绕轴方向呈现更负的静电势分布,贡献了有利的静电互补——两者竞争的结果解释了氟的EC打分略高于氢但活性略低于氢的现象。卤素系列的配体场清晰展示了σ-空穴强度随原子序数递增的特征(\(I > Br > Cl\)),与卤键形成能力及活性趋势高度一致。
第四,静电互补性分析为取代基静电贡献提供了定量评估。 EC打分与pIC50的相关性分析(R² = 0.853)表明,该方法能够有效量化取代基静电效应对结合亲和力的贡献。卤素系列的EC打分与活性呈现完美的协同趋势,进一步验证了静电互补性作为卤键设计定量指标的有效性。
第五,Flare提供的完整技术框架可系统实践卤键理性设计策略。 本研究展示了一套以可视化代理为核心的可操作工作流:(i)以GIST水密度为模板,识别可被替换的高能水合位点及其取向;(ii)以蛋白场为参照,识别卤键受体的静电特征;(iii)以配体场为工具,评估取代基的静电特征与σ-空穴强度;(iv)以静电互补性分析为度量,定量评估静电匹配程度。这一框架使Wang等人提出的理论策略能够在实际药物设计项目中得到有效实施。
值得指出的是,本研究尚未系统考虑配体从水相转移至结合位点过程中的去溶剂化自由能损耗,这将是后续研究的重要方向。尽管如此,本研究已充分展示:Flare场技术与GIST水分析技术的整合,通过将抽象的静电与水合位点性质转化为可视化的代理信息,为卤键理性设计提供了直观、可操作、可量化的工具集。
5. 在自己的项目中使用Flare的场技术与水分析技术
场技术与GIST水分析技术是药物设计平台Flare的模块,最新版Flare V11 引入了强大的新科学能力、更快的工作流和增强的易用性,为您提供高效、精准探索复杂体系的工具。
立即联系我们申请试用,体验 Flare 的最新功能。我们的专家团队将指导您完成安装与设置,同时我们丰富的教程库(从基础工作流到高级方法)确保您顺利上手。借助 Flare V11,您将能够更快推进研究、深入洞察机制,并设计出最具价值的分子。
联系我们: 020-38261356 info@molcalox.com
6. 文献
- Wang, Y.; Fu, Q.; Zhou, Y.; Du, Y.; Huang, N. Replacement of Protein Binding-Site Waters Contributes to Favorable Halogen Bond Interactions. J Chem Inf Model 2019, 59 (7), 3136–3143. https://doi.org/10.1021/acs.jcim.9b00128.
- Vinter, J. G. Extended Electron Distributions Applied to the Molecular Mechanics of Some Intermolecular Interactions. J Comput Aided Mol Des 1994, 8 (6), 653–668. https://doi.org/10.1007/BF00124013.
- Cheeseright, T.; Mackey, M.; Rose, S.; Vinter, A. Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665–676. https://doi.org/10.1021/ci050357s.
- Bauer, M. R.; Mackey, M. D. Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes. J Med Chem 2019, 62 (6), 3036–3050. https://doi.org/10.1021/acs.jmedchem.8b01925.
- Nguyen, C. N.; Kurtzman Young, T.; Gilson, M. K. Grid Inhomogeneous Solvation Theory: Hydration Structure and Thermodynamics of the Miniature Receptor Cucurbit[7]Uril. Journal of Chemical Physics 2012, 137 (4), 973–980. https://doi.org/10.1063/1.4733951.
- Science Overview. https://cresset-group.com/science/overview
- Flare (Version 11). https://cresset-group.com/software/flare
- Flare GIST. https://cresset-group.com/software/flare-3drism
- 基于GIST的水合位点分析及其在基于结构设计中的应用. Available at: http://blog.molcalx.com.cn/2024/10/04/gist-based-hydration-site-analysis.html



