摘要:通过比较蛋白家族内和跨蛋白质家族间的结合位点,可以提取到靶标蛋白的功能与选择性等相关细节,从而为开发新配体提供有用的见解。 典型的方法主要聚焦于蛋白序列分析,在序列相似性低的情况下会导致不确定的预测。SiteHopper为传统药物发现目的的序列比对方法提供了一种强大的替代方法。
SiteHopper简介
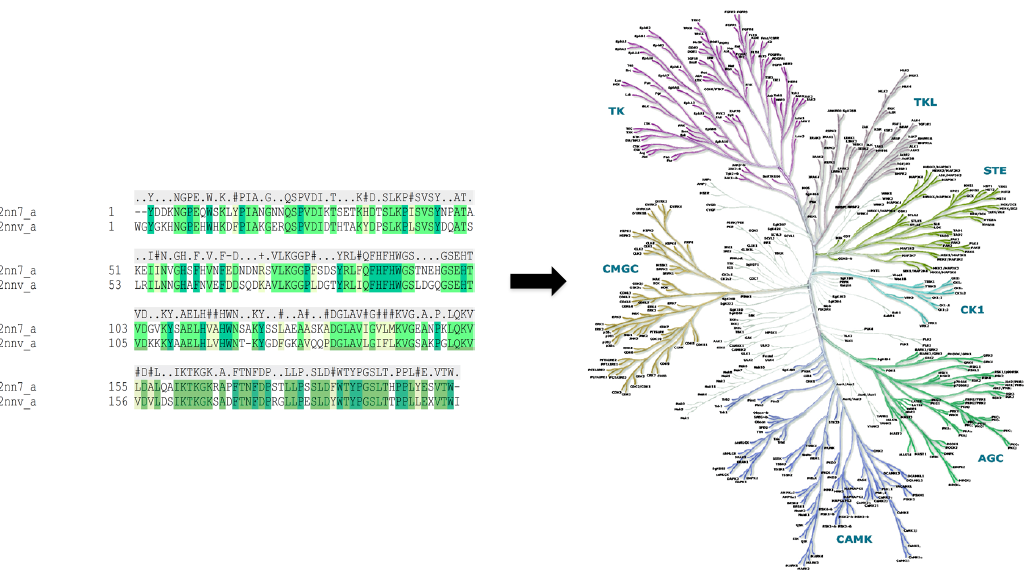
图1. 基于序列蛋白激酶家族聚类。这个例子说明了相似的蛋白可有非常多样的序列。
基于配体的虚拟筛选的核心思想是相似的配体通常结合到相似的蛋白质结合位点。这一想法的推论是,相似的结合口袋可能会结合相同的配体,这使得结合位点比较成为有用的工具,例如 用于选择性研究或脱靶效应分析。我们的新工具SiteHopper使用一种基于形状的方法来解决这个问题,Sitehopper用3D形状和表面化学特征来定义结合位点。这种称为补丁的口袋定义可以从公共或内部来源收集到数据库中。将感兴趣的口袋作为查询式用于搜索该数据库,生成一个叠合好的相似口袋的命中列表。
方法
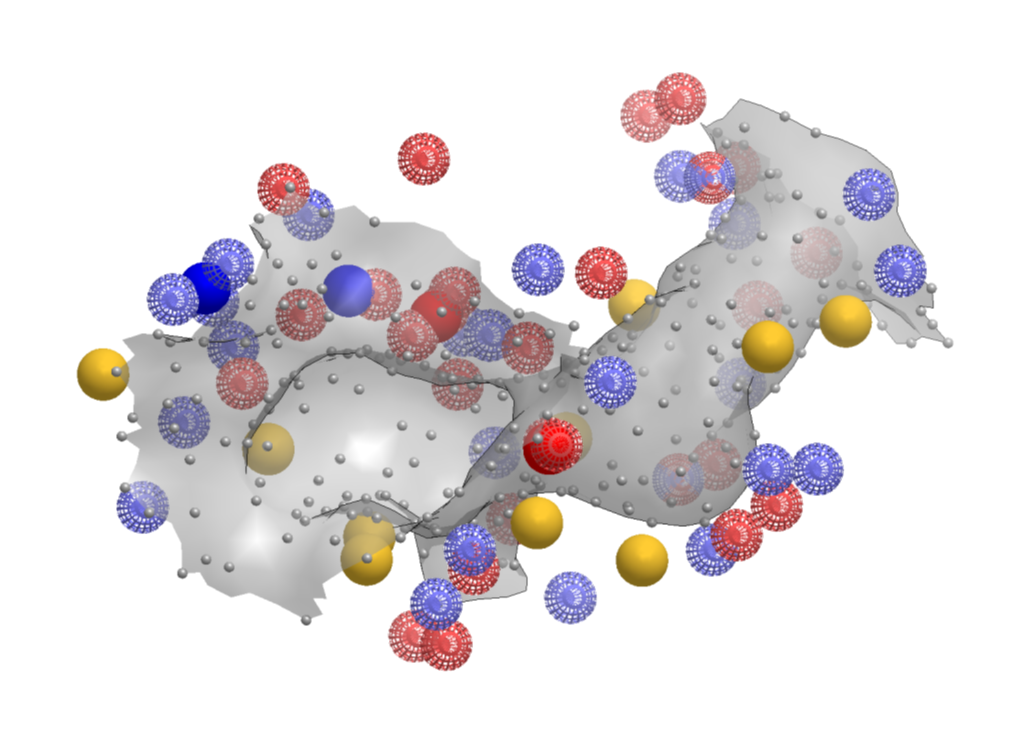
图2. SiteHopper结合位点模型(也称为补丁,“patch”)
SiteHopper依赖于通过多种方法获得的结合位点的表面来定义。结合位点的表面用碳原子装饰以提供3D表面表示,然后根据表面下方活性位点残基的化学特征来添加颜色点。然后将结合位点的这种完全三维表示用于结合位点的成对叠合并对其相似性进行打分。SiteHopper的算法如图3所示。
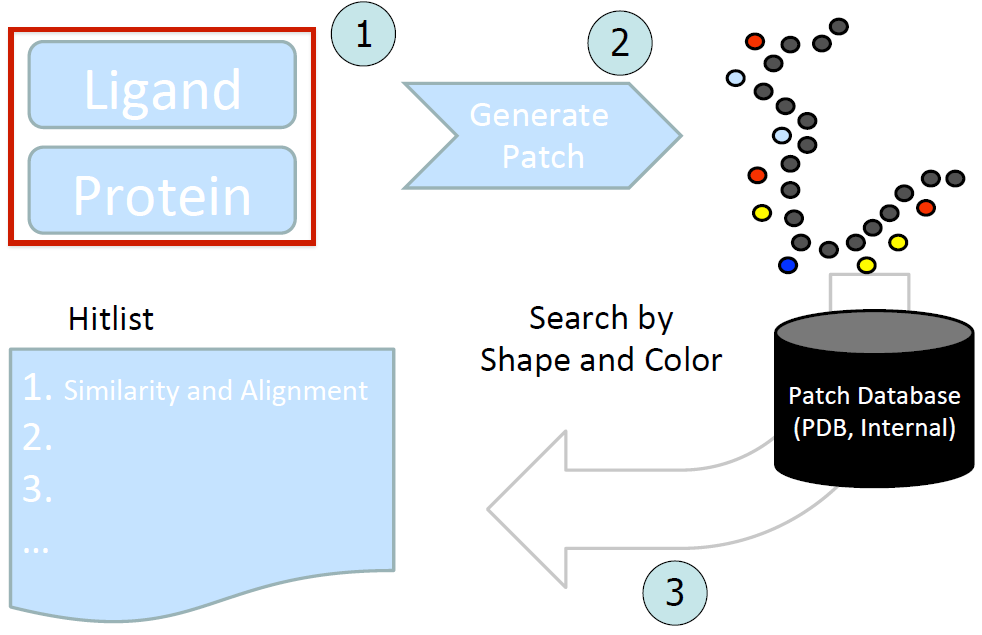
图3. SiteHopper的算法概述
SiteHopper算法中的叠合打分过程如图4所示:将数据库中的每一个补丁叠合到查询式补丁上,叠合根据补丁的形状与颜色(受体、供体与疏水性)特征最大相似性进行。叠合的质量用打分值,即形状叠合与颜色叠合的和,来定量表征。最后,用打分值对数据库补丁进行排序。
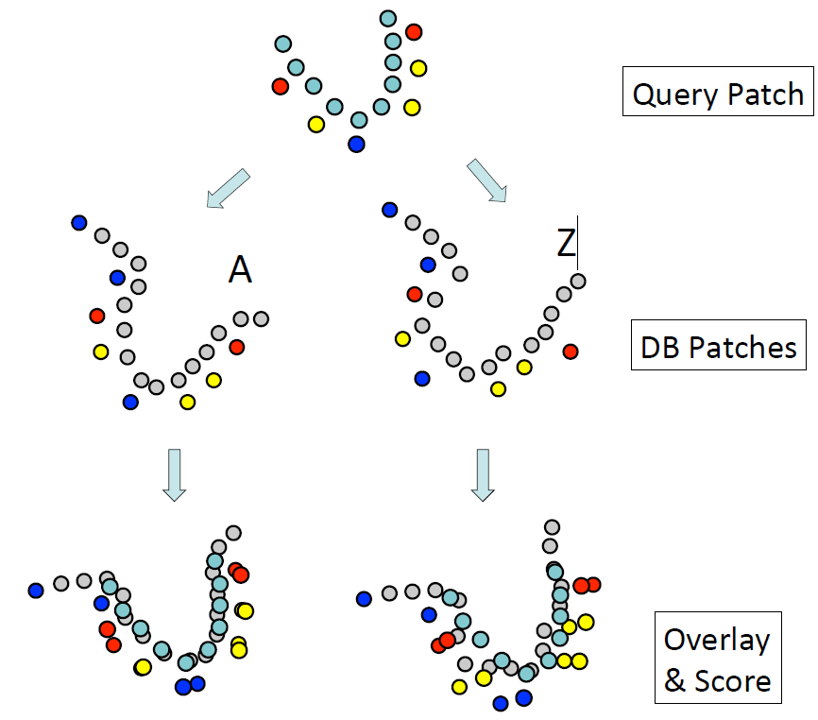
图4 图3步骤3的叠合、打分过程示意图。基于补丁的形状与颜色(受体、供体与疏水性)特征将数据库中的每一个补丁叠合到查询式补丁上,叠合的质量用打分值(为形状叠合与颜色叠合的和)来定量表征。最后,用打分值对数据库补丁进行排序。
结果
用SiteHopper分析激酶数据库能够从低序列相似性蛋白中找到相似的结合位点。一个例子如图5所示,相同的抑制剂与两种具有相似亲和力的激酶Wee-1和Traf-2结合,这意味着一个相似的结合位点。结合位点序列相似性较低(小于45%),而SiteHopper相似性非常显著(在1000次比较中出现小于3次)。
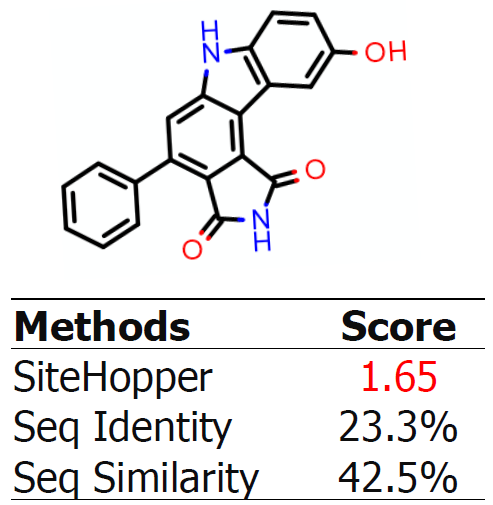
图5. SiteHopper检测到Wee-1与Traf-2激酶具有相似的结合位点,但是系列比对检测不到。
通过SiteHopper打分对丝氨酸蛋白酶数据库(从PDB提取)进行分层聚类,几乎精确地重现了由序列定义的亚家族之间的关系。然而,对于FXa分支,一个胰蛋白酶结构被聚类在因子Xa家族中,这看起来像是一个错误。文献检索显示,该胰蛋白酶结构是一个以重建FXa结合位点为目的理性突变项目的实验对象[1]。如预期的那样,所得到的突变胰蛋白酶(PDB代码1J17)具有与FXa非常相似的结合位点,而其序列几乎完全是胰蛋白酶的序列。
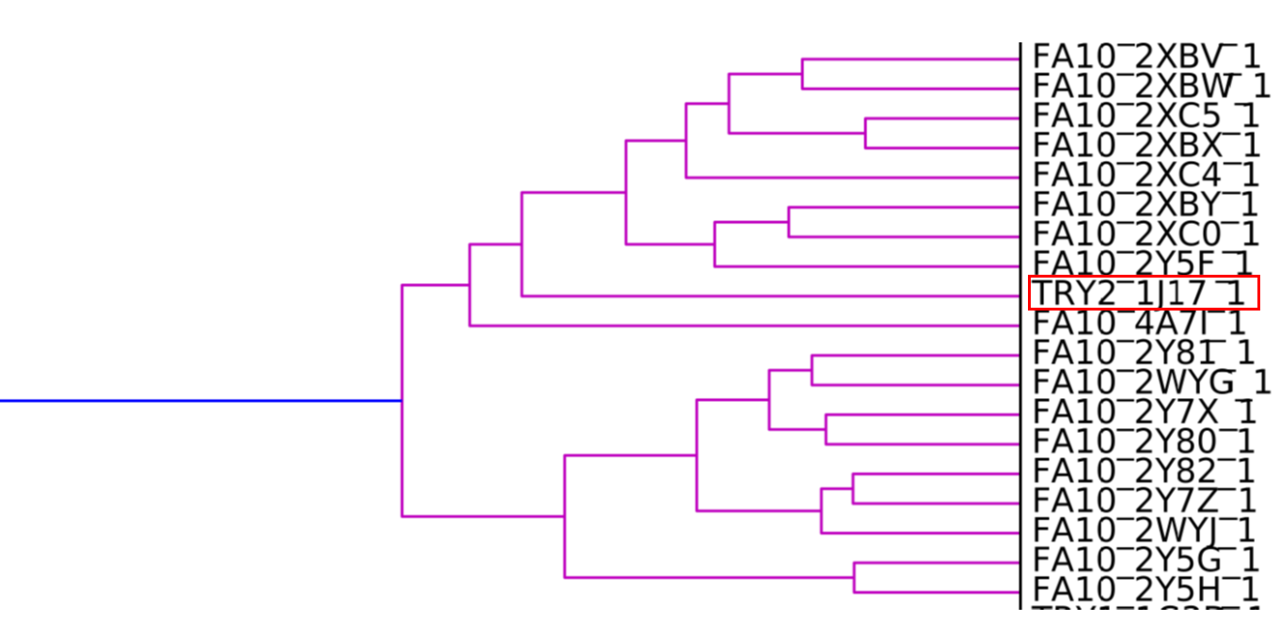
图6. FXa的SiteHopper分级聚类。胰蛋白酶结构所代表的突变型FXa结合位点用红框高亮显示。
最近一项研究比较了序列相似和SiteHopper相似性用于预测配体对激酶的选择性[2]。预测跨靶标配体结合的统计模型是采用高斯过程建模[3]对大约120种这些激酶的逐项相似矩阵模拟产生的。然后使用该模型来预测另外180个激酶的配体对原来120个激酶的结合。研究发现,在预测这些新配体的结合方面,SiteHopper相似性在统计学上显著地优于序列相似性[4]。
结论
SiteHopper是一个非常灵活的结合位点比较工具。它用3D形状与颜色特征来表示结合位点,因此独立于蛋白序列。早期的研究已经证明它可用于多重药理预测,尤其是这些蛋白序列相似很低的时候。
文献
- Reyda, S.; Sohn, C.; Klebe, G.; Rall, K.; Ullmann, D.; Jakubke, H. D.; Stubbs, M. T. Reconstructing the Binding Site of Factor Xa in Trypsin Reveals Ligand-Induced Structural Plasticity. J. Mol. Biol. 2003, 325 (5), 963–977. https://doi.org/10.1016/S0022-2836(02)01337-2.
- Dranchak, P.; MacArthur, R.; Guha, R.; Zuercher, W. J.; Drewry, D. H.; Auld, D. S.; Inglese, J. Profile of the GSK Published Protein Kinase Inhibitor Set Across ATP-Dependent and-Independent Luciferases: Implications for Reporter-Gene Assays. PLoS One 2013, 8 (3), 1–9. https://doi.org/10.1371/journal.pone.0057888.
- Gaussian Processes for Machine Learning.Rasmussen & Williams. MIT Press, 2006.
- Warren et al., manuscript in preparation.



