摘要:本次先睹为快介绍了即将发布的Flare™中两个药化相关的新功能:视频制作(屏幕录制)与R基团分析。Flare灵活的用户界面使得药物化学家能够在药物发现项目中设计新的配体并进行优先性排序。
本次先睹为快介绍了即将发布的Flare™中的一些新功能,Flare灵活的用户界面使得药物化学家能够在药物发现项目中设计新的配体并进行优先性排序。
视频制作
如果一张照片值一千字,那把它制作成视频会更好。
在“展示”比“讲述”更好或更快的情况下,视频都是无价的。例如,图1中的短片展示了如何在不到一分钟的时间内做到:
- 从RCSB下载PDB:1OIT蛋白
- 进行结构准备
- 将视图聚焦在活动位点上
- 以3D形式显示配体-蛋白相互作用
- 将2D接触显示为相互作用图
- 在3D窗口和相互作用图中自定义接触
- 添加渐变的彩色背景
图 1 短视频展示如何创建PDB 1OIT活性位点的自定义视图,以2D和 3D方式展示主要的配体-蛋白相互作用。
屏幕录制的配置非常灵活,您可以选择每秒帧数、窗口分辨率,以及是录制整个Flare窗口、仅录制主3D显示窗口,还是录制全屏(图 2)。
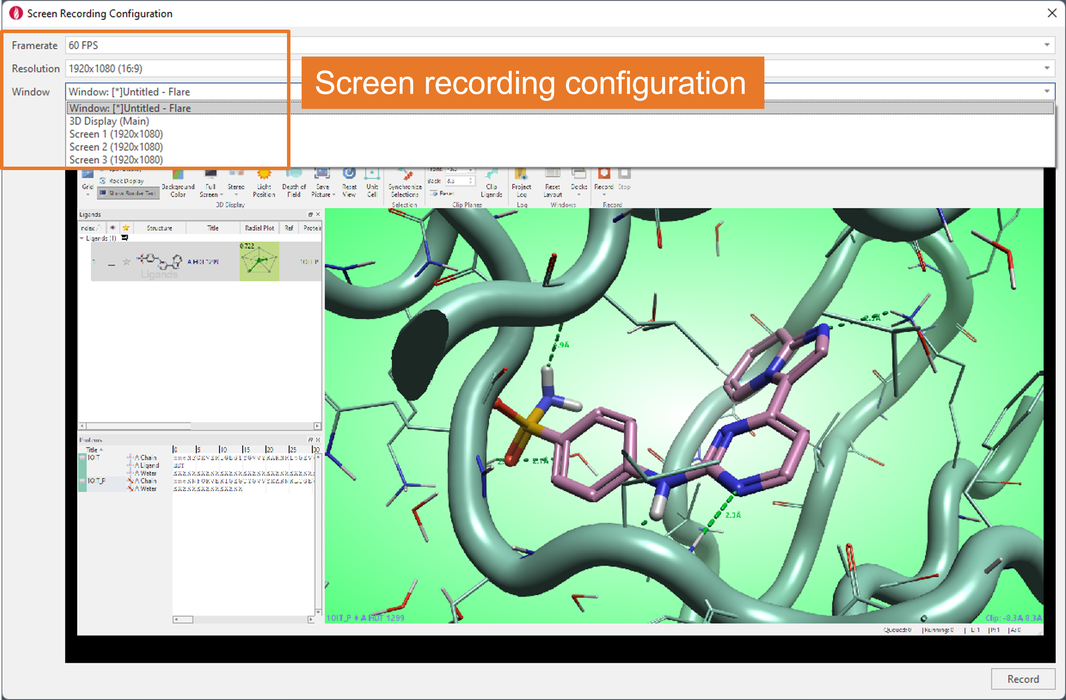
图2 屏幕录制按钮配置非常灵活
R基团分析
Flare V6.1将引入基于RDKit分解方法的R-基团分析 (R-group analysis,RGA)。该方法可用于快速分析具有公共母核的配体系列,探索取代基变化如何影响活性以及关键理化性质,并识别化学探索策略中潜在的空白区间。
本文用这种方法快速地解了来自Waaler等人的84个Tankyrase 2 (TNKS2) 抑制剂数据集的SAR。这一系列配体通过与生物靶标形成复杂的氢键网络和芳香族相互作用与TNKS2活性位点相互作用(图 3)。
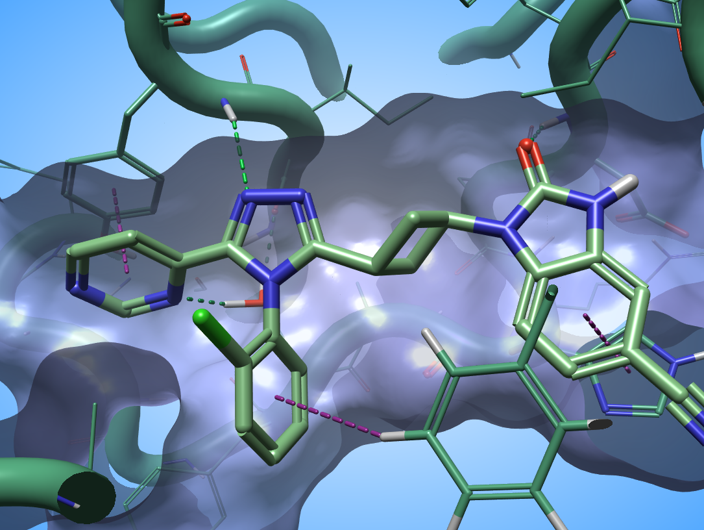
图3. PDB 5NOB的配体通过形成复杂氢键网络和芳香-芳香相互作用与 TNKS2的活性位点相互作用。 绿色虚线:氢键; 紫色虚线:芳香-芳香相互作用。
RGA使用图4-左所示的“母核”结构进行的,以探索改变R1、R2和R3位置上取代基(图 4-右)对TNKS2活性的影响。
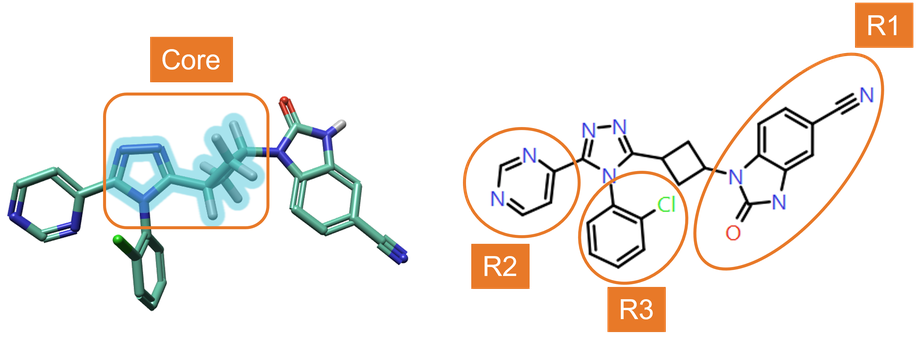
图4. 左:用于R基团分析(RGA)的母核结构。右:RGA发现的R基团编号
结果显示在图 5 中的“R基团析结果”表单中,表单概述了在每个配体的每个连接点上所发现的取代基,以及相关属性。
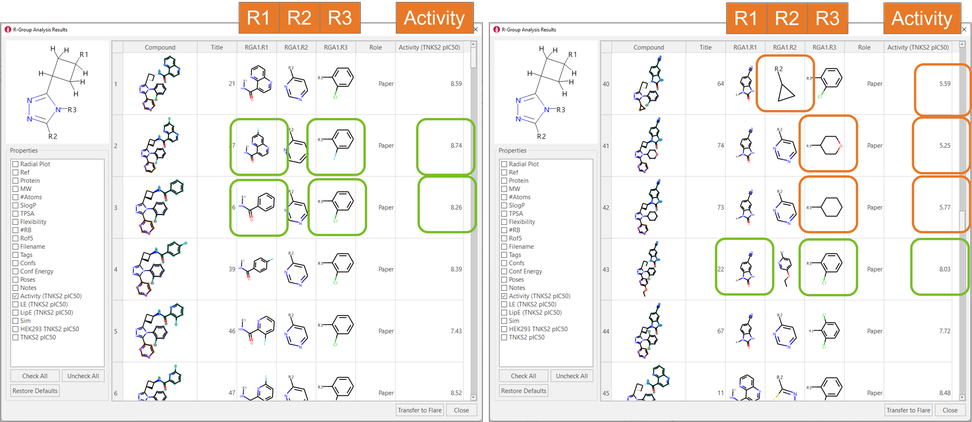
图5. R基团分析结果表单。绿色框:与更高生物活性相关的R基团。 橙色框:R2和R3不允许的脂肪族取代基。
快速检查RGA表可立即获得有关该配体系列SAR的有用见解。例如,在 R1位置,带有单环或双环芳香取代基的酰胺通常与良好的TNKS2 pIC50相关(图4-左):苯并咪唑酮也很好(图4-右)。R3中的2-Cl-苯基和2-F-苯基都经常与良好的TNKS2 pIC50 相关。 最后,R2和R3位上引入脂肪族取代基对生物活性不利。
箱线图和热图为该配体系列的SAR提供了额外的有用见解。
R3和R2 的箱线图(图 6)证实需要在这些位置需要引入芳香族取代基以实现高的生物活性。对于R2,在所有活性最好的化合物中,都具有邻位芳香N。这一经验证据与这些配体的结晶结合模式是一致的(参见图 3)。

图6. R3(左)和R2(右)的箱线图证实脂族取代基在这些位置上不能很好地耐受。
R1箱线图表明,在该位置上已经成功地尝试了更多种类的取代基:但是,叔酰胺、小脂肪族酰胺和芳香族取代基不直接连接到羰基上的酰胺是不容许的(图 7)。
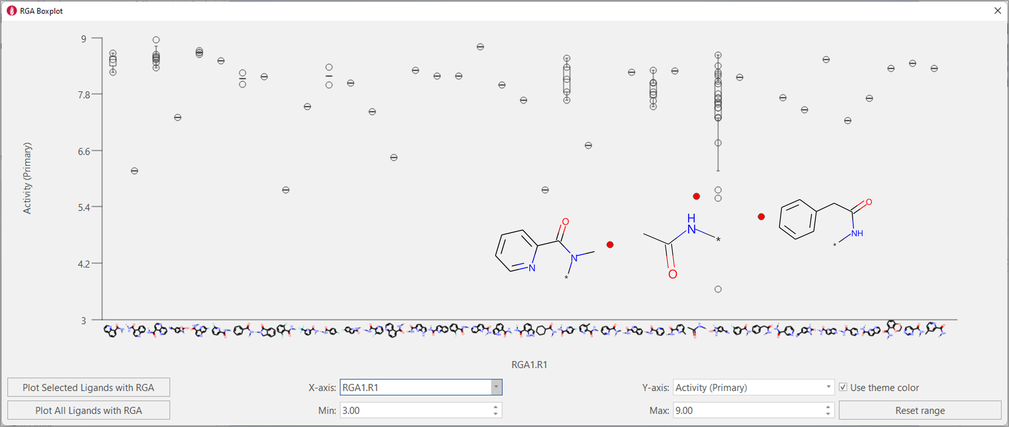
图7. R1箱线图。 已经在该位置尝试了多种芳族酰胺,然而有些变化对活性不利。
最后,热图不仅能够分析一次改变两个取代基对生物活性的影响,还能够识别化学探索中的潜在空白区,即未尝试过的有利取代基的组合。例如,图8显示了R2 = 2-Cl-苯基/R3 = 5-Mesyl-2-吡啶的组合从未尝试过,尽管该R3与R2 = 2-F-苯基组合与高生物活性相关。
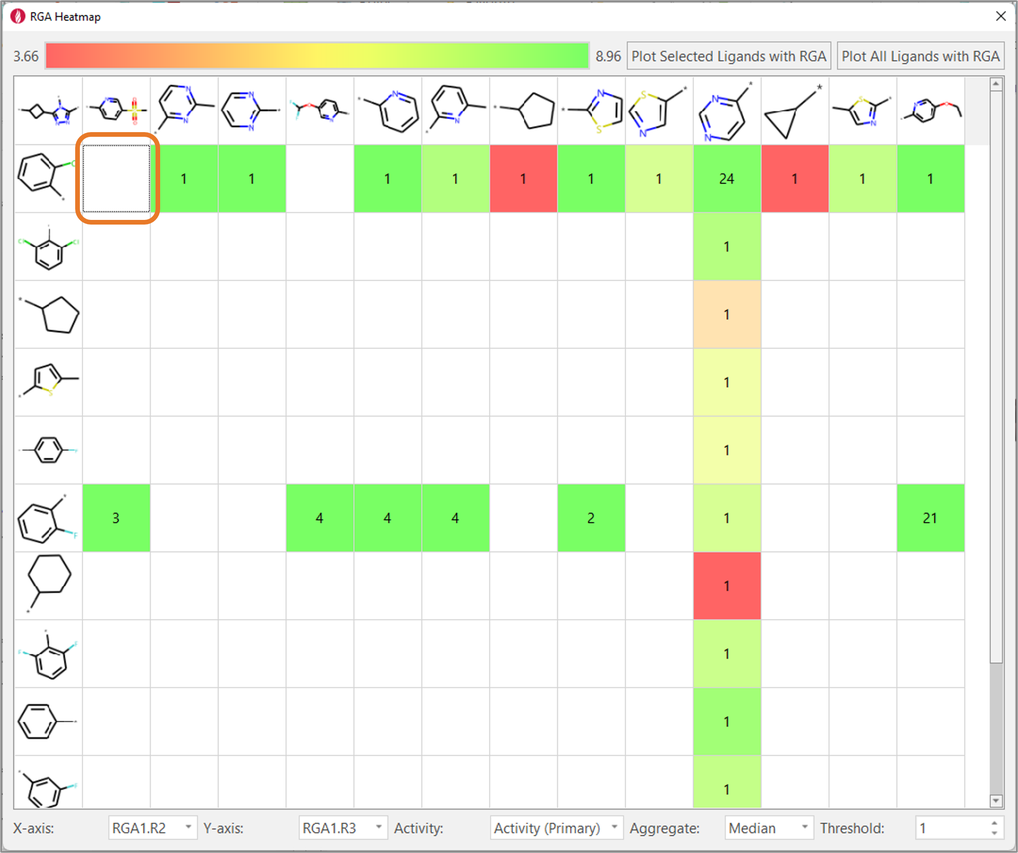
图8. 热图显示了一次改变两个取代基对感兴趣的配体性质(在本算例中为TNKS2的pIC50)的影响。它们也有助于发现化学探索中的空白:例如,这个配体系列从未被尝试过橙色方框中的组合。



