摘要:SPARK的分子大环化向导是一种快速且易于使用的工作流,可生成有意义且结构多样的计算结果,用于指导大环药物的发现。本文以一个非环的HPK1抑制剂为起点分子,用SPARK大环化实验成功地设计出与文献报道的大环HPK1抑制剂,复现了Wang等人从BMS的化合物4开始到大环5系列化合物的发现过程。
肖高铿/2023-02-23
前言
分子的大环化(Macrocyclization)有诸多优势,比如提供多样的官能团、改善类药性(尤其是多肽药物的大环化拟似物)、增加选择性1。分子大环化可以将分子锁定于一个低能结合构象、并减少结合熵变损失,因此可以用于提高化合物的结合亲和力2–4。分子大环化的难点在于分子的合成可行性,因此在合成前预测分子大环化策略的有效性是大环药物发现成功的关键。
最近,华中师范大学的Wang等人4报道了新的大环HPK1抑制剂,在酶学水平和小鼠模型中显示出抗癌潜力4 。Wang等人4从BMS报道的化合物4开始,采用基于结构的设计并进行大环化得到活性小于1纳摩尔水平的5系列化合物(图1),进一步对代谢稳定性优化之后,最终实现结合亲和力和药代动力学性质得到改善的化合物5i。
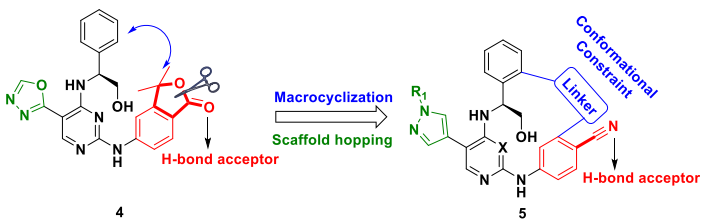
图1. Wang等人从BMS的HPK1抑制剂4出发进行侧链替换、大环化之后得到新的大环化合物5系列。
在本中,使用SPARK5–Cresset的生物电子等排体搜索和替换工具来设计大环化策略,并根据Wang等人4报道的实验数据来评估设计结果。
分子大环化的方法
SPARK利用具有相似静电和空间特性的片段来替换给定起点分子(starter)中的特定部分6, 其中静电和空间的相似性比较是基于XED力场计算得到的分子场来计算的7-9。SPARK的一个主要好处是它在搜索替换片段时保留了3D信息:考虑了整体分子的电子和构象效应,不是仅仅对相应的替换片段进行评分,而是对整个最终分子进行评分。替换片段是从SPARK的片段数据库中筛选出来的,这些数据库又是从可供商品销售的化合物数据库或文献报道的化合物生成的。当一个片段被识别为含有所需数量的连接点并具有与起点分子相似的形状/几何形状的片段之后,SPARK会使用XED力场对分子进行能量最小化计算以去除不良的碰撞和不利的几何形状,然后再进行最终分子的场计算和相似性优化以得出最终的打分值。
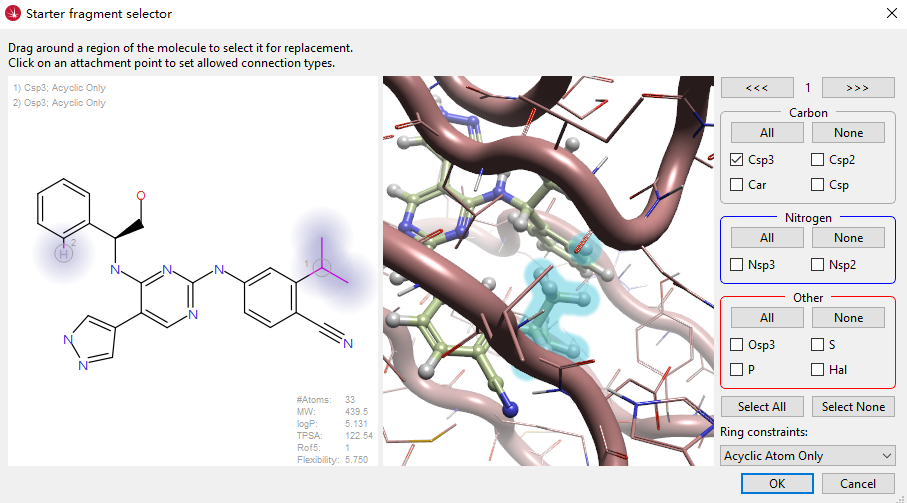
图2. 大环分子设计的起点分子以及在分子大环化向导里选择连接点。左边高亮部分是连接点:连接点1是苯环上腈基邻位的异丙基,连接点2是另一苯环2位氢原子。右边的3D视窗可以实时观察连接点以确保连接点的选择是正确的。
HPK1大环抑制剂的设计采用SPARK的分子大环化向导来实现。向导模块提供了快速且易于使用的图形界面工作流,包含了专为分子大环化设计而量身定制的专用参数设置(图2)。 在本实验中,我们使用了HPK1与非大环化合物4的复合物晶体结构(PDB 7KAC),并对复合物中的化合物4在结合位点内进行修改之后作为起点分子(图2)。
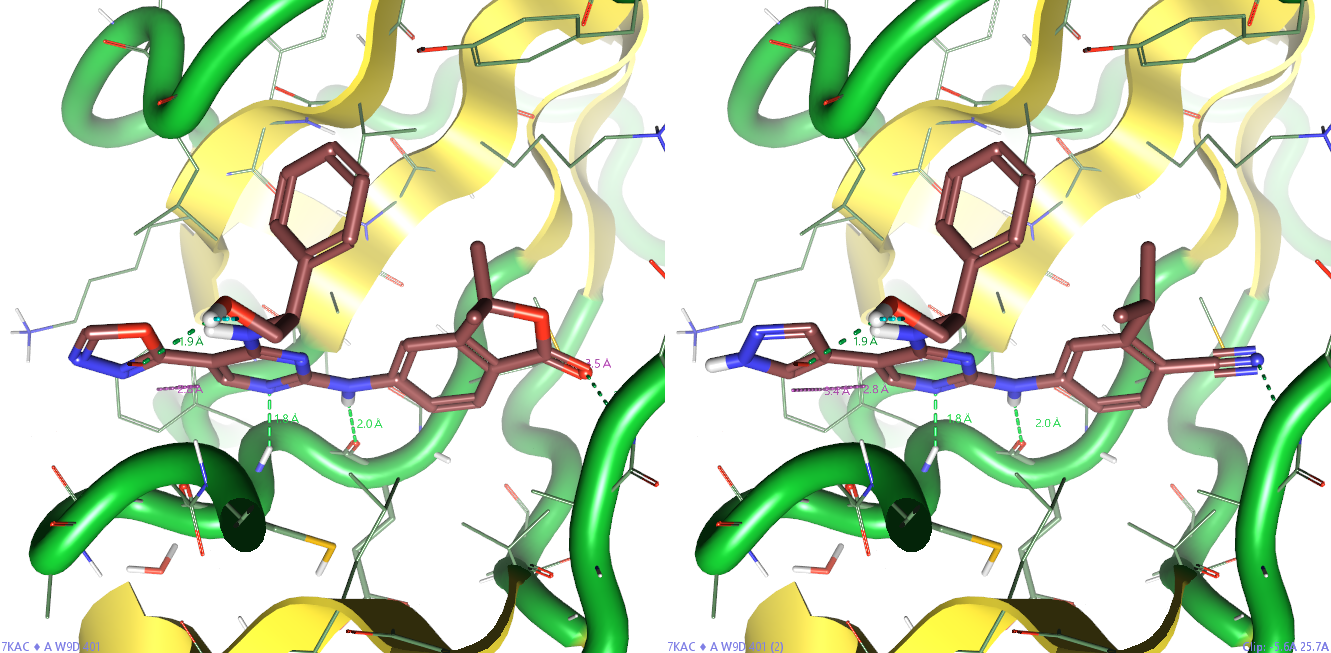
图3. 化合物4(左)以及起点分子(右)与靶标HPK1的复合物结构(PDB 7KAC)。其中,起点分子是根据化合物4在结合口袋里修改优化而来
起点分子根据Wang等人[4]的初步优化而设计:将五元内酯环开环并引入腈基,将1,3,4-噁二唑用吡唑替换。起点分子与化合物4具有一样的生物活性构象,如图3所示,连接点1和连接点2选择一方面依据Wang等人[4]的5系列化合物,另一方面是因为这两个原子在空间上紧密靠近。
为了让连接臂的设计偏向于已有的化合物,将sp3杂化碳指定为连接点1的连接原子,将sp3杂化的氧指定为连接点2的连接原子,这两个连接原子均为非环原子。此外,还将受体结构设置为排除体积来指导连接臂设计。
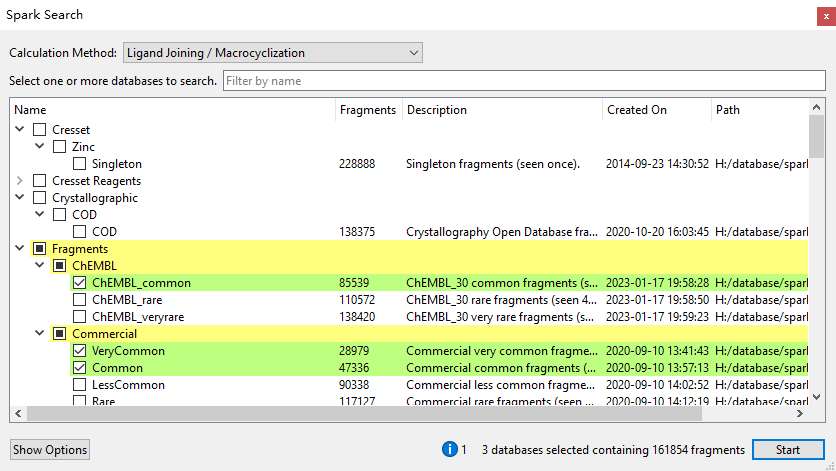
图4. 大环设计使用的片段数据库
该实验使用专用的“Ligand Joining / Macrocyclization”设置参数对片段数据库CHEMBL_common、Commercial_Common、Commercial_VeryCommon、Commercial_LessCommon、SureChEML_verycommon以及COD(图4)进行搜索。 最后将SPARK计算获得的结果导入到Flare10中进行可视化分析。
结果
本研究共命中760个化合物(连接臂,linker),按Bemis-Murcko clustering分析总共包含252种不同化学类型的连接臂。如图5所示,Wang等人4报道的5个连接臂类型及典型化合物(5a-e)全部被Spark大环化计算实验设计出来。
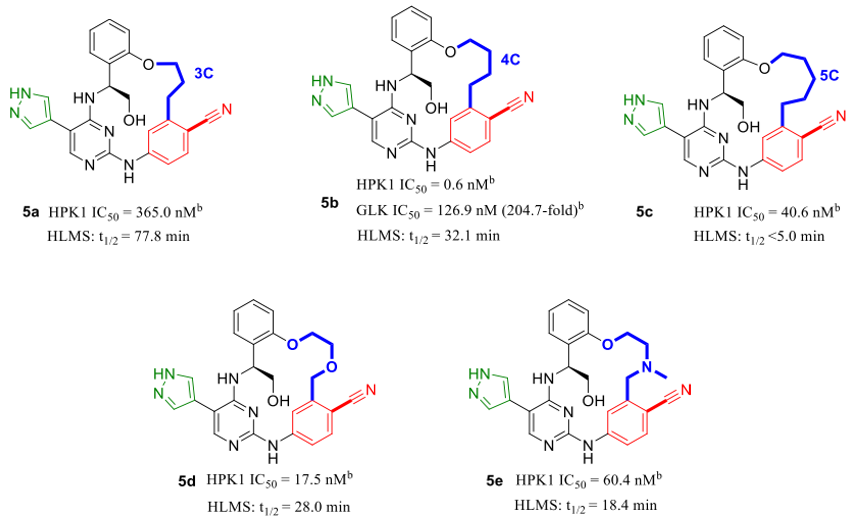
图5. Wang等人4报道的连接臂类型与化合物5a-e
SPARK命中的化合物5a-e以及各自所属的Linker类别按打分值排序如表1所示。可以看出,5个连接臂类别排序都比较靠前,尽管命中的化合物打分排序部分并不靠前,但是并不影响你关注到这些聚类排序靠前的连接臂,并通过简单的排除而关注到具体的化合物5a-e。
表1. Spark大环设计重复出Wang等人4报道的连接臂类型、化合物与排序
| Linker类别 | Cluster Rank | NC | Hit | Hit Rank |
|---|---|---|---|---|
| 3C | 5 | 142 | 5a | 33 |
| 4C | 7 | 36 | 5b | 525 |
| 5C | 25 | 4 | 5c | 52 |
| Ether | 54 | 16 | 5d | 156 |
| Amine | 72 | 3 | 5e | 602 |
NC: the number of molecules in the cluster
将SPARK结果里导出给Flare,进一步对命中的化合物进行分析。Flare支持多种基于结构与基于配体的分析方法。比如静电互补性,构象稳定性,两面角频次分布概率,相互作用分析等等。图5展示了5a-e在BMS化合物4与HPK1共晶结构(PDB 7KAC)结合位点里的结合模式,5a-e与化合物4具有相似形状与相似的相互作用模式。

图5. Spark大环化实验得到的化合物5a-e的3D结合模式
既然Spark设计的高分化合物可以重现Wang等人4报道的大环HPK1抑制剂,想必其它高分但没有文献报道的化合物应该也具有HPK1抑制剂的潜力。
结轮
本案例研究表明,以一个非环的HPK1抑制剂为起点,SPARK可以成功地设计出与文献报道的大环HPK1抑制剂相同的化合物。SPARK的分子大环化向导是一种快速且易于使用的工作流,可生成有意义且结构多样的计算结果,用于指导大环药物的发现。
文献
- Wagner, V.; Rarey, M; Christ, C.D. Computational Macrocyclization: From de Novo Macrocycle Generation to Binding Affinity Estimation. ChemMedChem 2017, 12 (22), 1866–1872.
- Yu, X.; Sun, D. Macrocyclic Drugs and Synthetic Methodologies toward Macrocycles. Molecules 2013.
- Wang, L.; McDaniel, K. F.; Kati, W. M. Fragment-Based, Structure-Enabled Discovery of Novel Pyridones and Pyridone Macrocycles as Potent Bromodomain and Extra-Terminal Domain (BET) Family Bromodomain Inhibitors. J. Med. Chem. 2017, 60 (9), 3828–3850.
- Wang, M.-S.; Wang, Z.; Li, Z.; Gong, Y.; Duan, C.; Cheng, Q.; Huang, W.; Yang, G. Discovery of Macrocycle-Based HPK1 Inhibitors for T-Cell-Based Immunotherapy. J. Med. Chem. 2023, 66 (1), 611–626. https://doi.org/10.1021/acs.jmedchem.2c01551.
- https://www.cresset-group.com/software/spark
- Tosco, P.; Mackey, M. Lessons and Successes in the Use of Molecular Fields. In Comprehensive Medicinal Chemistry III; Elsevier, 2017; pp 253–296.
- Vinter, J. G. Extended Electron Distributions Applied to the Molecular Mechanics of Some Intermolecular Interactions. J Comput Aided Mol Des 1994, 8 (6), 653–668.
- Cheeseright, T.; Mackey, M.; Rose, S.; Vinter, A. Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. Chem. Inf. Model. 2006, 46 (2), 665–676.
- Vinter, J. G. Extended Electron Distributions Applied to the Molecular Mechanics of Some Intermolecular Interactions. II. Organic Complexes. J Comput Aided Mol Des 1996, 10 (5), 417–426.
- http://www.cresset-group.com/software/flare



