摘要:对药化科研人员和计算化学专家来说,在3D可视化工具中摆放蛋白结构是一个简单且必要的操作,用于蛋白-配体结合位点的叠合、与比较,常用的方法有两种:第一种是根据蛋白序列进行叠合,配体跟随着蛋白叠合到另一个配体上;第二种是根据配体3D信息进行叠合,蛋白跟着配体叠合到另一个蛋白上。在本中,我们的应用科学家Scott Midgley概述了如何使用Flare Python API实现第二种蛋白叠合方法,即基于配体的原子叠合来调整蛋白的取向。
作者:Scott Midgley/2023-04-13
编译:肖高铿
在3D可视化时蛋白-配体取向的重要性
对药化科研人员和计算化学专家来说,在3D可视化工具中摆放蛋白结构是一个简单且必要的操作。在药物发现早期的苗头化合物与先导化合物发现阶段,计算工作往往依赖于一系列结合的小分子配体与其靶标活性位点之间相互作用的清晰视觉表征。
Flare的配体叠合(Ligand alignment)采用Cresset专有的XED力场来描述分子以进行快速、高效地3D配体叠合以及传统的基于最大公共子结构的配体叠合。此外,还可以简单地根据选取的原子进行配体叠合。在蛋白背景下处理配体时,将两个或多个共晶的配体叠合之后,让蛋白结构跟随着与其结合的共晶配体平移过来,这种蛋白叠合方式常常具有独到的好处。这种方法得到的蛋白叠合视图不是通过蛋白序列比对来实现,而是通过叠合它们的共晶配体来实现。
尽管Flare没有原生的能力根据共晶配体的叠合来摆放蛋白取向,但是Flare Python API允许用户通过Python模块访问Flare的大部分功能来创建自定义功能。这样做的好处是,除了通过图形用户界面(GUI)访问现有功能外,还能对应用提供更高的控制程度。用户可以创建高通量的工作流,或者使用这些功能来创建自定义工具。后者是本文的重点,我们展示了如何使用Flare Python API脚本来扩展基本应用的功能。
在此我们提出一种简单的方法,使用Flare Python API编写的脚本,采用基于原子的配体叠合来调整蛋白的取向。
用配体叠合来重新摆放蛋白的取向
在Flare中从PDB上下载蛋白4QAC作为算例,并进行结构准备。这是一个桶状蛋白,由10个生物多聚体组成,其单体适合在Flare中进行叠合。一旦蛋白结构准备完成,删除除了A、 B链之外的所有链,同时在Flare配体表中删除关联的配体。然后,将B链分离出来作为一个独立的蛋白,在Flare蛋白表单中得到两个对象:一个由原来的A链组成,另一个由原来的B链组成,如图1所示。
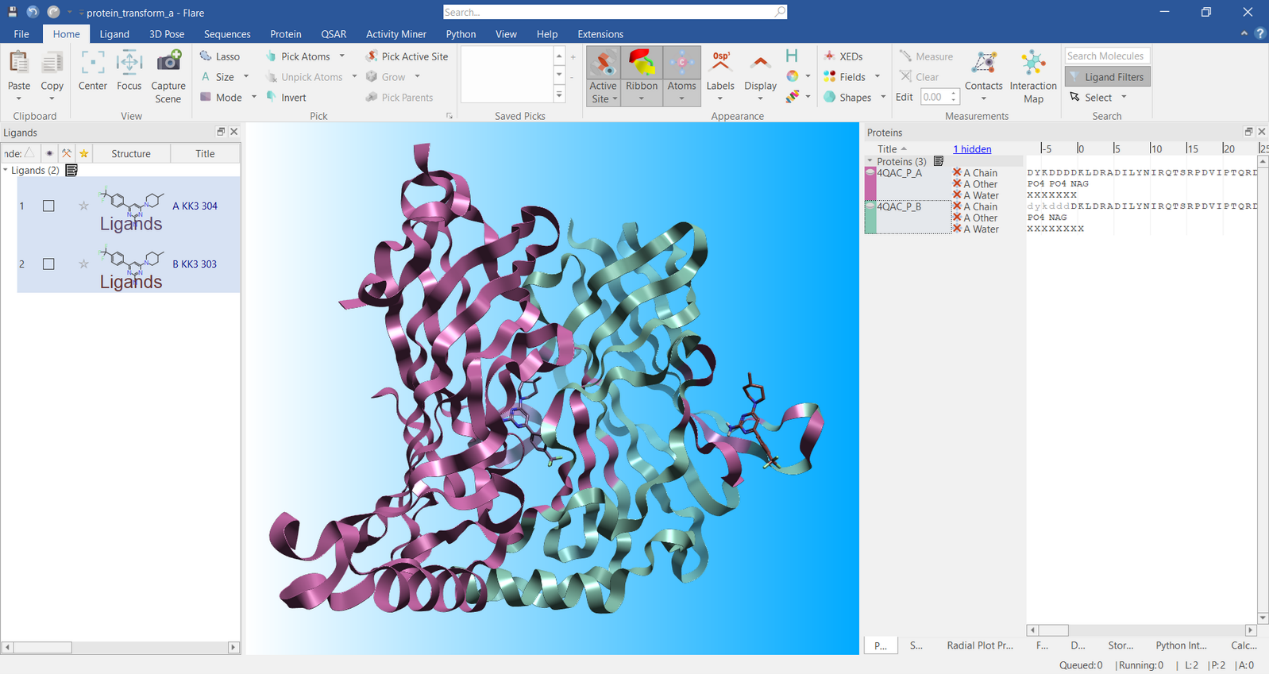
图1. 在Flare中蛋白A、B链的可视化
为了开始叠合配体,首先使用键盘快捷键 “Shift+i “显示原子编号。图2显示了两个配体的原子编号,也证实了它们在空间上没有重叠在一起。Flare要求至少有三对原子来进行配体叠合,但是更多的原子对可以通过帮助定义分子的三维形状来使整体叠加效果更好。在这个例子中,我们使用10、8、16和24号原子来叠合配体,因为这些原子捕捉住了配体纵向和横向的几何形状。

图2. 在空间上分开的两个配体分子及其原子编号
一旦我们选定了用于叠合的原子编号,接着可以将transform_qt.py脚本(专为本练习而写)载入Flare Python解释器。选择 “Python “标签页,然后选择 “Python Interpreter “选项,然后点击解释器中的 “Load… “按钮,导航到该脚本即可。
在运行脚本之前,按住’Ctrl’键,点击所需的两个配体和两个蛋白,以便两个配体和两个蛋白被选中。然后点击 “Run “按钮来执行脚本;这会弹出一个文本框供用户输入参比分子和叠合分子的原子编号。
需要注意的是,虽然本例是在同一蛋白质的两份拷贝分子上进行的,但是在两个不同的蛋白质中叠加两个不同的配体时,这个脚本也能发挥作用。无论配体之间的原子编号是否相同,文本框中指定编号的原子都将在垂直对上进行匹配。例如,如图3所示,即使配体不同,参比分子中的原子10将始终与叠合分子中的原子10相匹配。因此,必须在参比原子框和叠合原子框中始终指定相同数量的原子,每个框中至少有三个原子编号。每个原子编号之间应该用一个空格隔开。
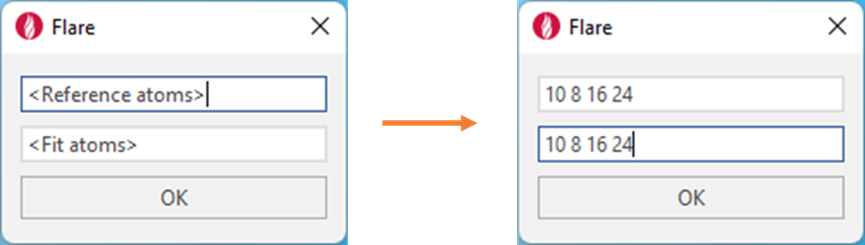
图3. 定义原子编号以使得参比分子与叠合分子能够匹配上
点击 “OK “按钮运行脚本,配体被叠合到指定的原子上。在配体叠加之后,紧接着使用与配体叠合相同的变换矩阵对蛋白进行叠合。变换的结果显示如图4所示。

图4. 通过配体叠合将两个算例蛋白进行坐标变换的可视化
总结
在Flare中可以通过共晶配体的叠合来调整蛋白的取向。在本文中,我们展示了如何对一个生物多聚体的两个单体实现这一功能。然而,该工作流也可以用来叠合不同的配体/配体-蛋白/蛋白对,前提是可以为配体的叠合确定合理的原子对。
联系我们
对于现有的Flare客户,如果想了解有关这一功能的更多信息,或者想知道如何获得本文中使用的Python脚本,请联系Cresset技术支持。如果你是Flare的新手,想更深入地探索该平台的全部功能,你可以申请免费试用版。



