摘要:为了支持靶向蛋白降解(Targeted Protein Degradation,TPD)疗法的研发,Cresset的骨架跃迁和生物等排体替换工具Spark发布了四个新的连接臂数据库。这些新数据库是从侯廷军课题组的PROTAC-DB库以及Enamine的Linker for Linkerology创建而来,总共包含9672个实验来源的连接臂,适合于靶向蛋白降解的研究。
Spark连接臂数据库发布
为了支持靶向蛋白降解(Targeted Protein Degradation,TPD)疗法的研发,Cresset的骨架跃迁和生物等排体替换工具Spark发布了四个新的连接臂(linker)数据库,见图1。这些新数据库是从侯廷军课题组的PROTAC-DB库1以及Enamine的Linker for Linkerology3创建而来。
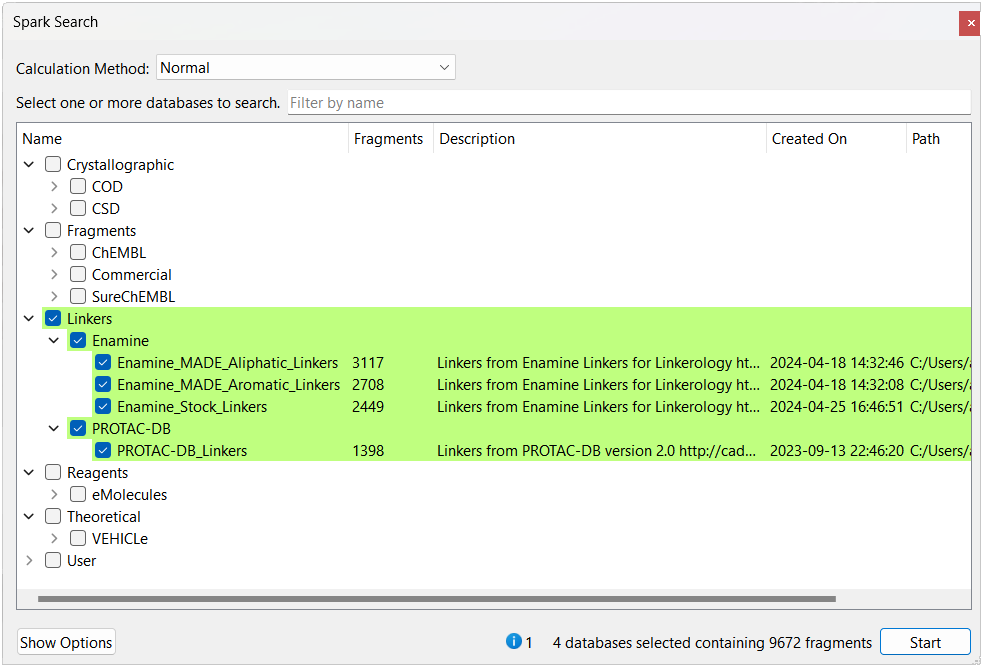
图1. Spark新增连接臂(Linker)数据库,包含9672个连接臂,适合于靶向蛋白降解研究。
在最近的一篇文章中,我们探讨了使用默认的Spark数据库(VeryCommon,Common和ChEMBL_common)以及新发布的Linker数据库(PROTAC-DB、Enamine Stock Linkers、Enamine MADE Aromatic和Enamine MADE Aliphatic),将Spark骨架跃迁实验应用于降解剂的设计(图2)。
结果证实了使用Spark连接臂集中库用来支持TPD研究的好处。虽然在默认Spark数据库中的161,854个可搜索片段中只发现了两个已知的连接臂,但在新数据库中仅对9,672个片段进行的搜索就检索到了10个已知TPD连接臂中的8个。
新的数据库包含了用于设计降解剂连接臂的专用片段。因此,这使得Spark是一种非常有前景的计算工具,不仅可用于高效的连接臂设计,还可探索靶向感兴趣靶标蛋白的配体设计,以及靶向E3连接酶配体的设计。
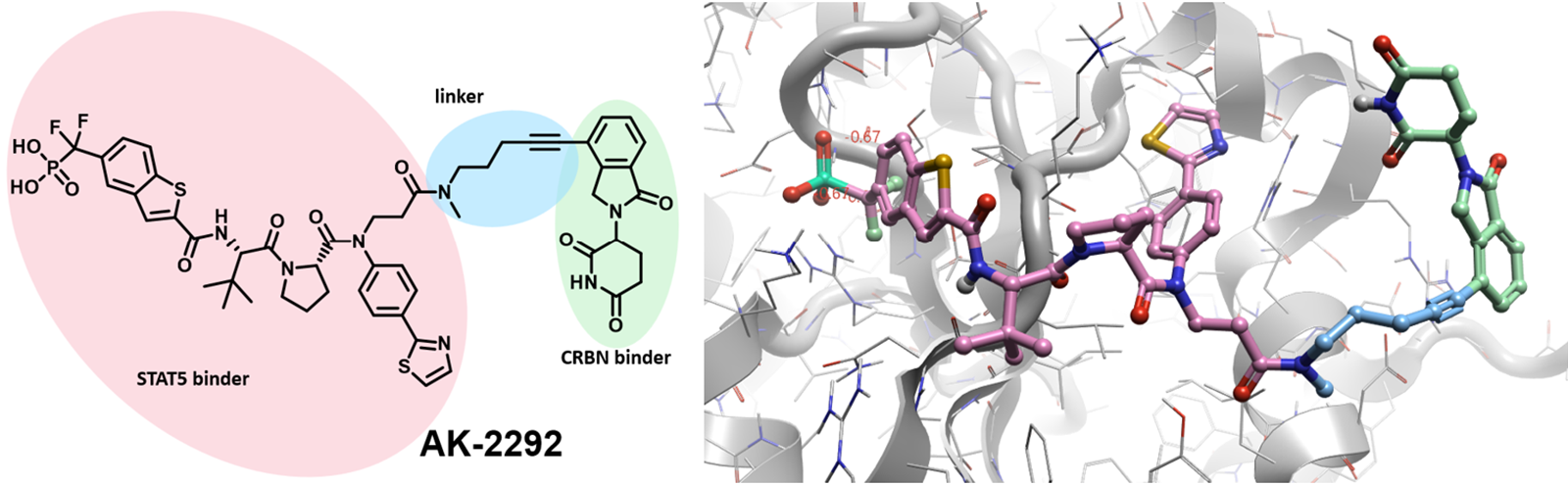
图2. Kaneshige A.等人3报道的降解剂AK-2292。在2D与3D图示中连接臂高亮为蓝色,PDB 7TVA。
新的连接臂数据库
新的连接臂数据库总共包括9600多个实验来源的、已知的和商业化的TPD连接臂。它们的性质总结在下表1中。
表1. Spark新的“连接臂”数据库的特性
| Database | Fragment | NC range | HA range | MW range | RB range |
|---|---|---|---|---|---|
| PROTAC-DB | 1398 | 1-9161 | 1-43 | 14-624 | 0-38 |
| Enamine Stock | 2449 | 1-2291 | 4-24 | 58-330 | 0-21 |
| Enamine MADE – Aromatic | 2708 | 1-2040 | 8-21 | 105-325 | 1-12 |
| Enamine MADE – Aliphatic | 3117 | 1-2482 | 4-22 | 58-324 | 0-21 |
片段(Fragment):数据库中的片段数量;NC:片段的构象数量;HA:片段的重原子数量;MW:片段分子量;RB:可旋转键的数量。
总体而言,新连接臂数据库中的片段与标准Spark数据库中的片段相比,其特征在于MW和RB范围明显更大,这反映在最大片段生成的构象数量上。
在你的TPD项目中使用Spark连接臂数据库
新的连接臂数据库扩展了目前已发布Spark数据库的庞大组合,这些数据库来源于商业上可供购买的筛选化合物、文献报道的化合物、专利公开的化合物、商业试剂、小分子晶体结构和理论环系统等等。Spark数据库提供了数以百万计的生物等排体以供搜索、产生新想法和推进项目。
新的连接臂数据库可立即供所有的Spark用户使用,并可以从Spark数据库更新程序下载(图3)。但是,搜索TPD集中库需要Spark V10.7.1,已与连接臂数据库一起发布。请联系我们索取Spark V10.7.1的下载链接。
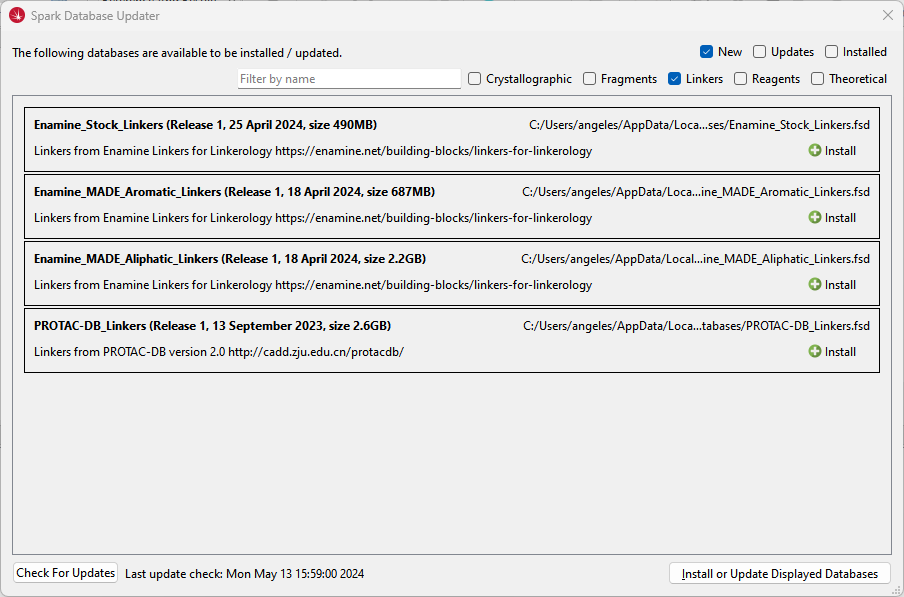
图3. 所有的Spark用户都可以用Spark升级器(Spark Database Updater)更新连接臂数据库,搜索该数据库需要使用最新的Spark V10.7.1版本,请联系我们获取下载链接。
对TPD连接臂设计和Spark新连接臂数据库的使用不感兴趣的现有客户可以继续使用之前发布的版本Spark V10.7,而不是必须更新。
文献
- http://cadd.zju.edu.cn/protacdb
- https://enamine.net/building-blocks-mob/linkers-for-linkerology
- Kaneshige, A. et al. (2023) “Discovery of a Potent and Selective STAT5 PROTAC Degrader with Strong Antitumor Activity In Vivo in Acute Myeloid Leukemia,” Journal of Medicinal Chemistry, 66(4). Available at: https://doi.org/10.1021/acs.jmedchem.2c01665.



