摘要:本文用Flare GIST分析了PDE10A的apo结合位点,用计算的水合位点与水合自由能解释了苗头化物与先导化合物优化过程中的SAR,回溯性地证明了Flare GIST在基于结构中指导分子设计的作用。根据GIST分析结果,选择感兴趣的修饰位置,用Hit Expander自动、快速地生成衍生物,实现对高能水进行置换的设计,其中包括文献报道活性提高30-300倍的设计,还包含尚未见文献报道的、另人感兴趣的设计。Flare GIST与Hit Expander联合使用是强大且可靠的基于结构的药物设计工具。
作者:肖高铿
日期:2024-05-17
前言
在从苗头到先导的优化过程中或先导化合物的优化通常会用口袋填充策略。当填充的空腔较小时,可以每次往配体上添加一个重原子进行细小的修饰。这种小修饰得当的话,可带来结合亲和力的巨大提升。比如,Merck公司在PDE10A抑制剂MK-8189的发现过程中,如图1所示,在苗头化合物9系列的噻唑环上引入甲基或甲氧基,带了活性高达30-300倍的提升1。
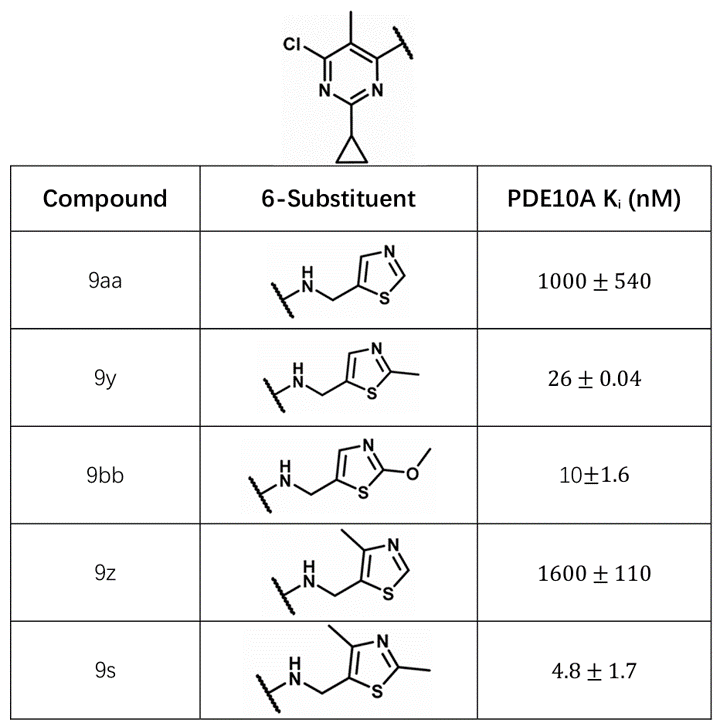
图1. PDE10A抑制剂9系列化合物及其Ki
在先导化合物优化时,如图2所示,在R1的吡啶环上引入甲基或甲氧基,也带来10倍的活性提升2。最终,化合物18(MK-8189)作为候选药物进入临床研究2。
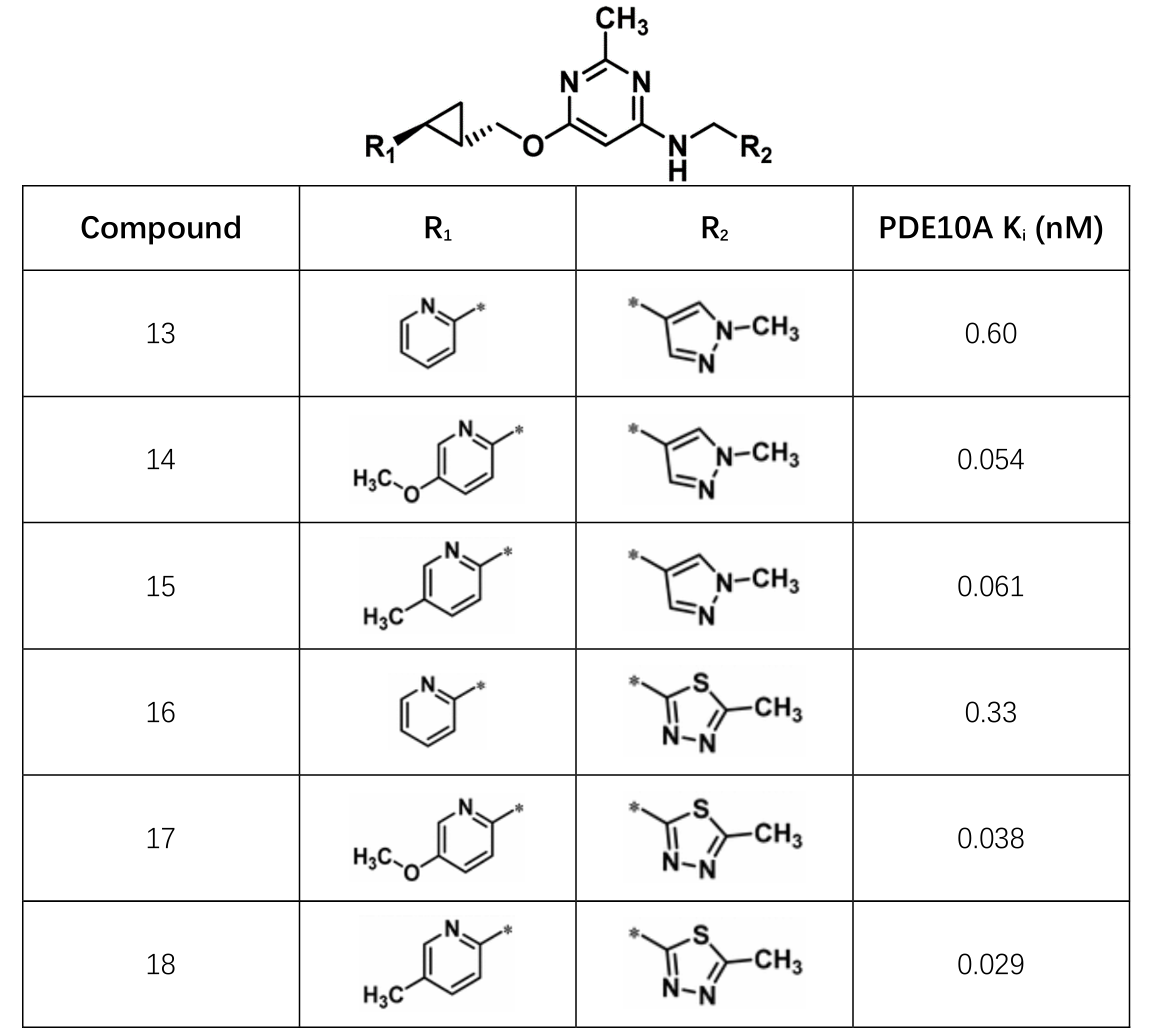
图2. PDE10A抑制剂13-18的化学结构式及其Ki
本文的目的是以上述的苗头化合物以及先导化合物的优化为例,演示如何使用计算机辅助药物设计平台Flare™3的水分析工具GIST4对PDE10A的结合口袋进行分析,并利用水合位点信息建立先导化合物优化策略;然后利用FlareTM的Hit Expander5对苗头化合物与先导化合物进行快速修饰、生成新的结构;最后选择满足优化策略的化合物进入下一轮测试。
结果
分析PDE10A的结合位点
图3. 化合物9h
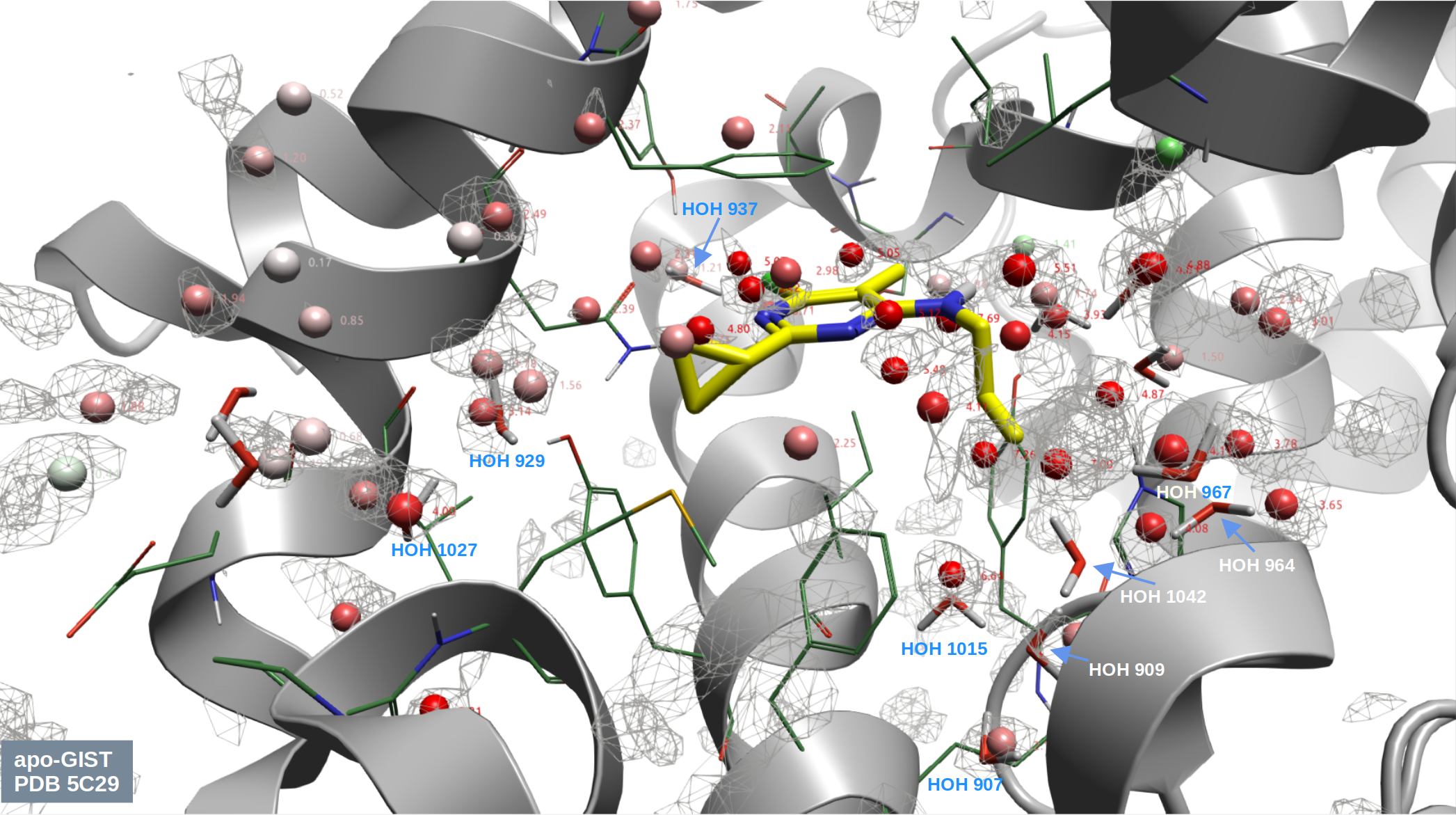
用Flare Pocket6分析化合物9h(图3)与PDE10A共晶结构(PDB 5C29)的结合位点,结果如图4所示。除了化合物9h占据的位置外,该空腔其余部分由12个水分子填充。具体而言,HOH1027和HOH927所处的子口袋对应于先导化合物系列13-18中R1基团的结合位点;而HOH1015、HOH909和HOH907所在的子口袋则与该系列中的R2基团以及片段苗头化合物9系列中6位噻唑取代基的结合相关。理解这些水分子在apo结构中的水合自由能对于基于结构的设计至关重要。
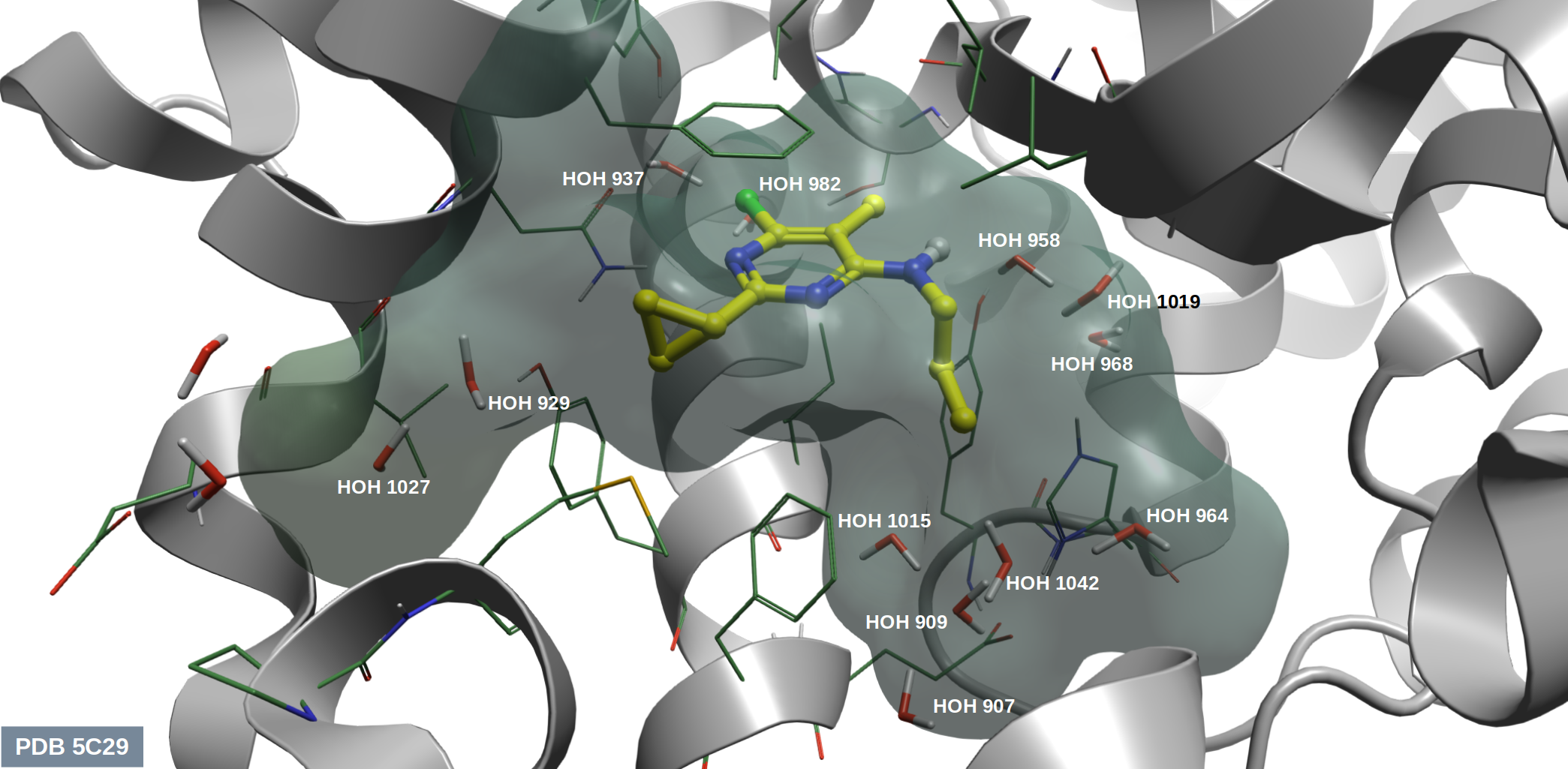
图4. 化合物9h与PDE10A的共晶结构PDB 5C29,口袋用Flare Pocket检测
PDE10A结合位点的apo-GIST分析
采用FlareTM GIST4在默认参数下分析PDB 5C29结合位点的apo结构,结果如图5所示。
图5. 化合物9h与PDE10A共晶结构PDB 5C29结合位点的apo-GIST分析结果。其中黄色化合物:9h;飘带:PDE10A;网格:GIST Water Density\(\ge\)4;棒状水:实验结合水;球状分子:预测的水合位点(红色:unhappy,绿色:happy)
GIST将水的性质表示为其空间位置的连续且平滑的函数,水合位点由水分子密度高的区域自然显现。观察图5的水密度图,可以发现,结合口袋里,几乎全部的水都被water density\(\ge\)4的等值图覆盖,这说明GIST预测的水合位点与实验值非常一致。
表1. PDB 5C29结合口袋的结合水与apo-GIST预测的水合位点比较
| Items | Expt. Water | Hydration Site | Distance(Å) |
|---|---|---|---|
| 1 | HOH1027 | W25 | 1.2 |
| 2 | HOH929 | W33 | 0.6 |
| 3 | HOH937 | W4 | 0.7 |
| 4 | HOH982 | W41 | 0.7 |
| 5 | HOH958 | W51 | 0.6 |
| 6 | HOH1019 | W20 | 1.2 |
| 7 | HOH968 | W1 | 0.7 |
| 8 | HOH1015 | W48 | 1.1 |
| 9 | HOH964 | W61 | 1.3 |
| 10 | HOH1042 | W61 | 1.5 |
| 11 | HOH909 | W19 | 1.3 |
| 12 | HOH907 | W5 | 0.6 |
水分子按图4从左到右、从上到下的顺序出现。
对水密度进行聚类分析可得到显式的水合位点(图5-球形分子),其颜色根据GIST计算的水合自由能进行着色:红色越深,能量越高;绿色越深,能量越低。几乎所有的水合位点都是红色的(Unhappy),因此这些水都是可被置/替换的。尤其注意到,结合口袋里的12个结晶水(图4)都有预测的水合位点与之重合,表1给出了它们之间的对应关系以及距离。最大的预测误差为1.5Å,这进一步证明了GIST水合位点预测的准确性。
13-18系列的SAR
将化合物18(MK-8189)与PDE10A的共晶结构PDB 8DI4按Cα原子叠合到9h与PDE10的共晶结构PDB 5C29,可以获得化合物18与PDE10A apo结合位点水的关系。如图6所示,可以发现化合物13-18系列(图2)的R1基团与HOH929、1027重合,这意味R1的芳香基团与甲基将这两个水置换;同样,我们也清晰的看到R2基团(见图2)与HOH1015重合,甲基置换了HOH1015。
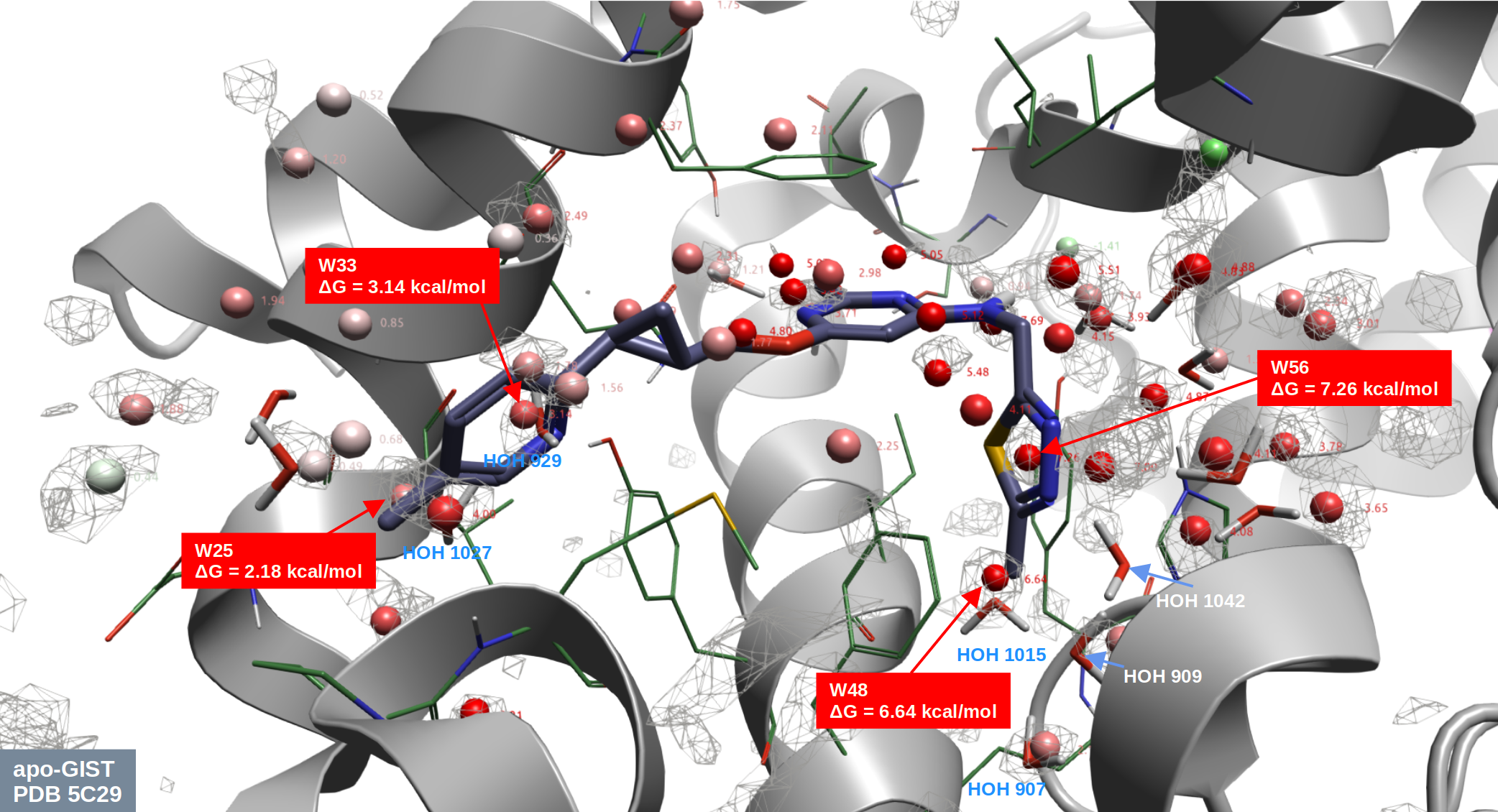
图6. 关键水合位点的热力学性质。棍状分子:化合物18;棍状水:实验解释的水;球状水:预测的水合位点;飘带:PDE10A(PDB 5C29);灰色网格:GIST Water Density \(\ge\)6.0。
HOH929、1027分别与显示水合位点W33、25重合,其水合自由能ΔG分别为3.14与2.18 kcal/mol,都是Unhappy水。化合物13-18系列的R1吡啶环置换了H929,这对结合亲合力是有利的;在R1吡啶-4位引入的甲基置换了高能的HOH1027,这可进一步提高了化合物的结合自由能,与化合物13-18系列的SAR一致(图2):引入一个甲基或甲氧基,Ki降低一个数量级(~10倍)。
9系列的SAR
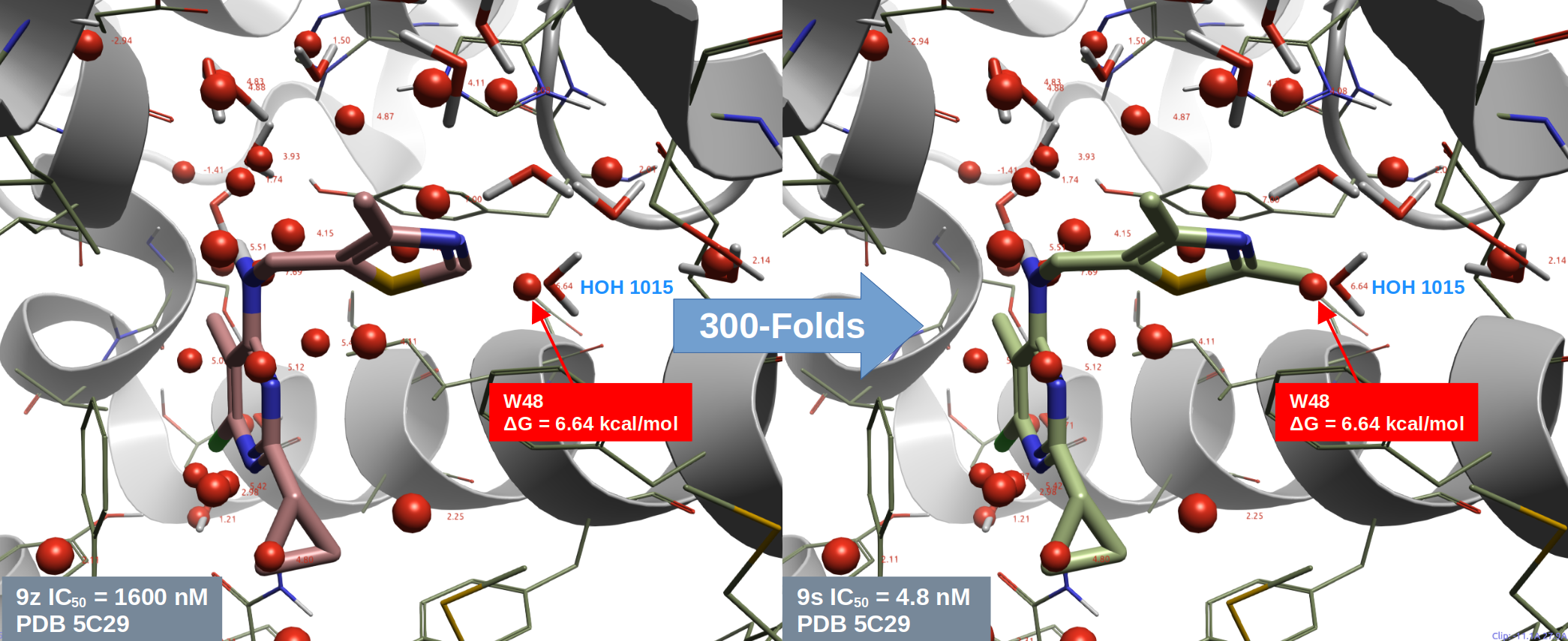
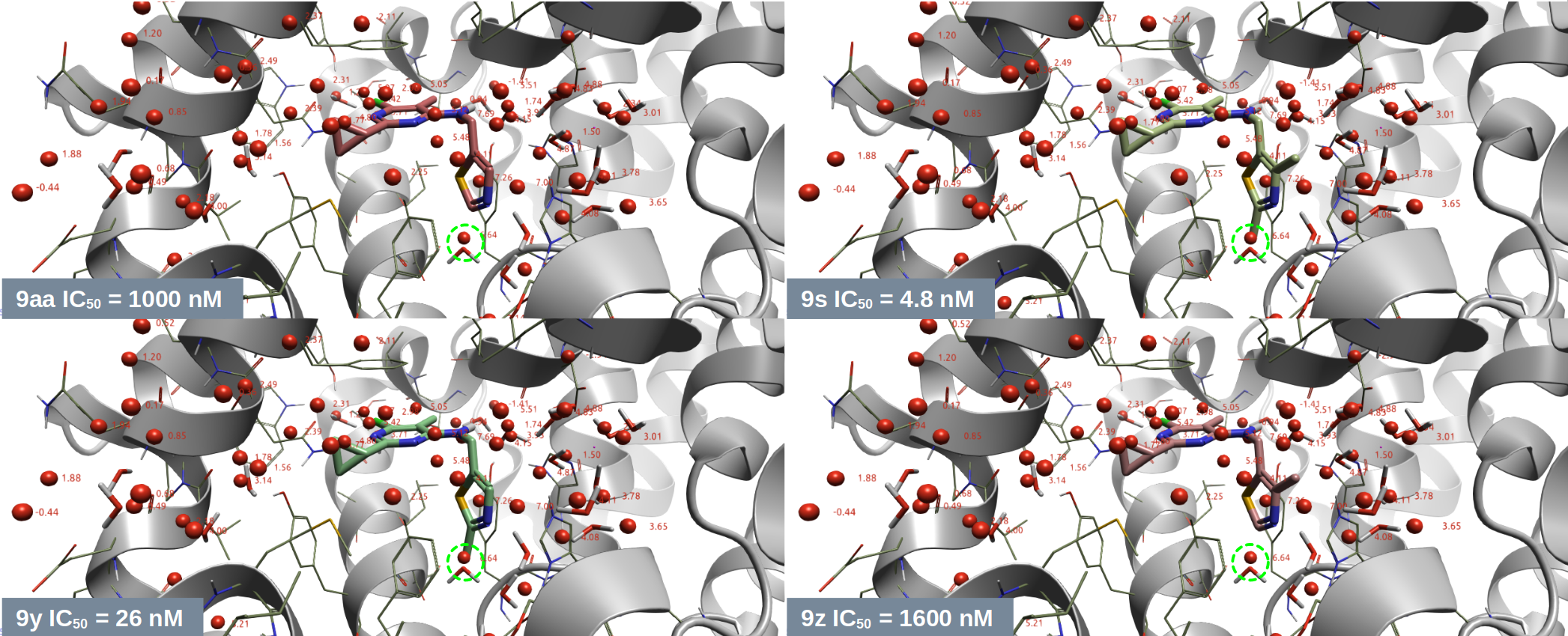
图7. 9系列化合物的结合模式及其水置换模式。飘带:PDE10A(PDB 5C29);棍状水:实验解释的水;球状水:GIST预测的水合位点;绿色高亮区球形:水合位点W48;绿色高亮区棍状:结晶水1015。
13-18系列的R2取代基末端甲基(见图2)置换HOH1015的SAR可以从9系列化合物6-位取代的SAR(图1)获得。将9s与PDE10A共晶结构PDB 5C2A按Cα原子叠合到9h与PDE10的共晶结构PDB 5C29,然后在结合位点编辑获得9z、9aa、9y的结合模式,结果如图7所示。可以发现,在9系列的噻唑环上引入置换高能、Unhappy的HOH1015(图7-绿色高亮区)之后活性提高了30-300多倍。比如,9aa引入甲基(9y)与甲氧基(9bb)后活性分别提高了38与100倍;9z在噻唑环上引入甲基(9s)后,活性提高了333倍。
综上所述,用GIST分析apo结合位点可以为基于结构的设计提供信息非常丰富、明确的水合位点及其热力学信息,可以定性地解释化合物18(MK-8189)的R1与R2的SAR,用来指导新化合物的设计。
定量计算高能水置换的结合自由能增益:ΔGwatdisp
在上一小节中,我们用GIST计算的水合位点上的ΔG来说明哪些是高能的“unhappy“水,哪些是低能的“happy”水,并据此来评估哪些水被配体置换后可以获得结合自由能的增加。当配体与蛋白结合时,配体的原子占据了结合位点里水分子的位置时,会发生对应的蛋白去溶剂化自由能变化,配体获得对应的结合自由能增益,可以定量表示为ΔGwatdisp 。9系列化合物9s与9z在结构上的差异仅在于9s在噻唑环上多了一个置换HOH1015的甲基,但这导致9s比9z的活性增强300多倍,这可用它们的ΔGwatdisp来加以解释。
分别计算HOH1015的水合自由能,以及9z与9s的ΔGwatdisp及其差值,结果如表2所示。
表2. 化合物9z与9s的ΔGwatdisp值及其差值(单位:kcal/mol)
| Items | ΔGwatdisp | ΔΔGwatdisp | Exp. ΔΔGbind |
|---|---|---|---|
| HOH1015 | 5.0* | – | – |
| 9s | -41.599 | 3.37 | 3.44 |
| 9z | -38.233 |
*:水合自由能,与置换自由能符号相反。
对HOH1015水合自由能计算的结果表明,在距离HOH1015水分子1.4 Å范围之内,包含了40个体素:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 | x y z distance dG dG_trunced -------- --------- -------- -------- --------- ---------- 10.19521 13.17006 41.7274 1.39791 5.19446 3.00000 10.19521 13.17006 42.2274 1.25321 2.33735 2.33735 10.19521 13.17006 42.7274 1.29882 0.03409 0.03409 10.19521 13.67006 41.7274 1.25108 3.21279 3.00000 10.19521 13.67006 42.2274 1.08702 0.83338 0.83338 10.19521 13.67006 42.7274 1.13930 0.01786 0.01786 10.19521 14.17006 41.7274 1.29471 0.10853 0.10853 10.19521 14.17006 42.2274 1.13695 0.01672 0.01672 10.69521 12.67006 42.2274 1.28051 0.79780 0.79780 10.69521 13.17006 41.7274 1.06506 20.36070 3.00000 10.69521 13.17006 42.2274 0.86646 7.79934 3.00000 10.69521 13.17006 42.7274 0.93121 0.15240 0.15240 10.69521 13.67006 41.2274 1.27633 2.22786 2.22786 10.69521 13.67006 41.7274 0.86337 17.14060 3.00000 10.69521 13.67006 42.2274 0.60151 5.86556 3.00000 10.69521 13.67006 42.7274 0.69153 0.16267 0.16267 10.69521 14.17006 41.2274 1.31912 0.19725 0.19725 10.69521 14.17006 41.7274 0.92546 1.19197 1.19197 10.69521 14.17006 42.2274 0.68766 0.22516 0.22516 11.19521 12.67006 41.7274 1.30518 1.86438 1.86438 11.19521 12.67006 42.2274 1.14887 0.28206 0.28206 11.19521 13.17006 41.2274 1.30314 1.33371 1.33371 11.19521 13.17006 41.7274 0.90253 6.82475 3.00000 11.19521 13.17006 42.2274 0.65648 2.68514 2.68514 11.19521 13.17006 42.7274 0.73984 0.07187 0.07187 11.19521 13.67006 41.2274 1.14421 1.21156 1.21156 11.19521 13.67006 41.7274 0.65240 4.87310 3.00000 11.19521 13.67006 42.2274 0.20500 1.47881 1.47881 11.19521 13.67006 42.7274 0.39803 0.03380 0.03380 11.19521 14.17006 41.2274 1.19176 0.13567 0.13567 11.19521 14.17006 41.7274 0.73259 0.24281 0.24281 11.19521 14.17006 42.2274 0.39126 0.08861 0.08861 11.19521 14.17006 42.7274 0.51912 0.01662 0.01662 11.69521 12.67006 41.7274 1.37248 0.06875 0.06875 11.69521 13.17006 41.2274 1.37054 0.10683 0.10683 11.69521 13.17006 41.7274 0.99738 0.17787 0.17787 11.69521 13.17006 42.2274 0.78178 0.04549 0.04549 11.69521 13.17006 42.7274 0.85298 0.02345 0.02345 11.69521 13.67006 41.2274 1.22042 0.09250 0.09250 11.69521 13.67006 41.7274 0.77835 0.06222 0.06222 Total unique voxels: 40 dG : 11.200 kcal/mol dG_trunced: 5.291 kcal/mol |
其中18个体素的下限截断值高于 0.5 kcal/mol,它们的GGIST范围在0-20 kcal/mol/Å3:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 | x y z distance dG dG_trunced -------- --------- -------- --------- ----------- ------------------- 10.19521 13.17006 41.7274 1.39791 5.19446 3.00000 10.19521 13.17006 42.2274 1.25321 2.33735 2.33735 10.19521 13.67006 41.7274 1.25108 3.21279 3.00000 10.19521 13.67006 42.2274 1.08702 0.83338 0.83338 10.69521 12.67006 42.2274 1.28051 0.79780 0.79780 10.69521 13.17006 41.7274 1.06506 20.36070 3.00000 10.69521 13.17006 42.2274 0.86646 7.79934 3.00000 10.69521 13.67006 41.2274 1.27633 2.22786 2.22786 10.69521 13.67006 41.7274 0.86337 17.14060 3.00000 10.69521 13.67006 42.2274 0.60151 5.86556 3.00000 10.69521 14.17006 41.7274 0.92546 1.19197 1.19197 11.19521 12.67006 41.7274 1.30518 1.86438 1.86438 11.19521 13.17006 41.2274 1.30314 1.33371 1.33371 11.19521 13.17006 41.7274 0.90253 6.82475 3.00000 11.19521 13.17006 42.2274 0.65648 2.68514 2.68514 11.19521 13.67006 41.2274 1.14421 1.21156 1.21156 11.19521 13.67006 41.7274 0.65240 4.87310 3.00000 11.19521 13.67006 42.2274 0.20500 1.47881 1.47881 Total unique voxels: 18 dG : 10.904 kcal/mol dG_trunced: 4.995 kcal/mol |
用方程2(见方法部分)计算这些体素的自由能贡献为10.9kcal/mol,按方程3取上限截断值后计算的水合自由能为5.00 kcal/mol,这可预期配体原子对HOH1015的置换可带来-5.0kcal/mol的结合自由能增加。注意到,9s因甲基带来的结合亲合力增加未达到预期对HOH1015置换带来的自由能增值(-5.0kcal/mol),这与此前的研究结论10-12一致,即水分子的水合自由能与置换该水分子的配体亲合力没有直接的相关性。
9s、9z的ΔGwatdisp分别为-41.599、-38.233 kcal/mol,它们的差值ΔΔGwatdisp = 3.37 kcal/mol,也就是9s比9z多获得的结合自由能,这与它们的实验结合自由能差值 3.44 kcal/mol几乎一致!
分子对接研究
用分子对接打分来预测化合物的结合亲和力是常用的方法之一。GNINA是流行的分子对接软件AutoDock Vina的深度学习打分版本7-9。用GNINA将化合物16、18在PDB 8DI4的结合口袋里进行score_only模式的对接计算,计算时将蛋白里面的水全部保留,结果如表3所示。
表3. 化合物16、18的分子对接GNINA打分结果
| Items | 16 | 18 |
|---|---|---|
| gauss 1 | 127.01881 | 135.60562 |
| gauss 2 | 1568.71167 | 1632.83203 |
| repulsion | 4.74825 | 4.80877 |
| hydrophobic | 40.51922 | 44.30338 |
| H-Bond | 5.04394 | 5.04394 |
| Vina score(kcal/mol) | -9.22 | -9.74 |
| CNNaffinity(pKd) | 8.31 | 8.42 |
| CNNscore | 0.99 | 1.0 |
| PDE10A Exp. Ki (nM/pKd) | 0.33/9.48 | 0.029/10.54 |
| PDE10A Exp. ΔG(kcal/mol) | -12.92 | -14.36 |
注:AutoDock Vina的打分项为加权之前的值;实验值pKd与ΔGbinding是根据Ki计算转化而来。
16比18少了一个置换HOH1027的甲基,16与18的主要差异体现在与蛋白的形状互补与疏水相互作用上:16的gauss项与疏水相互作用项比18的略小,因此16比18预测的结合自由能(Vina score)略高,分别为-9.22、-9.74 kcal/mol,但并没有显著的差异。相似的,GNINA深度学习打分CNNaffinity预测16、18的结合亲和力分别为8.31与8.42(pKd),也没有显著差异。这说明,无论是经典的Vina打分、还是深度学习亲和力打分都不能解释实验观察到的活性差异。
用GNINA将化合物9z、9s在PDB 5C2A的结合口袋里进行score_only模式的对接计算,计算时将蛋白里面的水全部保留,结果如表4所示。
表4. 化合物9z、9s的分子对接GNINA打分结果
| Items | 9z | 9s |
|---|---|---|
| gauss 1 | 90.46870 | 102.15282 |
| gauss 2 | 1146.61963 | 1210.75623 |
| repulsion | 3.08232 | 3.13979 |
| hydrophobic | 39.80846 | 53.22118 |
| H-Bond | 2.60499 | 2.60512 |
| Vina score(kcal/mol) | -7.87 | -8.62 |
| CNNaffinity(pKd) | 7.79 | 8.28 |
| CNNscore | 0.98 | 0.99 |
| PDE10A Exp. Ki (nM/pKd) | 1600/5.80 | 4.8/8.31 |
| PDE10A Exp. ΔG(kcal/mol) | -7.90 | -11.34 |
注:AutoDock Vina的打分项为加权之前的值;实验值pKd与ΔGbinding是根据Ki计算转化而来。
9z比9s少了一个置换HOH1015的甲基,在Vina Score里9z与9s的差异也仅在与蛋白的形状互补与疏水上:9z的gauss项与疏水相互作用项比9s的略小,因此9z比9s预测的结合自由能(Vina score)略高,分别为-7.87、-8.62 kcal/mol,预测的相对活性0.84kcal/mol,远远低于实验的相对值3.44kcal/mol。GNINA深度学习打分CNNaffinity预测9z、9s的结合亲和力分别为7.79、8.28(pKd),相对值为0.49pKd,远低于实验相对值2.51(pKd)。虽然GNINA的CNNaffinity准确地预测了化合物9s的结合亲和力,但是不能明确地、显著地与化合物9z区分开。无论是经典的Vina打分、还是深度学习亲和力打分都不能确切、有信心地解释实验观察到R1与R2置换水HOH1027、HOH1015带来的活性差异。
两种基于结构方法的比较:分子对接打分与水合位点信息对基于结构设计指导作用的比较
首先,在业务流程上两者有着显著的区别。分子对接需要同时利用蛋白与配体的信息,先要有设计出来的化合物,然后进行分子对接打分,最后比较打分值、进行优先级排序。而水合位点信息仅需蛋白结构信息,计算水合位点及其热力学性质,然后以水为中心,产生分子设计的想法与建议,需要构思、设计新化合物该是什么样子。从这一点上看,我更倾向于用虚拟筛选与基于结构设计来区分两者:虚拟筛选方法需要有现成的化合物、然后打分、进行优先级排序;而以水为中心的设计,不直接得到化合物,而是根据水合位点及其热力学的信息去构思、设计新化合物。
在这个例子中,显然分子对接打分对9z/9s,16/18这两个分子对没有足够的区分度,不能信心充足地排序、进行优先级排序;而根据GIST给出的水合位点与自由能,利用在R1、R2末端两处足够大且显著的水合位点自由能,可以充满信心地用来指导新化合物的优化设计,并与实验结果匹配良好。
用Hit Expander进行自动配体生成
从上述GIST分析结果可知,在R1与R2取代基末端的芳香环附近有高能的水可供置换,仅需引入一个单原子或双原子小基团可以实现水分子替换。Hit Expander5是Flare™一个组件,它通过通过添加小官能团或进行芳香单原子替换(例如,芳香碳原子转化为芳香氮原子)快速地生成衍生物库,随后可以使用Flare的其他组件进行分析。接下来我们开始用Hit Expander5自动生成潜在的水分子置换设计。
如图8所示,我们选中化物9aa 6位取代基末端噻唑环上靠近HOH1015的碳原子(图8-高亮原子),然后点击Hit Expander,选择添加甲基、甲氧基、氯、腈基以及三氟甲基。
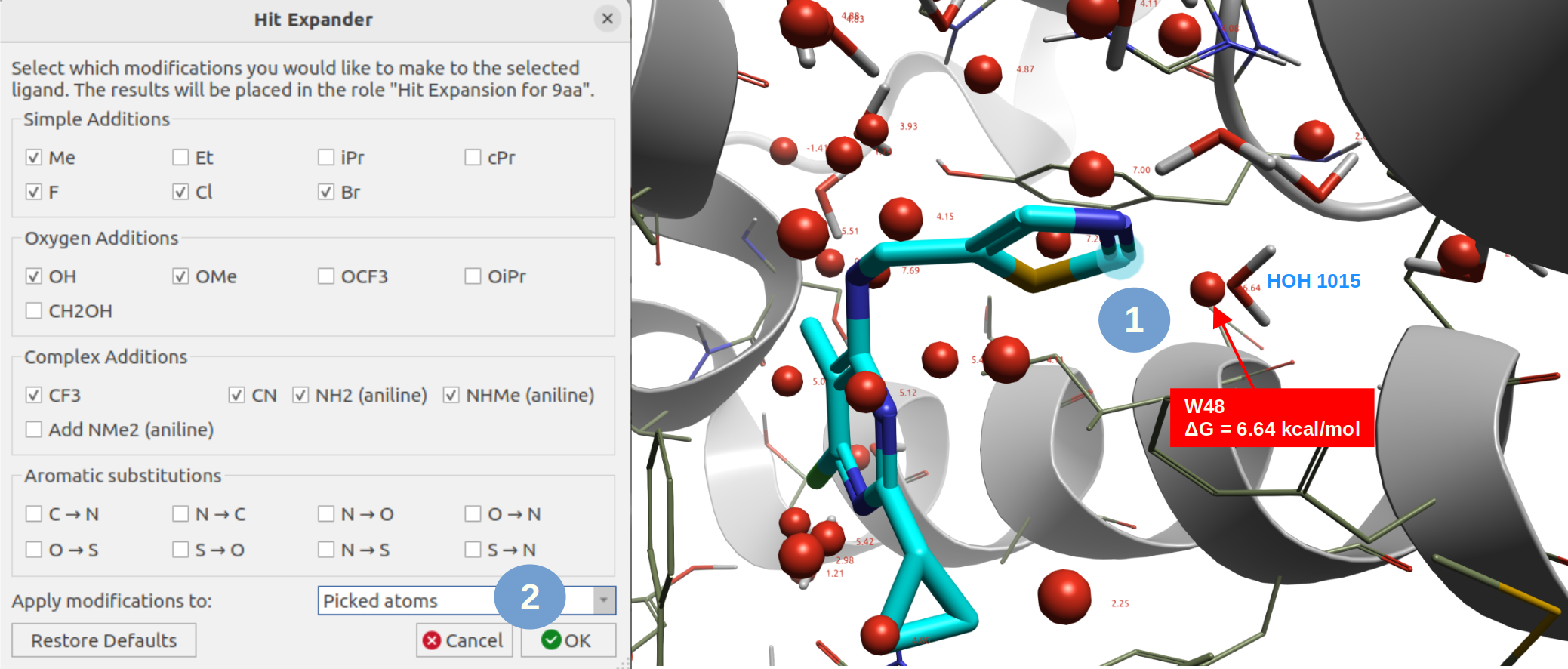
图8. 用Hit Expander对9aa末端噻唑选中的原子进行自动修饰。飘带:PDE10A(PDB 5C29);棍状水:实验解释的水;球状水:GIST预测的水合位点。
Hit Expander5生成了5个新化合物,结果如图9所示,它自动进行了几何优化、叠合到起始化合物9aa上,我们仅需要考察结果即可。我们可以看到,新生成的5个化合物都很好地实现了对HOH1015的置换。不仅包含了Shipe等人1报道的甲基与甲氧基取代设计,还包含了令人感兴趣但未见文献报道的设计。
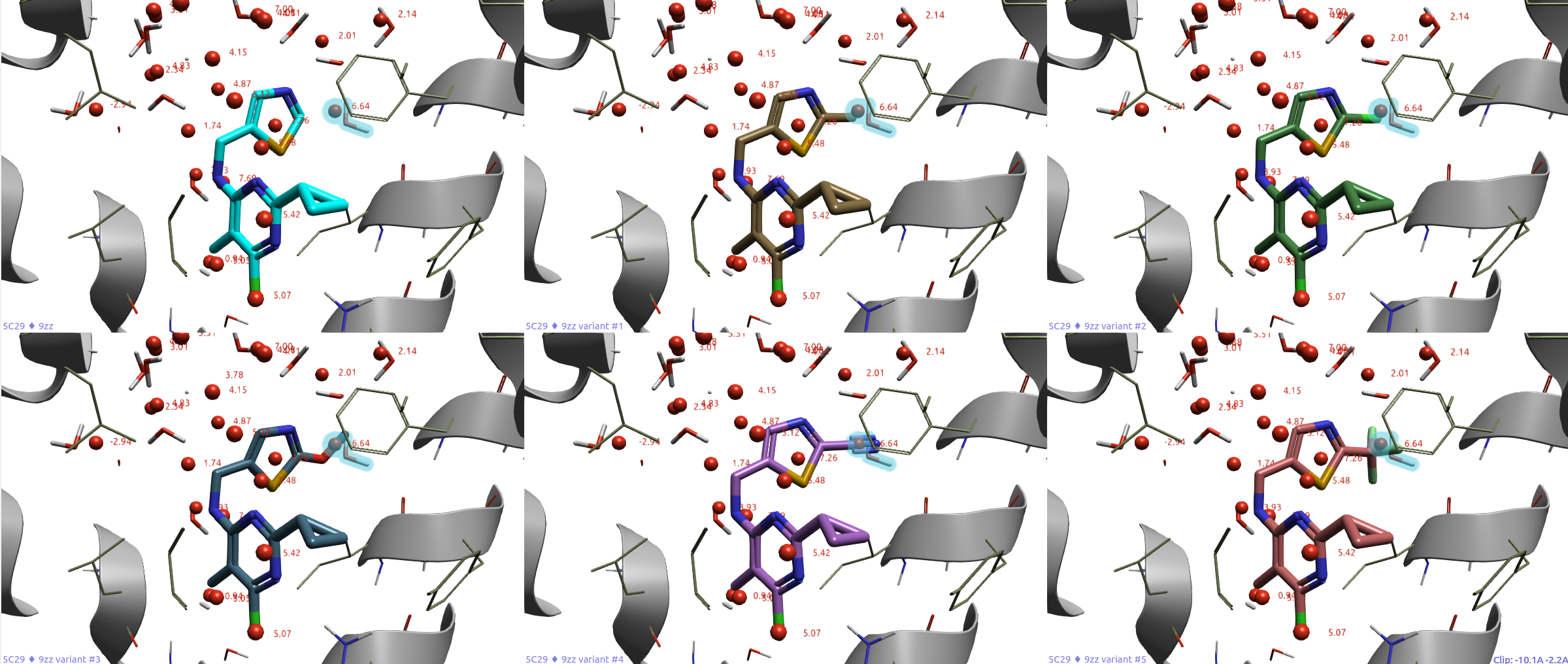
图9. 起始化合物9aa(青色)以及Hit Expander自动修饰的5个结果。飘带:PDE10A(PDB 5C29);棍状水:实验解释的水;球状水:GIST预测的水合位点;高亮球形:水合位点W48;高亮区棍状:结晶水1015。
计算9aa以及5个Hit Expander衍生物ΔGwatdisp,结果如表5所示。与起始化合物9aa相比,5个衍生物的ΔGwatdisp都获得了-3kcal/mol以上的增加(ΔΔGwatdisp),这表明这些衍生有望具有更好的结合亲合力。
表5. 化合物9aa以及5个衍生物的ΔGwatdisp
| Items | ΔGwatdisp | ΔΔGwatdisp |
|---|---|---|
| 9aa(starter) | -36.83 | – |
| 9aa-CH3 (#1) | -40.08 | -3.25 |
| 9aa-Cl (#2) | -40.37 | -3.54 |
| 9aa-OCH3 (#3) | -39.78 | -2.95 |
| 9aa-CN (#4) | -41.57 | -4.74 |
| 9aa-CF3 (#5) | -39.96 | -3.13 |
Unit:kcal/mol
至此,Hit Expander显示出与GIST水分析绝佳搭配的潜力,与GIST水分析结果组合可提供快速、自动地分子设计。新设计的分子可以进一步用进行互补性打分、FEP等进行进一步的分析、过滤。
方法
蛋白与配体结构准备
将共晶结构下载到Flare V83中,并用Protein Prep工具进行结构准备以添加氢原子、优化氢键、消除原子冲突并给蛋白结构与配体分配最佳质子化状态,任何截短的蛋白质链被封端作为蛋白质准备的一部分。
GIST水分析
用Flare V83对“湿”结合位点Apo结构(不包含配体与金属离子)进行GIST分析,共晶结构的A链水包含在GIST分析里面,具体的GIST分析条件如下:
- Calculation method: Normal
- Ligand: None
- Grid spacing: 0.5 Å
- Grid Definition:Ligand
- Chains: A Chain, A Water
- Simulation length: 20ns
- Solvent Model: explicit TIP4Pew Water
在这个计算中,在分子动力学模拟之前将共晶结构的水链包含在GIST分析里。
水合位点预测与水合自由能计算
对水密度进行聚类分析得到显式的水合位点。首先,选择具有最高水氧密度的体素来确定第一个水合位点的位置。然后排除所有在第一个水合位点2.5Å范围内的体素,不再考虑。然后,对下一个最高密度的体素重复此过程,直到没有剩下密度高于2倍本体水氧密度的体素为止。计算采用基于pyflare的脚本来实现。
然后识别每个水合位点周围1.4Å半径内的体素(Voxel):
$$
V = \left\{v_{i} | v_{i} \in hydration\ site\right\} \cdots(1)
$$
如方程4所示,如果一个体素包含在水合位点的范德华半径内,则累加这些体素的能量,并将总和乘以体素的体积(vol = 0.125 Å3),得到以kcal/mol为单位的值,如方程5所示:
$$
ΔG = vol \times \sum_{v_{i} \in V}G_{GIST}(v_{i})\cdots(2)
$$
方程2的ΔG即是水合位点的水合自由能。在计算的时候,对体素vi上的GGIST值设置了下限截断值±0.5kcal・mol-1・Å-3来去掉贡献不显著的体素,使用上限截断值+3kcal・mol-1・Å-3去掉极端高值的体素,如方程6所示:
$$
G_{GIST}(v_{i}) = \left \{
\begin{aligned}
& 0\ &if\ |G_{GIST}(v_{i})| \lt 0.5 \\
& +3\ &if\ G_{GIST}(v_{i}) \ge +3 \\
& G_{GIST}(v_{i}) &\ otherwise
\end{aligned}
\right.\cdots(3)
$$
计算采用基于pyflare的脚本来实现。
配体结合的溶剂置换自由能:ΔGwatdisp
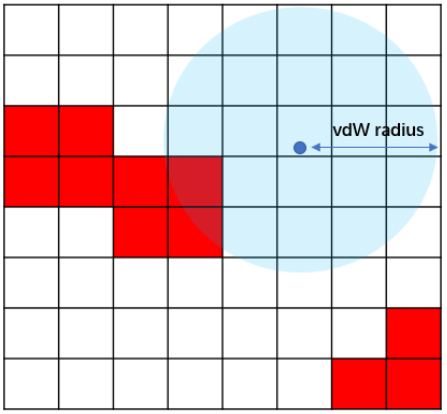
图10. 用GIST来计算当配体原子占据结合水分子的位置时发生的去溶剂化自由能。蓝色球:配体的原子𝑖;红色网格:“unhappy“水的网格k。
当配体原子占据了结合位点里水分子的位置时,会发生对应的蛋白去溶剂化自由能变化,配体获得对应的结合自由能增益,表示为ΔGwatdisp。为了评估受体在结合过程中的去溶剂化自由能,首先识别被配体原子置换的体素(Voxel),如方程4所示:
$$
V = \left\{v_{i} | v_{i} \in ligand\right\} \cdots(4)
$$
图10解释了计算方法,在配体结合到蛋白口袋的过程中,如果一个体素包含在一个配体原子的范德华半径内,则认为该体素被配体置换。累加这些体素的能量值,并将总和乘以体素的体积,得到以kcal/mol为单位的值,如方程5所示:
$$
ΔG_{watdisp} = \alpha \times vol \times \sum_{v_{i} \in V}G_{GIST}(v_{i})\cdots(5)
$$
其中α是缩放因子,在这里α=-1。而在Balius等人13虚拟筛选目的的研究中,探索了多种α值,当α=-0.5时,比起没有使用基于GIST的蛋白去溶剂化的打分方法,大部分情况下虚拟筛选的性能得到显著地提高。在GIST计算时格点为0.5 Å(space=0.5 Å),因此这里的vol = 0.125 Å3。而且,在计算的时候,对体素vi上的GGIST值设置了方程3同样的截断值处理。
计算采用基于pyflare的脚本来实现。
结论
在本文中,用Flare GIST分析了PDE10A的apo结合位点,用水合位点与水合自由能解释了苗头化物与先导化合物优化过程中的SAR,回溯性地证明了Flare GIST在基于结构中指导分子设计的作用。还根据GIST分析结果,选择感兴趣的修饰位置,用Hit Expander自动、快速地生成衍生物,实现对高能水进行置换的设计,其中包括文献报道活性提高30-300倍的设计,还包含尚未见文献报道的、另人感兴趣的设计。Flare GIST与Hit Expander联合使用是强大且可靠的基于结构的药物设计工具。
文献
- Shipe, W.D. et al. (2015) “Discovery and Optimization of a Series of Pyrimidine-Based Phosphodiesterase 10A (PDE10A) Inhibitors through Fragment Screening, Structure-Based Design, and Parallel Synthesis,” Journal of Medicinal Chemistry, 58(19). Available at: https://doi.org/10.1021/acs.jmedchem.5b00983.
- Layton, M.E. et al. (2023) “Discovery of MK-8189, a Highly Potent and Selective PDE10A Inhibitor for the Treatment of Schizophrenia,” Journal of Medicinal Chemistry, 66(2). Available at: https://doi.org/10.1021/acs.jmedchem.2c01521.
- Flare V8. http://www.cresset-group.com/software/flare
- FlareTM GIST. https://www.cresset-group.com/software/flare-3drism/#GIST
- FlareTM Hit Expander. https://www.cresset-group.com/software/flare-hitexpander
- FlareTM Pocket. https://cresset-group.com/software/flare-pocket-detection
- Trott, O. and Olson, A.J. (2009) “AutoDock Vina: Improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading,” Journal of Computational Chemistry, 31(2), pp. 455–461. Available at: https://doi.org/10.1002/jcc.21334.
- Sunseri, J. and Koes, D.R. (2021) “Virtual Screening with Gnina 1.0,” Molecules, 26(23), p. 7369. Available at: https://doi.org/10.3390/molecules26237369.
- GNINA 1.0, https://github.com/gnina
- Haider, K. and Huggins, D.J. (2013) “Combining solvent thermodynamic profiles with functionality maps of the Hsp90 binding site to predict the displacement of water molecules,” Journal of Chemical Information and Modeling, 53(10). Available at: https://doi.org/10.1021/ci4003409.
- Abel, R. et al. (2008) “Role of the active-site solvent in the thermodynamics of factor Xa ligand binding,” Journal of the American Chemical Society, 130(9), pp. 2817–2831. Available at: https://doi.org/10.1021/ja0771033.
- Nguyen, C.N. et al. (2014) “Thermodynamics of Water in an Enzyme Active Site: Grid-Based Hydration Analysis of Coagulation Factor Xa,” Journal of Chemical Theory and Computation, 10(7), pp. 2769–2780. Available at: https://doi.org/10.1021/ct401110x.
- Balius, T.E. et al. (2017) “Testing inhomogeneous solvation theory in structure-based ligand discovery,” Proceedings of the National Academy of Sciences, 114(33), pp. E6839–E6846. Available at: https://doi.org/10.1073/pnas.1703287114.
获取试用,请联系我们
想要在您自己的项目上尝试Flare GIST与Hit Expander,您可申请Flare试用版并了解如何将GIST与Hit Expander方法组合应用于其他基于结构设计的项目。