摘要:本文分享了Mpakali等人用Blaze虚拟筛选发现靶向IRAP酶活性开放构象抑制剂的研究案例。通过在3D场描述符空间中进行虚拟筛选,使得作者能够用肽类苗头化合物作为查询化合物来搜索小分子化学空间,以识别能够针对靶标的小分子药物化学项目的苗头化合物。从苗头到先导化合物的衍生物实现了更高的活性和所需的物理化学性质。这体现了Blaze识别新颖而相关化合物的能力,这些化合物对于推动分子设计项目向着高效且有前景的化合物发展至关重要。
作者:Oliver Hills/2024-10-28
编译:肖高铿
前言
胰岛素调节氨基肽酶(Insulin-regulated aminopeptidase,IRAP)是一种跨膜蛋白,对多种独特且独立的生物过程至关重要,包括肽调控、胰岛素应答性转运、心血管调控和认知功能等。事实上,与认知功能相关的作用使得IRAP成为阿尔茨海默症研究的核心,因为小分子IRAP抑制剂有可能通过稳定神经肽和改善突触可塑性来提高阿尔茨海默病患者的认知功能。最近,Mpakali等人1发表了一项研究,该研究靶向IRAP的酶活性开放构象,并专注于使用Cresset的3D虚拟筛选解决方案Blaze™发现小分子IRAP抑制剂。
在本文中,我们强调了Cresset虚拟筛选技术在真实世界小分子IRAP抑制剂识别的成功应用。讨论了如何使用Cresset基于配体的3D虚拟筛选技术筛选数百万种化合物,获得了良好的命中率。
用Blaze在3D场描述符空间进行虚拟筛选
在筛选活动中,通常会对包含数百万化合物预先枚举的化学库进行虚拟筛选。一般而言,用计算成本较低的一维或二维指纹描述符来评估化合物的相似性是常见的做法。然而,由于忽略了查询分子的三维静电场和形状场信息,因此此类方法在提供结构新颖的结果方面存在局限性。具体来说,仅基于连接性和子结构描述分子是过于简化的,常常无法避免化学类型偏差。
Blaze通过在三D静电场和形状场空间中进行筛选克服了这个问题。通过利用XED计算的场点模式3,该模式本质上包含了配体与蛋白质发生相互作用所需的所有相关信息。这类描述符具有“化学无关性”,即基于蛋白质如何“感知”候选化合物来进行评估。
与查询分子在静电和形状场点模式上具有高相似性的化合物预计也会与查询分子具有相似的生物活性。为此,Blaze的工作流程如下:
- 提供查询分子的3D生物活性构象,计算其场点模式。
- 基于此,Blaze对多构象“现货供应的”商业化合物数据库(3000多万化合物)执行多级搜索(图1)。
- 查询分子与数据库中化合物之间的场点模式相似性是基于50%的静电势3贡献和50%的形状4贡献来计算总体相似性打分值。尽管在实际操作中,这些参数可以根据需要进行调整以增加多样性。
- 然后,与查询分子具有高度静电和形状场点模式相似性的化合物被确定为苗头化合物。
- Blaze返回一个按与查询分子的三维静电和形状相似性排序的“苗头化合物列表”,这种方法区别于基于药效团的虚拟筛选中通常观察到的二元“通过/不通过”输出。
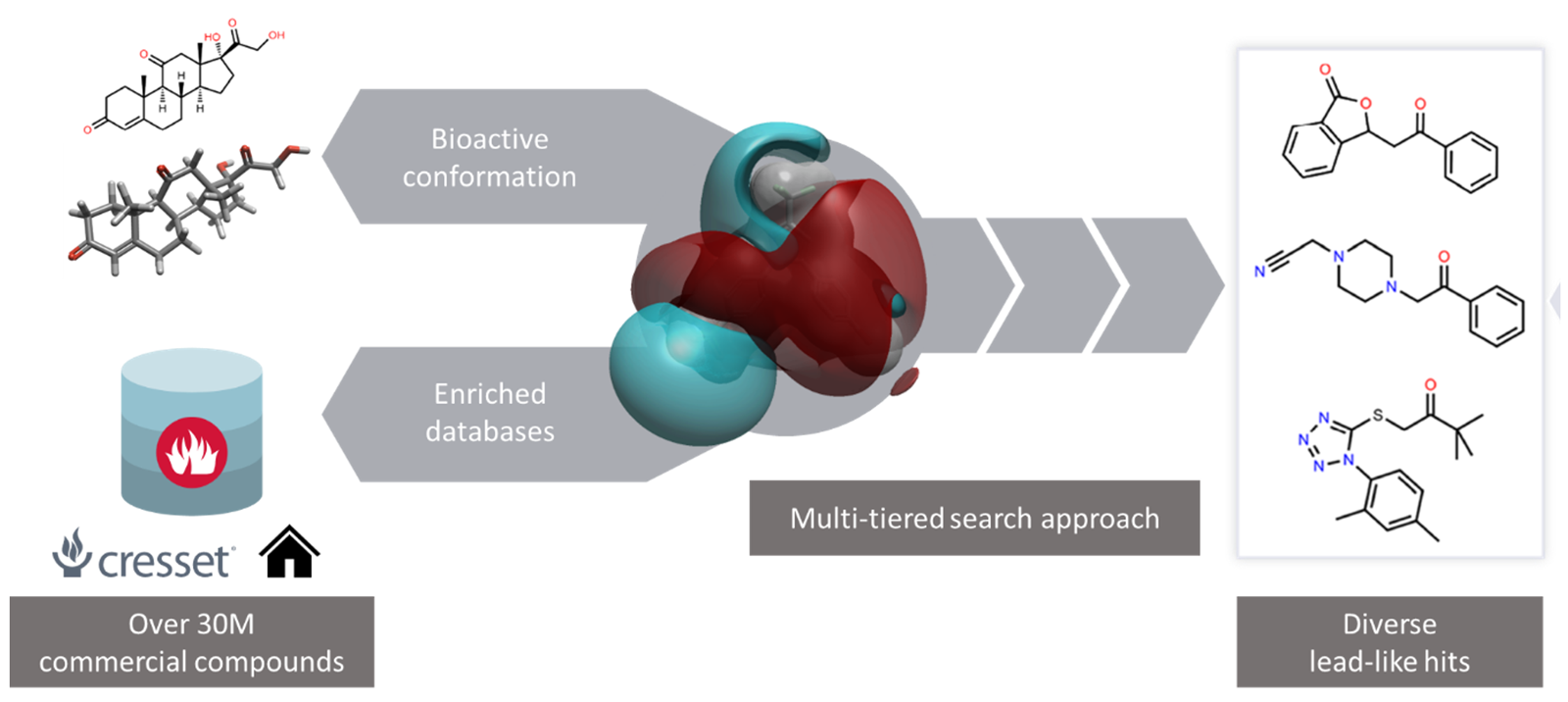
图1. Blaze 3D虚拟筛选工作流
Blaze发现针对IRAP的苗头化合物并启动药物化学优化项目
在Mpakali等人的研究中,Blaze搜索的查询分子是与IRAP共晶结构(PDB: 4Z7I,图2)的过渡态抑制剂DG025(序列2X0-4L8-KHHAFSFK)5。
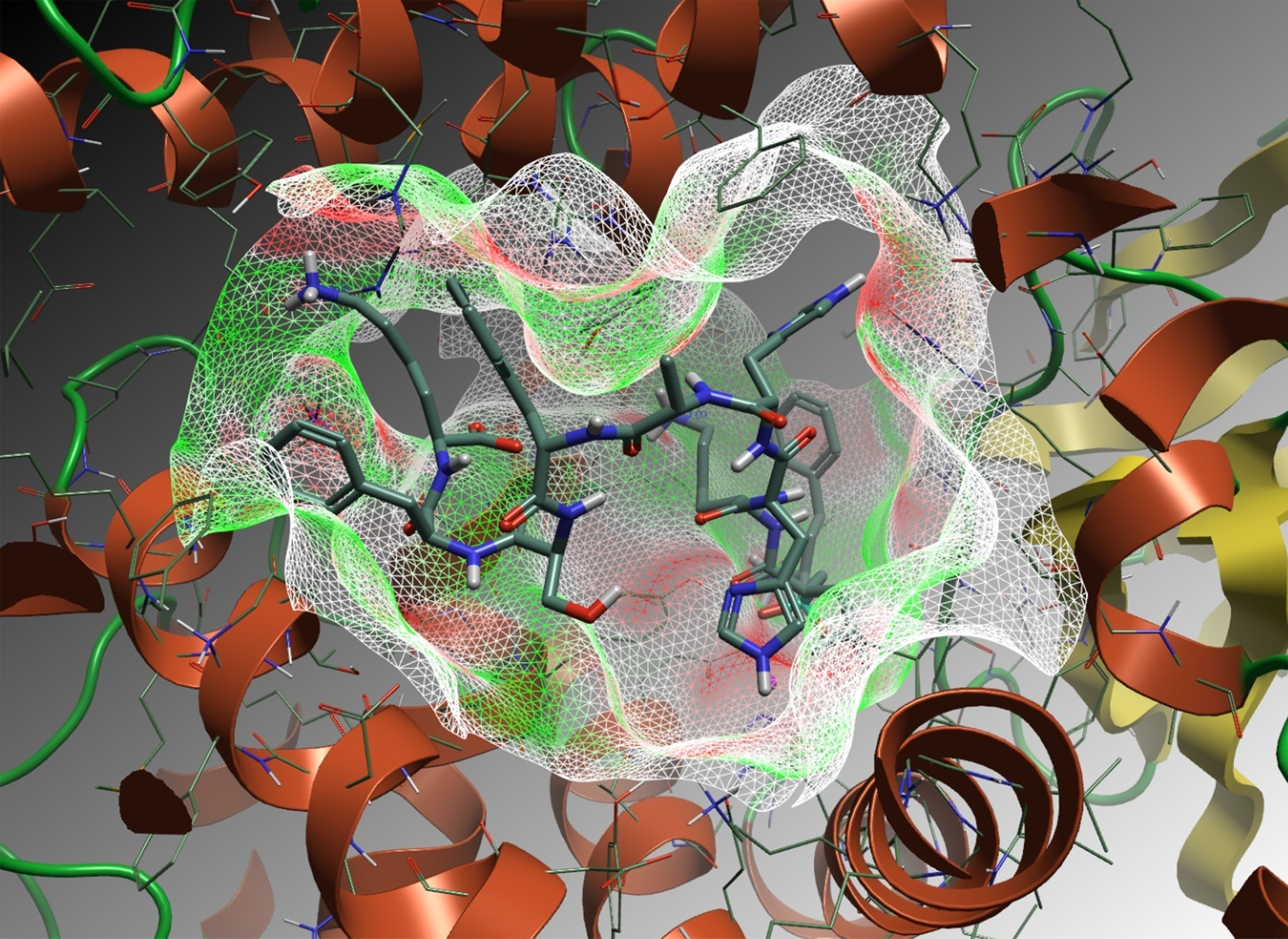
图2. DG025过渡态类似物与IRAP的共晶结构(PDB: 4Z7I)。电荷互补性™ (EC) 等值面已渲染在蛋白质的活性位点上。所有图像均使用Flare™生成3,6。
DG025本身是一个伪十肽,为了此次研究目的,将其截短至前四个残基。将这个查询分子提交给Blaze,对其中商业数据库和ChEMBL数据库进行了虚拟筛选实验。所有的苗头化合物都用PAINS过滤器等筛选以去除不希望的化合物,并最终按照化学相似性进行聚类。从每个聚类中,购买打分最高的苗头化合物并进行了体外测试。
Blaze识别到的苗头化合物1(图3)表现出适度的活性(IC50 = 55 μM)。首先,也是最重要的一点是,尽管从一个伪肽查询分子开始,Blaze却检索到了一个小分子。这是用3D场描述符进行虚拟筛选优势的一个例证,因为场点是“化学无关性”的,这开启了搜索到2D相似性低但3D场相似性高的化合物的潜力。图3显示了化合物1与四肽伪肽查询分子的场叠合,在骨架酰胺、吡唑和氮杂环庚烷区域分别聚集了静电场点和疏水场点。可以理解的是,鉴于这些保守的场特征,Blaze搜索到了一个苗头化合物,具有与四肽伪肽查询分子相同的结合构象、并具备结合到IRAP结合位点相同区域所需的3D静电和形状特征。
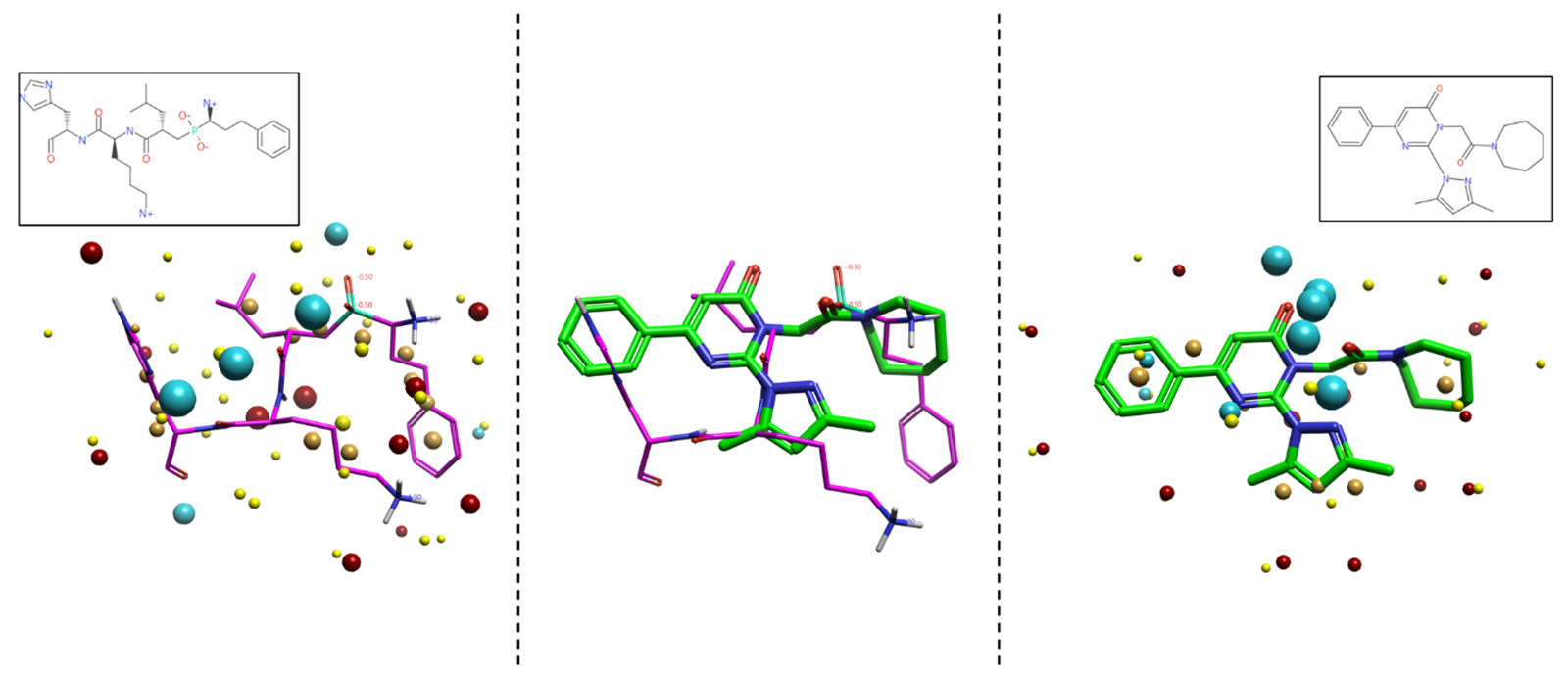
图3. Blaze查询化合物(左),Blaze苗头化合物 1(右)以及苗头化合物1与查询化合物的3D叠合(中间)。红色场点为静电正值,蓝色场点为静电负值,黄色表示形状,桔黄色表示疏水。
尽管如此,重要的是要注意,这项研究并不仅仅停留在从虚拟筛选中识别出化合物1。相反,作者将化合物1作为药物化学优化的起点,不仅提高了其活性,还改善了化合物1的物理化学性质。具体来说,末端苯基和氮杂环庚烷取代基通过中间化合物2进行了迭代转化,从而得到了化合物3(图4)。进一步的体外测试表明,化合物3是一种高选择性的IRAP抑制剂,能够以IC50 = 157 nM的浓度与催化活性开放构象的IRAP结合。
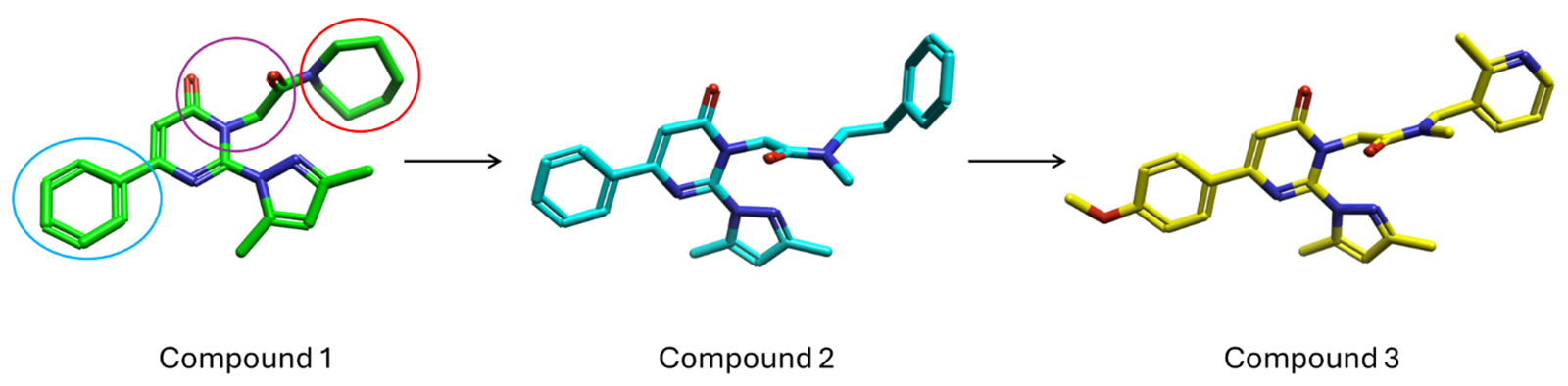
图4. 初始的苗头化合物1、中间优化的合物2和进一步优化后的化合物3的分子结构。化合物1上的圆圈标出了药物化学项目的重点。
使用Blaze快速推进分子发现项目
通过在3D场描述符空间中进行虚拟筛选,使得作者能够用肽类苗头化合物作为查询化合物来搜索小分子化学空间,以识别能够针对靶标的小分子药物化学项目的苗头化合物。从苗头到先导化合物的衍生物实现了更高的活性和所需的物理化学性质。这体现了Blaze识别新颖而相关化合物的能力,这些化合物对于推动分子设计项目向着高效且有前景的化合物发展至关重要。
文献
- Mpakali, A.; Georgaki, G.; Buson, A.; Findlay, A. D.; Foot, J. S.; Mauvais, F.; van Endert, P.; Giastas, P.; Hamprecht, D. W.; Stratikos, E. Stabilization of the Open Conformation Οf Insulin‐regulated Aminopeptidase by a Novel Substrate‐selective Small‐molecule Inhibitor. Protein Sci 2024, 33 (9), e5151. https://doi.org/10.1002/pro.5151.
- Cheeseright, T. J.; Mackey, M. D.; Melville, J. L.; Vinter, J. G. FieldScreen: Virtual Screening Using Molecular Fields. Application to the DUD Data Set. J. Chem. Inf. Model. 2008, 48 (11), 2108–2117. https://doi.org/10.1021/ci800110p.
- Cheeseright, T.; Mackey, M.; Rose, S.; Vinter, A. Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665–676. https://doi.org/10.1021/ci050357s.
- Grant, J. A.; Gallardo, M. A.; Pickup, B. T. A Fast Method of Molecular Shape Comparison: A Simple Application of a Gaussian Description of Molecular Shape. J. Comput. Chem. 1996, 17 (14), 1653–1666.
- Mpakali, A.; Saridakis, E.; Harlos, K.; Zhao, Y.; Papakyriakou, A.; Kokkala, P.; Georgiadis, D.; Stratikos, E. Crystal Structure of Insulin-Regulated Aminopeptidase with Bound Substrate Analogue Provides Insight on Antigenic Epitope Precursor Recognition and Processing. J. Immunol. 2015, 195 (6), 2842–2851. https://doi.org/10.4049/jimmunol.1501103.
- Bauer, M. R.; Mackey, M. D. Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein-Ligand Complexes. J. Med. Chem. 2019, 62 (6), 3036–3050. https://doi.org/10.1021/acs.jmedchem.8b01925.
联系我们
Blaze可以通过软件方式、SaaS云计算方式或FET服务方式来使用,商务合作请联系我们:info@molcalx.com



