摘要:HPK1作为一种靶标,其特征在于结合口袋具有较高的构象柔性,并涉及到溶剂化作用的影响,这为使用自由能微扰(FEP)方法预测其抑制剂的结合自由能带来了挑战。本文阐述了如何运用CADD平台Flare中的一系列工具来执行蛋白质结构准备、水分析、分子动力学模拟以及配体叠合等步骤,从而为体系进行充分的前期准备。通过这些准备工作,我们能够在FEP基准测试计算中取得令人满意的结果。
作者:Federico Issoglio & Stuart Firth-Clark. Cresset, Cambridgeshire, UK.
编译:肖高铿/2025-01-09
前言
本研究展示了一次相对结合自由能(RBFE)计算,包括一组28个与HPK1结合的化合物1。HPK1是一种丝氨酸/苏氨酸激酶,属于STE20家族的一员。由于它负向调控T细胞抗原受体的激活信号,并且其激酶活性在这个过程中起着关键作用,因此HPK1是癌症和传染病治疗中的一个令人感兴趣的靶标。
人们已经确认,在溶液中,HPK1激酶域(HPK1-KD)的激活段涉及单体和二聚体之间的平衡,而二聚体通过结构域交换构象在活性/非活性状态之间交替2。
对于这种结构柔性的蛋白靶标,为了从自由能微扰(FEP)计算中获得准确的预测,对受体和配体进行仔细准备是必不可少的。利用Flare中的各种工具,设计了一个全面的工作流来选择和准备受体,生成配体同系物系列的紧密叠合,并创建用于后续FEP计算的连接图。
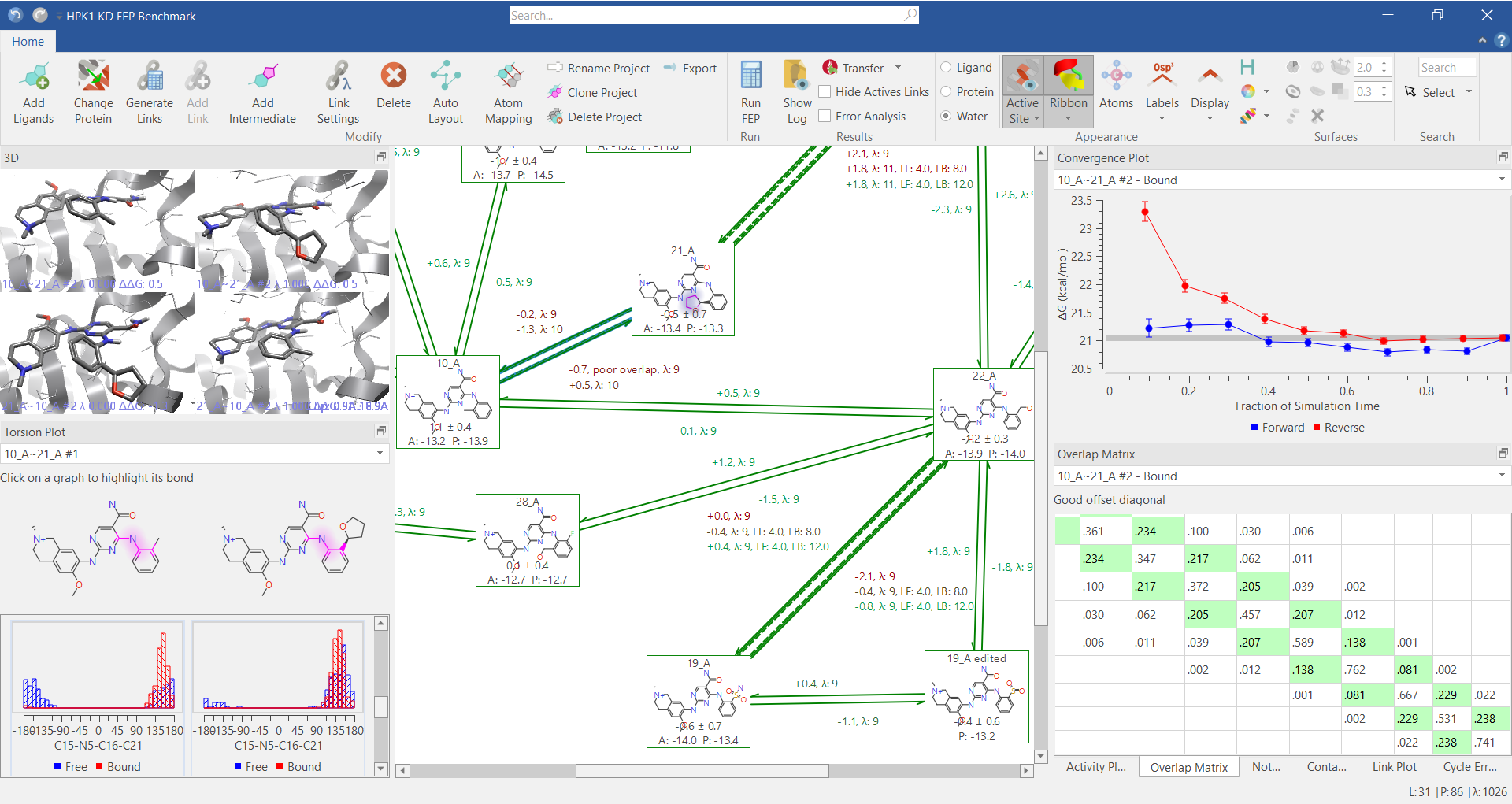
图1. Flare FEP图形界面,包括FEP结果分析工具与故障排除工具(比如扭转角图,收敛图与叠合矩阵等)。
方法
每一个步骤都使用了计算机辅助药物设计(CADD)平台Flare,包括蛋白质和配体的准备、水分析、分子动力学模拟、配体叠合和自由能微扰(FEP)计算等。
受体准备
口袋分析和3D-RISM被用于评估溶剂可及性,并生成结合位点溶剂化缺失的信息,利用现有的实验数据来证实和验证这些预测。
分子动力学模拟
- 200 ns production runs: 7M0K, 7M0L, 7M0M.
- Protein forcefield: Amber ff14SB
- Small molecule forcefield: OpenFF 2.2.0
- Charge method: AM1-BCC
- Water model: TIP3P, 在平衡阶段使用GCNCMC协议
配体叠合
- Method: 用最大公共子结构(MCS)方法进行同系物的叠合
- Reference:在第1轮中用共晶配体,在第2轮中用打分最高的结果
- Refinement: 利用静电互补性(EC)和场点信息来指导叠合地优化
Flare FEP
- 参数:同分子动力学模拟准备阶段
- 每个λ窗口模拟时长:初始设为4ns,需要更长时间采样的链路可增加至最高15ns。
- 相空间重叠不足的情况下,会额外计算超出自适应λ调度所定义的λ窗口数量(最小值=9)。
结果
受体准备
迄今为止报道的27个蛋白结构揭示了HPK1-KD(HPK1激酶域)具有高度的构象可塑性,这涉及到包含催化结构域与DFG所在的激活环(A-loop)。这种异质性反映在含有共结晶参比分子的三个PDB结构中。通过口袋分析和使用3D-RISM进行的水分析得到的结果表明,对活性位点处水网络的适当描述是至关重要的。
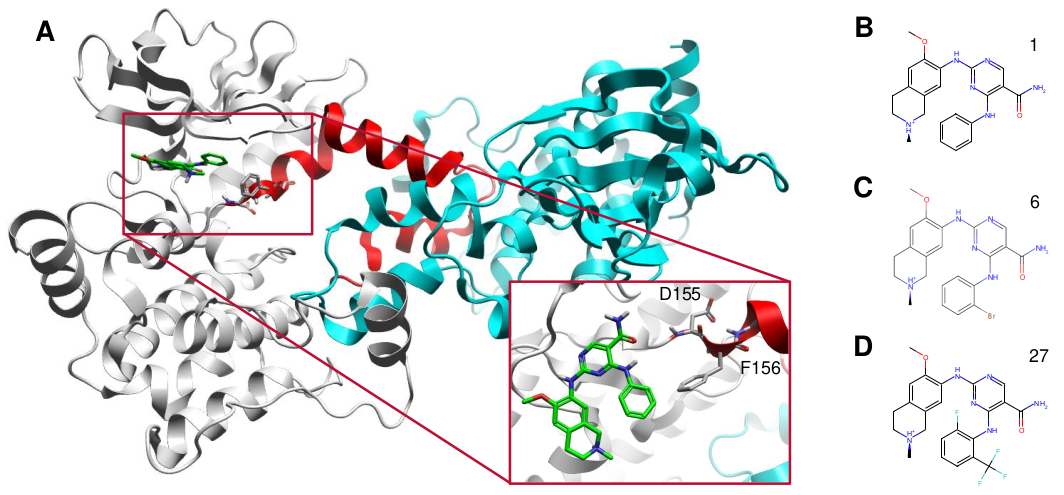
图2. A) HPK1 (PDB 7M0K) 的飘带图表示,包括一个内嵌的放大图,显示了链A的活性位点(链B以青色显示,来自链A的A-loop以红色显示),并展示了DFG中的D155和F156的相对位置(化合物#1以绿色棍状模型显示)。B-D) 分别为PDB 7M0K、7M0M和7M0M中共晶化合物的二维结构表示,分别为化合物#1、#6和#27。
分子动力学模拟(Molecular Dynamics Simulations)
使用Flare的Dynamics模块评估了复合物的构象稳定性。在蛋白质准备过程中可知,结合位点的溶剂化对于获得良好的预测可能至关重要。因此,在分子动力学模拟(MDs)的平衡步骤期间采用了GCNCMC协议,以改善活性位点处水分子的采样。结合使用Flare图形用户界面上的MD分析工具和内置的Python环境pyflare进行了详尽的检查。在仔细检查了RMSD(均方根偏差)和主成分分析(PCA)的结果后,选择了对应于PDB 7M0K的结构用于FEP模拟。对图3-A到C的PCA图分析显示,7M0K的分子动力学模拟呈现出更窄的构象采样,并且更接近初始构象。
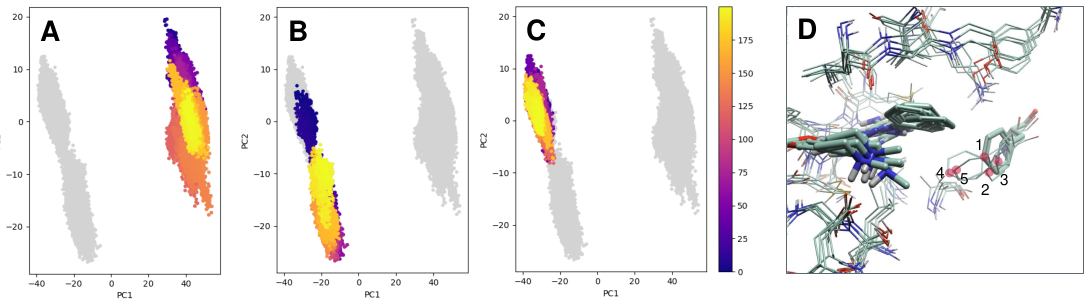
图3. A-C) 包括活性位点侧链所有重原子获得的主成分分析(PCA)图。可以使用颜色编码条追踪模拟的时间长度(纳秒,ns)。D) 在7M0K轨迹中对活性位点残基应用聚类分析得到的5个质心的叠加。
配体叠合
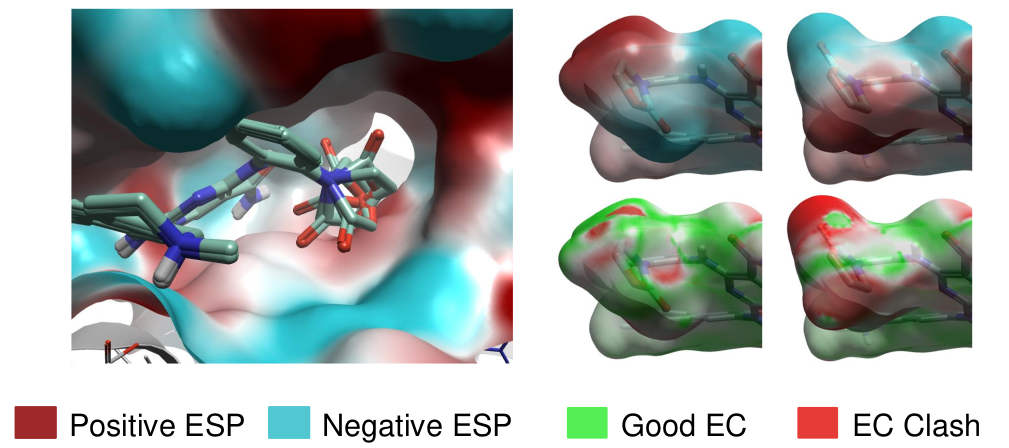
图4. 胺甲酰基的不同取向对静电互补性(EC)有较大影响。
从7M0K轨迹中选择了一个参考构象,对结合位点的蛋白质残基进行聚类分析,如图3D所示。在将同系物配体与参比配体(化合物#1)进行最大公共子结构(MCS)叠合后,使用静电互补性(EC)为部分配体选择优选的R基团的取向(图4),这有助于决定后续FEP运行的最佳起点。对于其他配体,则利用场点(Field Points)提供的信息,在最终优化过程中检查配体的结合模式(图5)。
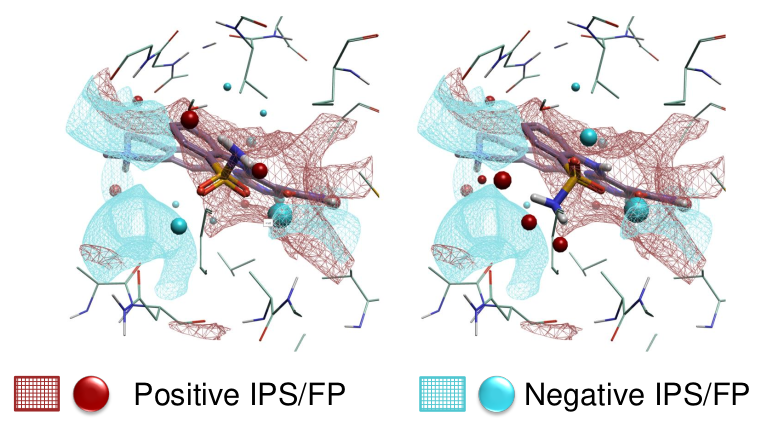
图5. 在磺酰胺基团不同取向的叠合时,蛋白质的静电分子相互作用势表面(IPS)与小分子场点(FP)。
Flare FEP基准测试
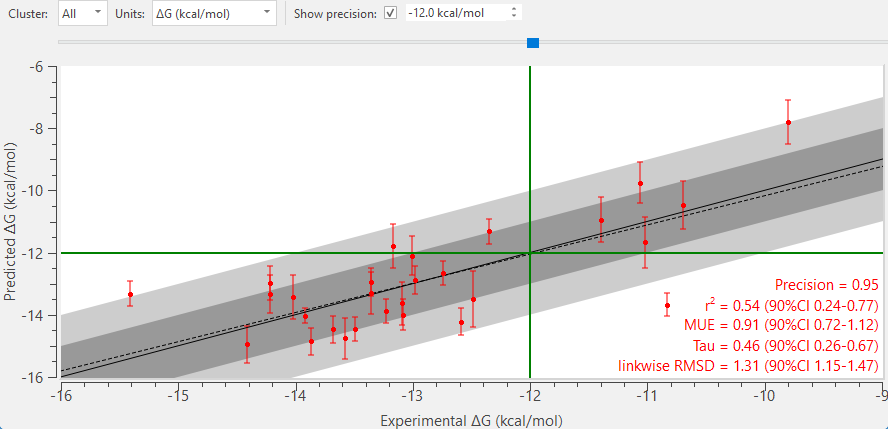
图6. FEP计算数据集中28个配体的活性图
- 与先前的分子动力学(MD)使用相同的设置(包括GCNCMC)
- 平均无符号误差(MUE)小于1 kcal/mol
- 需要使用3个中间体
- 总体较低的链接迟滞(link hysteresis)
- 高精度的预测结果
- 22个配体的预测误差在约1 kcal/mol以内
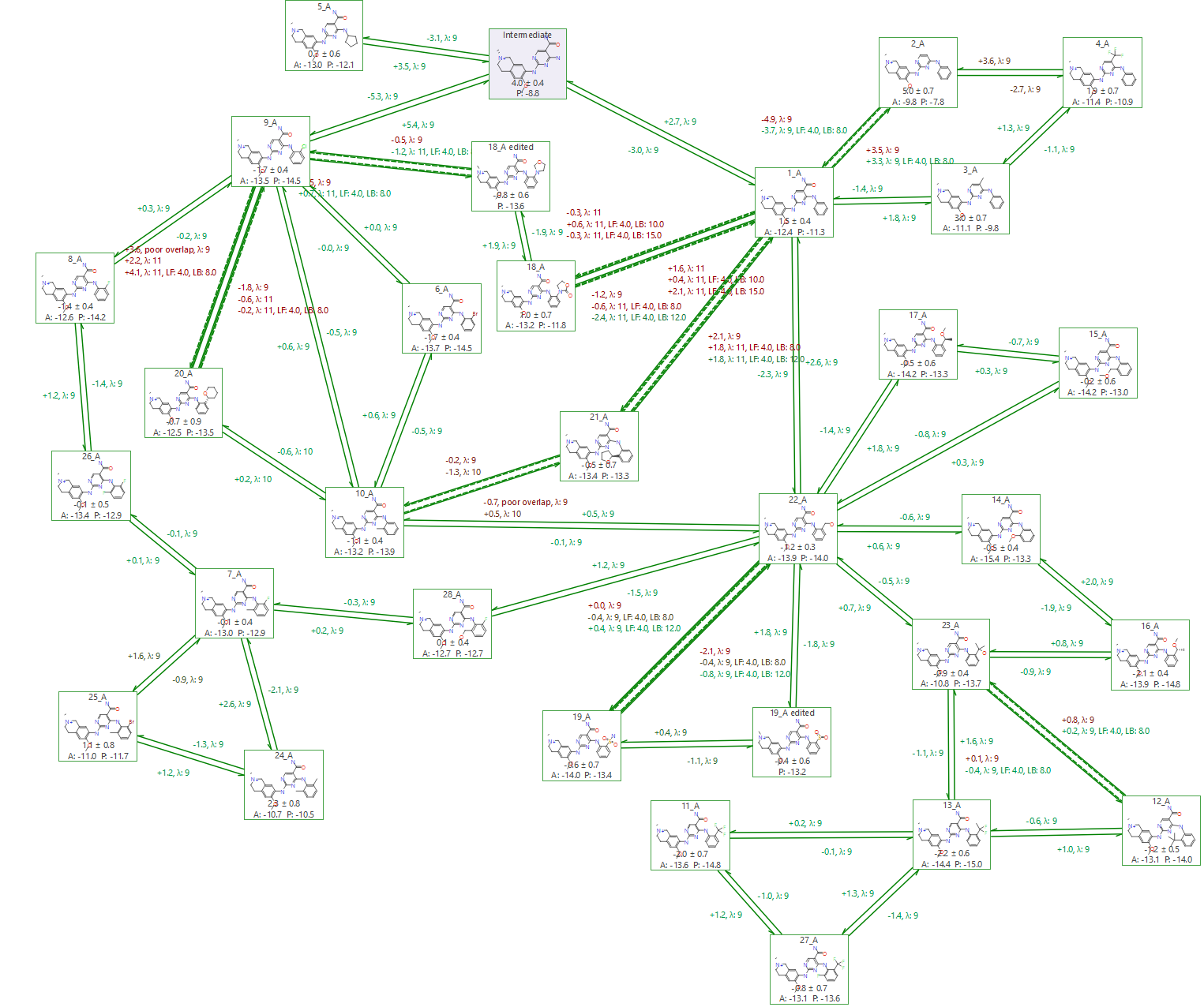
图7. 包含所有转化的连接图
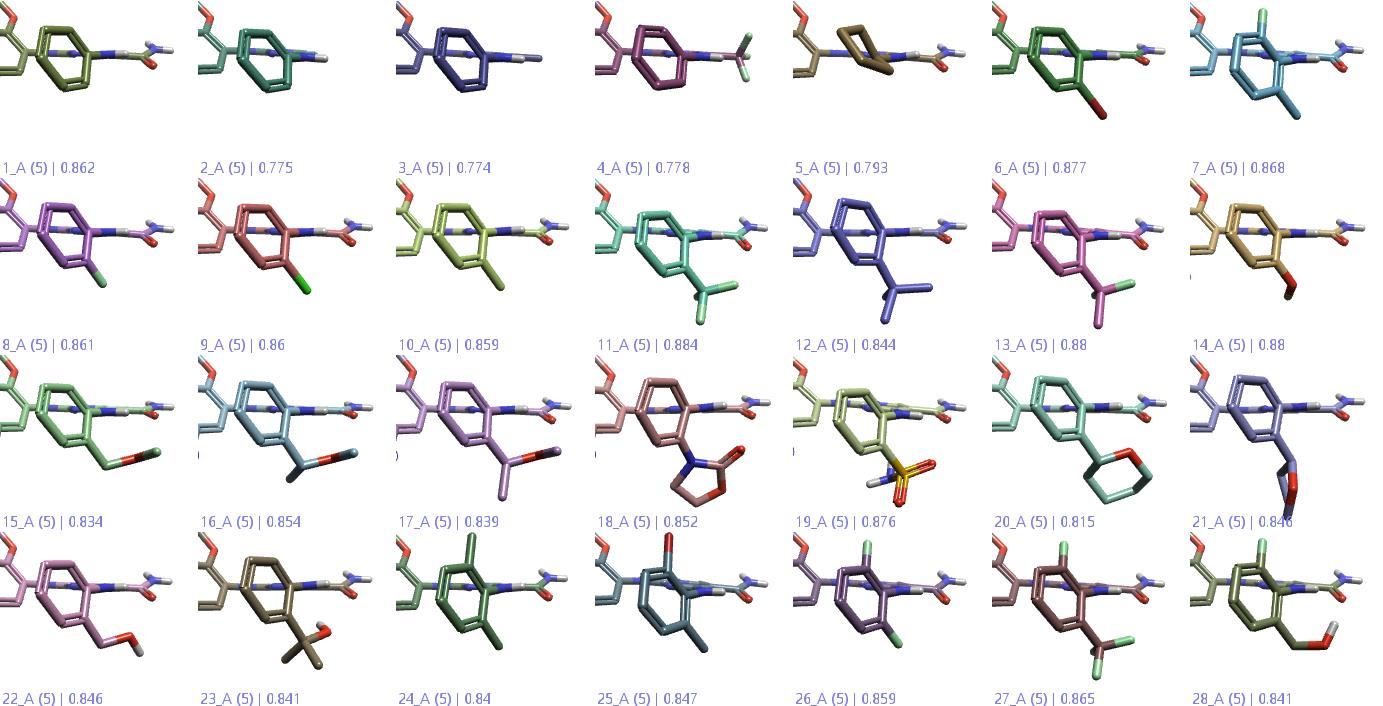
图8. FEP计算数据集中28个配体的3D视图,聚焦于发生取代的部分。
结论
仔细的受体准备对项目的成功至关重要,它提供了宝贵的信息,在定义工作流中的后续步骤时起到重要作用。
良好的叠合是实现准确自由能预测的基础,Flare分析工具在对这个流程的优化中是强大的助力。
HPK1的构象可塑性构成了重大挑战,但Flare FEP能够产生可靠的基准运行,为新设计的化合物进一步预测提供了坚实的基础。
文献
- Vara, Brandon A., et al. Discovery of diaminopyrimidine carboxamide HPK1 inhibitors as preclinical immunotherapy tool compounds. ACS Medicinal Chemistry Letters. 2021, 12 (4), 653-661.
- Wu, Ping, et al. Hematopoietic progenitor kinase-1 structure in a domain-swapped dimer. Structure. 2019, 27 (1) 125-133
原创文章,作者:小墨,如若转载,请注明出处:《用Flare FEP计算预测HPK1抑制剂的结合自由能》http://blog.molcalx.com.cn/2025/01/09/predicting-hpk1-inhibitor-binding-free-energy-using-flare-fep.html



