SPARK应用案例——RSV聚合酶抑制剂从DEL苗头到类药先导化合物的优化
摘要:在本研究中,来自强生的研究人员利用已知的封帽结构域结合物JNJ-8003进行了一次有针对性的DNA编码库(DEL)筛选,发现了一种结构新颖的三肽母核RSV聚合酶抑制剂,即苗头化合物16。为了克服16潜在的代谢不稳定问题,通过平行药物化学策略和SPARK骨架跃迁方法,成功将三肽母核替换为新型骨架,显著减少了酰胺键和手性中心数量。这些新骨架化合物在生化和/或细胞实验中仍保持活性,并表现出改善的人肝微粒体体外清除率。总的来说,初步的药物化学研究工作证实,这些新的化学类型具有开发前景,可以作为未来研发呼吸道合胞病毒(RSV)聚合酶抑制剂有价值的起点。证明了Spark在DEL Hit-to-Lead过程中丰富骨架化学多样性的强大能力。
原文:Carney, S.M. et al. (2025) “DNA-Encoded Library Screen Identifies Novel Series of Respiratory Syncytial Virus Polymerase Inhibitors,” Journal of Medicinal Chemistry, 68(6), pp. 6407–6430. Available at: https://doi.org/10.1021/acs.jmedchem.4c02906.
编译:肖高铿
前言
呼吸道合胞病毒(RSV)是导致婴儿、老年人、免疫功能低下者以及患有基础疾病的患者发生严重下呼吸道感染的主要病因之一1-5。然而,截至目前,尚未有任何针对呼吸道合胞病毒(RSV)的特异性抗病毒小分子药物获得重要的批准。
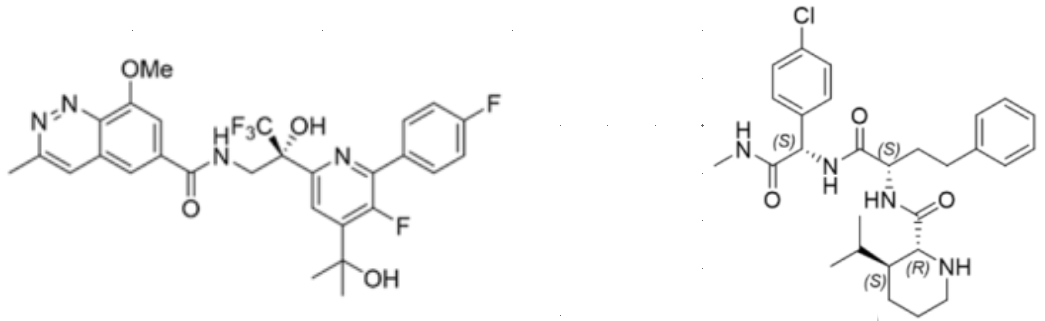
图1. JNJ-8003(左)与DEL苗头化合物16(右)
之前,强生的团队6报道了RSV聚合酶与非核苷类小分子JNJ-8003(图1)结合的cryo-EM结构,揭示了在RSV聚合酶封帽结构域上存在一个诱导契合的结合口袋。小分子JNJ-8003在封帽结构域上的结合能够抑制RdRp在从头合成(de novo)和早期延伸阶段的核苷酸添加活性,表明封帽结构域与RdRp结构域之间存在功能上的相互作用。JNJ-8003不仅在体外实验中表现出亚纳摩尔级(sub-nM)的结合亲和力、有效的聚合酶抑制活性以及细胞水平的抗病毒活性,并且在小鼠和羔羊模型中展现出明显的体内疗效,显示出针对这一结合口袋开发高效抑制剂的巨大潜力6,7。为了寻找新型的RSV聚合酶抑制剂骨架,强生团队的Carney等人8最近开展了一项DNA编码化合物库(DEL)筛选研究,重点聚焦于封帽结构域的结合口袋。
DNA编码化学库(DELs)相比传统的高通量筛选可以探索更大的化学空间9。利用DEL技术的发现策略已经为多种治疗靶点生成了强效且可进一步开发的抑制剂10,11。DEL分子的亲和选择旨在识别能够与感兴趣靶标直接结合的小分子。亲和选择过程可以利用已知的结合物进行偏向性筛选,从而从DEL中发现与已知结合物竞争结合同一靶点的分子,这有助于在对特定结合口袋或界面有重大研究兴趣时,聚焦再合成(resynthesis)的努力方向。根据团队建立的DEL选择策略,并由此发现了新型RSV聚合酶抑制剂(化合物16,图1),它与JNJ-8003一样,都结合到了相同的封帽结构域位点。化合物16的活性和结合模式得到了表征,并通过初步的结构-活性关系(SAR)验证了该化学类型的有效性。
然而,根据文献报道12,预计DEL苗头化合物16的三肽类母核存在代谢不稳定等问题与缺陷。肽类化合物通常会在血液、肝脏和肾脏中被内源性蛋白酶广泛水解,导致其发生显著的蛋白酶降解13。这种不稳定性可能导致在体内作用时间短、生物利用度差,从而限制其作为药物的应用潜力12。
为了克服苗头化合物16潜在的问题,研究团队开展了结构-活性关系(SAR)研究,旨在寻找替代三肽类母核的结构以提升其代谢稳定性。为此,采用了两种互补的方法来降低酰胺键和手性中心的数量:
- 以库模式进行平行合成
- 骨架跃迁策略
其中骨架跃迁策略是通过Cresset的Spark软件实现,本文的主要目的是分享Spark在DEL Hit-to-Lead骨架跃迁中的应用,并展示其强大的性能。
SPARK骨架跃迁(scaffold hopping)实验方法
在本研究中,Cresset的SPARK软件用于骨架跃迁搜索。使用了强生公司的化合物数据库和ChEMBL数据库 作为片段来源。
SPARK软件通过将符合查询分子角度和距离要求的匹配片段进行合并,生成可能的产物结构。然后根据这些产物结构与查询分子在静电场和分子形状上的相似性对其进行打分。为了便于计算,软件使用“场点”(field points)来构建一个可用于评估静电和形状相似性的计算框架,提供了两个指标对结果进行排序:
- BIF%(BioIsostere Factor,生物等排体因子):用于衡量分子片段之间在生物活性方面的可替换性
- Score(综合打分):结合了静电场相似性和形状相似性的总分
最终保留打分最高的前500个化合物用于进一步分析。
Spark骨架跃迁结果
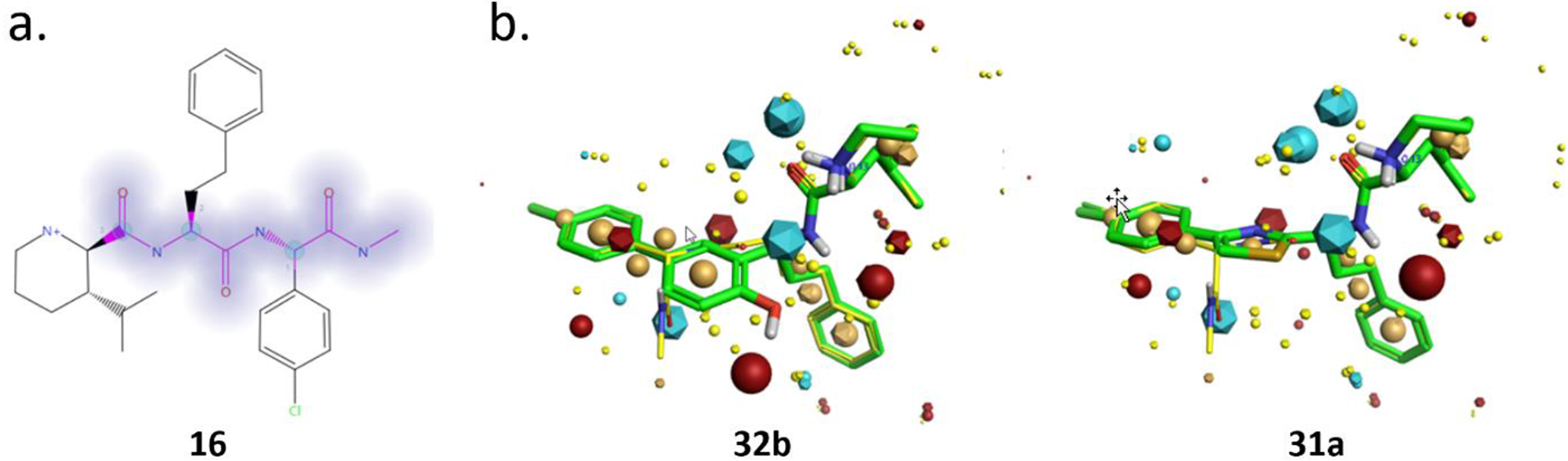
图2. SPARK骨架跃迁搜索。(a)与RSV聚合酶结合的化合物16的冷冻电镜结构(PDB 9ECV)作为计算的起点。(a)紫色高亮部分为化合物16的肽类骨架片段。(b)展示了化合物16(黄色)与苗头化合物32b和31a(绿色)的叠合图。同时还显示了查询化合物16(多面体)和苗头化合物(球体)的场点叠合结果:正静电场点,红色;负静电场点,蓝色;疏水场点,橙色;立体场点,黄色。苯酚和噻唑化合物32b(BIF=48%,Score=0.880)和31a(BIF=45%,Score = 0.873)在排名最高的500化合物中分别为排名第14和23,并使用基于多参数优化(MPO)的打分方法进行进行合成的优先级别排序。
SPARK骨架跃迁14以DEL苗头化合物16结合到RSV聚合脢的冷冻电镜结构(PDB 89CV)的生物活性构象为起点;通过搜索片段库来替换图2a中以蓝色/紫色突出显示的肽类骨架片段。然后根据BIF%(生物等排体因子)对打分排名前500的化合物进行初步筛选,并去除含有反应性官能团的化合物。随后,根据MPO(Multi-Parameter Optimization,多参数优化)打分选择了一部分化合物。该MPO打分综合了BIF%、合成可行性打分以及预测的人肝微粒体代谢稳定性15,16,各项的权重比为10:3:3。对MPO打分大于0.4的化合物进行人工结构分析,选择了双环系的31和32(图6b)并进行了合成,并进一步推动其近似类似物33–35的合成(表3)。
表3. Spark的三肽类骨架跃迁结果
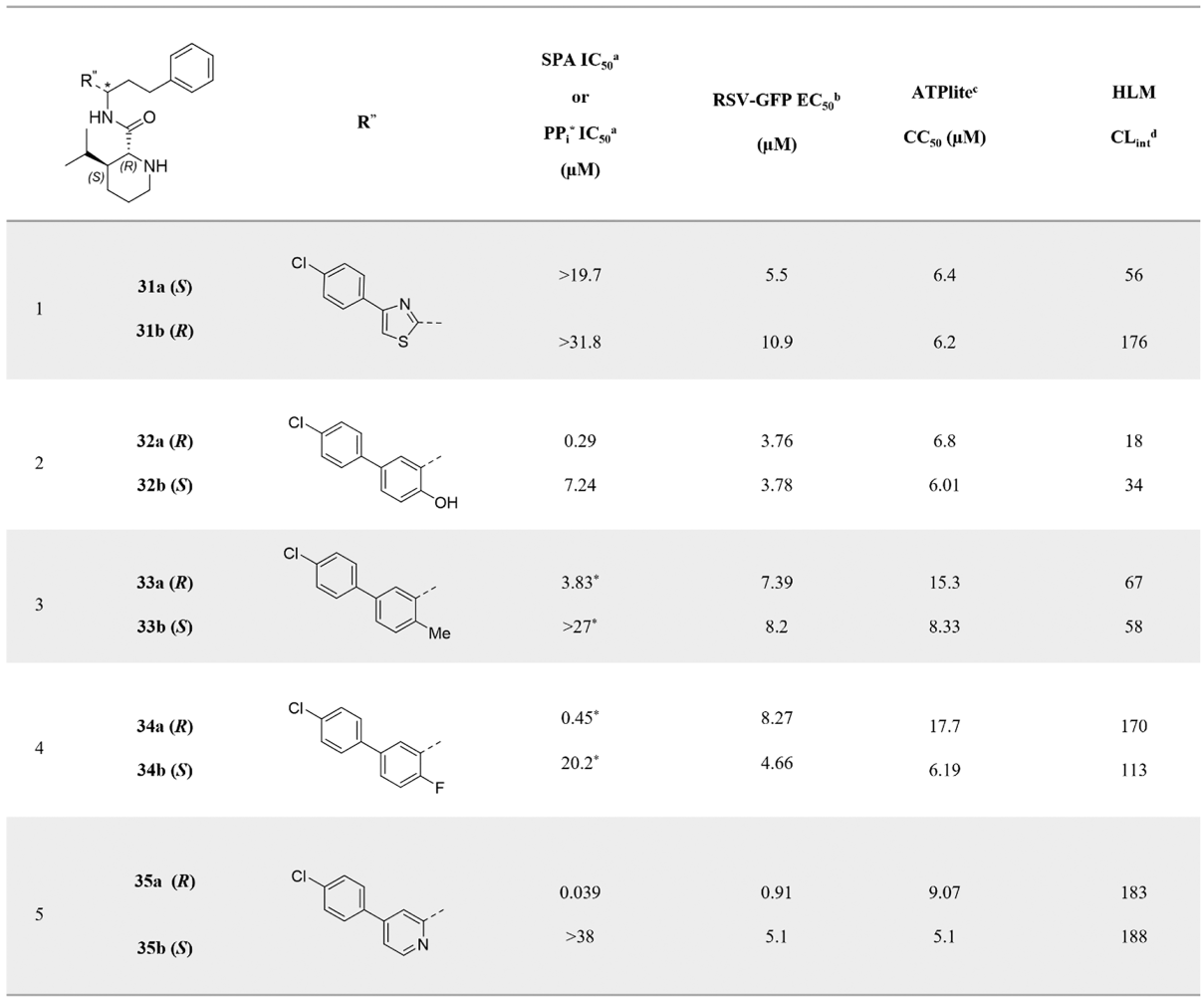
表3中的所有化合物均以外消旋混合物的形式合成,并在生物活性测试前通过超临界流体色谱法(SFC)进行了分离。随后通过核磁共振(NMR)构象研究(如下所述)确定了其绝对立体化学构型,并如表中所示进行标注。
尽管含有噻唑环的衍生物31a和31b没有活性(表3,条目1),但相应的苯酚类似物32a表现出令人满意的生化活性,其SPA IC50=290 nM(表3,条目2)。得益于其与苗头化合物16在结构上的差异,以及酰胺键和手性中心数量的减少,这一新型联芳基母核骨架展现出作为进一步研究方向的巨大潜力。此外,化合物32在人体肝微粒体中的代谢稳定性也优于化合物16。对羟基进行初步的结构-活性关系(SAR)修饰后,发现对位氟取代的类似物34a是一个良好的替代选择,其在生化和细胞实验中均显示出活性,尽管其选择性指数(SI)仅为2(表3,条目4)。另一方面,相应的甲基类似物 33a 和 33b 的两种对映异构体均无活性(表3,条目3)。有令人感兴趣的是,吡啶基化合物 35a 在生化实验中表现出两位数纳摩尔(double digit nM)级别的活性,在细胞实验中也显示出亚微摩尔(sub μM)的活性,但该化合物伴随有一定的细胞毒性,导致其选择性指数(SI)为10(表3,条目5)。
NMR构象分析同样用于合理解释联芳基系列的SAR。化合物16的3D溶液构象显示,其肽类主链是刚性的,保持了NH和C=O基团的相对取向,而每个单元中的“侧链”则表现出一定程度的柔性(图3a)。使用NMR温度系数方法可以确定在化合物16中没有酰胺形成分子内氢键。溶液中16的3D表示与冷冻电镜结构中发现的生物活性构象高度相似(图3a),这表明该分子在结合到靶标时不需要付出显著的构象能代价。
通过核磁共振(NMR)对SPARK骨架跃迁得到的化合物32a、33a、34a和35a进行了研究,以确定芳环上邻位取代的构象效应,从而解释所观察到的活性差异。在所有情况下,与实验NMR数据拟合最佳的构象模型均在哌啶环、酰胺基团以及乙基-芳基部分展现出相同的三维特征。该系列化合物之间最主要的区别在于邻位取代芳环的取向。
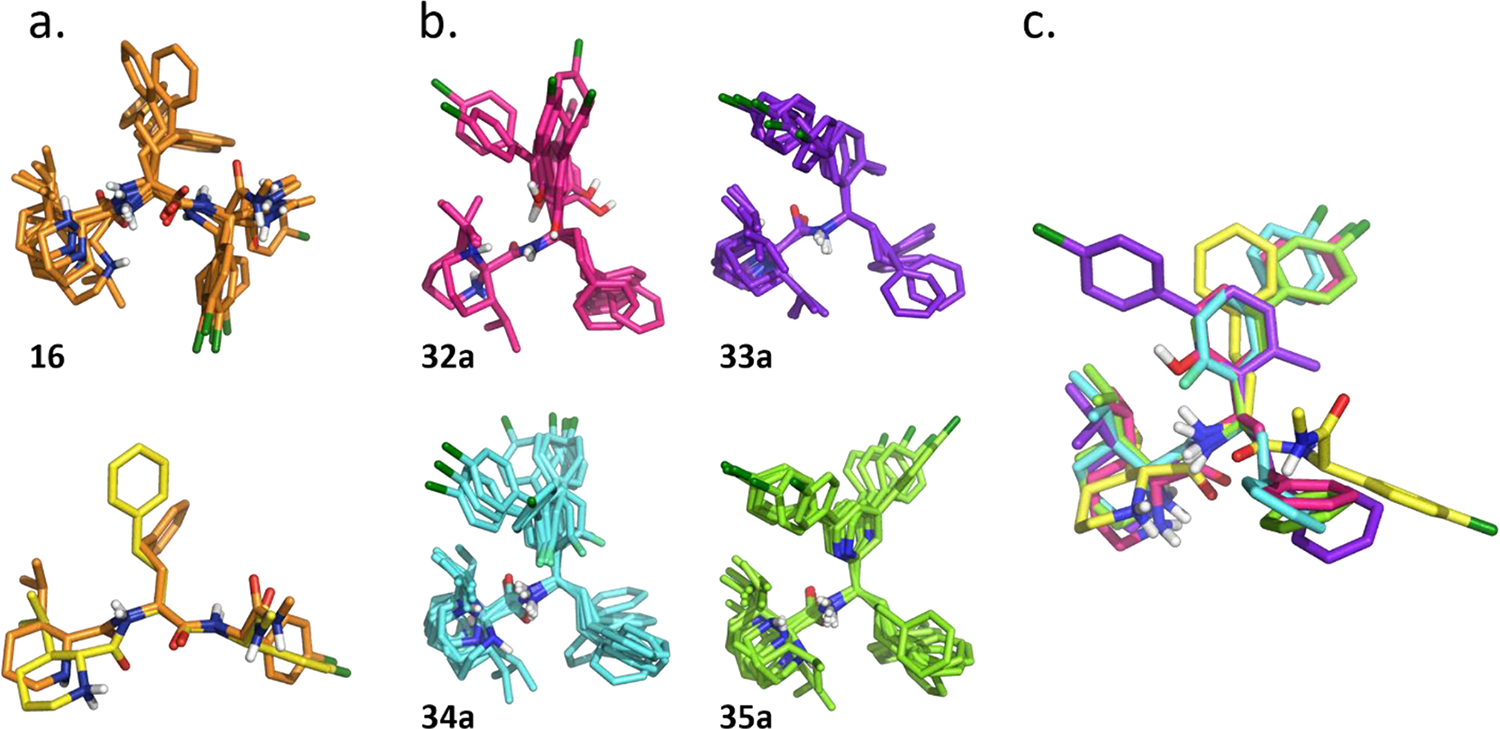
图3. NMR溶液构象。(a)上:化合物16的3D溶液构象集,来源于NMR数据以及通过REST-MD模拟生成的构象;下:代表性的NMR构象(橙色)与化合物16的冷冻电镜结构(黄色)的叠合图。(b)化合物32a、33a、34a和35a的3D溶液构象集合,同样来源于NMR数据以及通过REST-MD模拟生成的构象。(c)化合物16结合于靶标蛋白(PDB 9ECV)的冷冻电镜结构(黄色)与其中一个代表性的溶液构象叠合图:32a(粉色)、33a(紫色)、34a(青色)和35a(绿色)。
通过分析33a中邻位H与Me之间的NOE信号强度,以及32a、34a和35a中邻位H的NOE信号(见图3b),发现对于含有苯酚、氟原子和吡啶基取代的化合物(即32a、34a、35a),芳环可以在两种主要取向之间切换:其中OH、F或N基团要么位于“前方”(即生物活性构象),要么位于“后方”(非生物活性构象)。这三个分子能够采取生物活性构象的能力与其所表现出的高活性是一致的。
相比之下,化合物33a的结构更为刚性(见图3b,c),其甲基更倾向于朝向“后方”,可能是为了获得更大的立体位阻释放;相应地,联芳基则朝向“前方”,处于非生物活性的取向。这种取向可能会带来巨大的能量惩罚,包括构象重排和构象张力,这可能部分解释了33a活性下降的原因。
总的来说,通过SPARK骨架跃迁方法,成功将三肽母核替换为新型骨架,显著减少了酰胺键和手性中心数量。这些新骨架化合物在生化和/或细胞实验中仍保持活性,并表现出改善的人肝微粒体体外清除率。初步的药物化学研究工作证实,这些新的化学类型具有进一步开发的前景,可以作为未来研发呼吸道合胞病毒(RSV)聚合酶抑制剂有价值的起点。
结论
在本研究中,来自强生的研究人员利用已知的封帽结构域结合物JNJ-8003进行了一次有针对性的DNA编码库(DEL)筛选,发现了一种结构新颖的三肽母核RSV聚合酶抑制剂,即苗头化合物16。为了克服16潜在的代谢不稳定问题,通过平行药物化学策略和SPARK骨架跃迁方法,成功将三肽母核替换为新型骨架,显著减少了酰胺键和手性中心数量。这些新骨架化合物在生化和/或细胞实验中仍保持活性,并表现出改善的人肝微粒体体外清除率。总的来说,初步的药物化学研究工作证实,这些新的化学类型具有开发前景,可以作为未来研发呼吸道合胞病毒(RSV)聚合酶抑制剂有价值的起点。证明了Spark在DEL Hit-to-Lead过程中丰富骨架化学多样性的强大能力。
联系我们
想在自己的项目中亲自使用SPARK进行生物等排体替换,请联系我们获取免费的试用版;或者联系我进行在线演示;你还可以采购软件或委托研究与我们进行项目合作。
- 电邮:info@molcalx.com
- 电话:020-38261356
文献
- Chaw, P. S.; Wong, S. W. L.; Cunningham, S.; Campbell, H.; Mikolajczyk, R.; Nair, H.; RESCEU Investigators. Acute Lower Respiratory Infections Associated With Respiratory Syncytial Virus in Children With Underlying Congenital Heart Disease: Systematic Review and Meta-analysis. J. Infect. Dis. 2020, 222 (Suppl 7), S613−S619.
- Chatzis, O.; Darbre, S.; Pasquier, J.; Meylan, P.; Manuel, O.; Aubert, J. D.; Beck-Popovic, M.; Masouridi-Levrat, S.; Ansari, M.; Kaiser, L.; Posfay-Barbe, K. M.; Asner, S. A. Burden of severe RSV disease among immunocompromised children and adults: a 10 year retrospective study. BMC Infect. Dis. 2018, 18 (1), 111.
- Shi, T.; Denouel, A.; Tietjen, A. K.; Campbell, I.; Moran, E.; Li, X.; Campbell, H.; Demont, C.; Nyawanda, B. O.; Chu, H. Y.; Stoszek, S. K.; Krishnan, A.; Openshaw, P.; Falsey, A. R.; Nair, H.; RESCEU Investigators. Global Disease Burden Estimates of Respiratory Syncytial Virus-Associated Acute Respiratory Infection in Older Adults in 2015: A Systematic Review and Meta-Analysis. J. Infect. Dis. 2020, 222 (Suppl 7), S577−S583.
- Li, Y.; Johnson, E. K.; Shi, T.; Campbell, H.; Chaves, S. S.; Commaille-Chapus, C.; Dighero, I.; James, S. L.; Mahe, C.; Ooi, Y.; Paget, J.; van Pomeren, T.; Viboud, C.; Nair, H. National burden estimates of hospitalisations for acute lower respiratory infections due to respiratory syncytial virus in young children in 2019 among 58 countries: a modelling study. Lancet Respir Med. 2021, 9 (2), 175−185.
- Chaw, P. S.; Hua, L.; Cunningham, S.; Campbell, H.; Mikolajczyk, R.; Nair, H.; RESCEU Investigators. Respiratory Syncytial Virus-Associated Acute Lower Respiratory Infections in Children With Bronchopulmonary Dysplasia: Systematic Review and Meta-Analysis. J. Infect. Dis. 2020, 222 (Suppl 7), S620−S627.
- Yu, X.; Abeywickrema, P.; Bonneux, B.; Behera, I.; Anson, B.; Jacoby, E.; Fung, A.; Adhikary, S.; Bhaumik, A.; Carbajo, R. J.; De Bruyn, S.; Miller, R.; Patrick, A.; Pham, Q.; Piassek, M.; Verheyen, N.; Shareef, A.; Sutto-Ortiz, P.; Ysebaert, N.; Van Vlijmen, H.; Jonckers, T. H. M.; Herschke, F.; McLellan, J. S.; Decroly, E.; Fearns, R.; Grosse, S.; Roymans, D.; Sharma, S.; Rigaux, P.; Jin, Z. Structural and mechanistic insights into the inhibition of respiratory syncytial virus polymerase by a non-nucleoside inhibitor. Commun. Biol. 2023, 6 (1), 1074.
- Grosse, S.; Cooymans, L.; Embrechts, W.; McGowan, D.; Jacoby, E.; Stoops, B.; Gupta, K.; Ackermann, M.; Alnajjar, S.; Guillemont, J.; Jin, Z.; Kesteleyn, B.; Matcha, K.; Sriboonyapirat, P.; Truong, A.; Van Den Berg, J.; Yu, X.; Herschke, F.; Roymans, D.; Raboisson, P.; Rigaux, P.; Jonckers, T. H. M. Discovery of gem-Dimethyl-hydroxymethylpyridine Derivatives as Potent Non-nucleoside RSV Polymerase Inhibitors. J. Med. Chem. 2024, 67 (16), 13723−13736.
- Sean M. Carney, Sandrine Grosse, Yanting Yin, Minh T. Tran, Jay H. Kalin, Edgar Jacoby, Amy Fung, Nicholas Simmons, Xiaoming Xie, Anusarka Bhaumik, Rodrigo J. Carbajo, Madison Piassek, Robyn Miller, Lili Hu, Cynthia Lemmens, Ferdinand H. Lutter, Serge Pieters, Geert Rombouts, Irene Vetrano, Daniel Oehlrich, Sonia Tomaso, Kate Lozada, Miguel Osorio Garcia, Brandon Anson, Suzanne De Bruyn, Constance Smith-Monroy, Jean-Marc Neefs, Nádia Conceição-Neto, Bart Kesteleyn, Roberto Fino, Bart Stoops, Herman van Vlijmen, Aaron N. Patrick, Xiaodi Yu, Victoria Wong, Daniel J. Krosky, Pravien Abeywickrema, Rodrigo F. Ortiz-Meoz, Stephen W. Mason, Zhinan Jin, Sujata Sharma, and Tim H. M. Jonckers. DNA-Encoded Library Screen Identifies Novel Series of Respiratory Syncytial Virus Polymerase Inhibitors. Journal of Medicinal Chemistry 2025 68 (6), 6407-6430
DOI: 10.1021/acs.jmedchem.4c02906 - Goodnow, R. A., Jr.; Dumelin, C. E.; Keefe, A. D. DNA-encoded chemistry: enabling the deeper sampling of chemical space. Nat. Rev. Drug Discov 2017, 16 (2), 131−147.
- Gironda-Martinez, A.; Donckele, E. J.; Samain, F.; Neri, D. DNA-Encoded Chemical Libraries: A Comprehensive Review with Succesful Stories and Future Challenges. ACS Pharmacol Transl Sci. 2021, 4 (4), 1265−1279.
- Reiher, C. A.; Schuman, D. P.; Simmons, N.; Wolkenberg, S. E. Trends in Hit-to-Lead Optimization Following DNA-Encoded Library Screens. ACS Med. Chem. Lett. 2021, 12 (3), 343−350.
- Diao, L.; Meibohm, B. Pharmacokinetics and pharmacokinetic-pharmacodynamic correlations of therapeutic peptides. Clin Pharmacokinet 2013, 52 (10), 855−868.
- Lai, X.; Tang, J.; ElSayed, M. E. H. Recent advances in proteolytic stability for peptide, protein, and antibody drug discovery. Expert Opin Drug Discov 2021, 16 (12), 1467−1482.
- Spark™, version , Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/spark/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665-676
- Kumar, K.; Chupakhin, V.; Vos, A.; Morrison, D.; Rassokhin, D.; Dellwo, M. J.; McCormick, K.; Paternoster, E.; Ceulemans, H.; DesJarlais, R. L. Development and implementation of an enterprise-wide predictive model for early absorption, distribution, metabolism and excretion properties. Future Med. Chem. 2021, 13 (19), 1639−1654.
- Ertl, P.; Schuffenhauer, A. Estimation of synthetic accessibility score of drug-like molecules based on molecular complexity and fragment contributions. J. Cheminform. 2009, 1 (1), 8.



