摘要:Spark数据库含有来自有商品可供购买的筛选化合物、科技文献、专利文献、商品可供购买的试剂、小分子晶体结构(结晶学开放数据库和剑桥结构数据库)以及理论环系(VEHICLe)等等。本文详细说明了这些片段库与试剂库的组成、来源以及分布特点,并介绍了新增的农业化学品数据库。
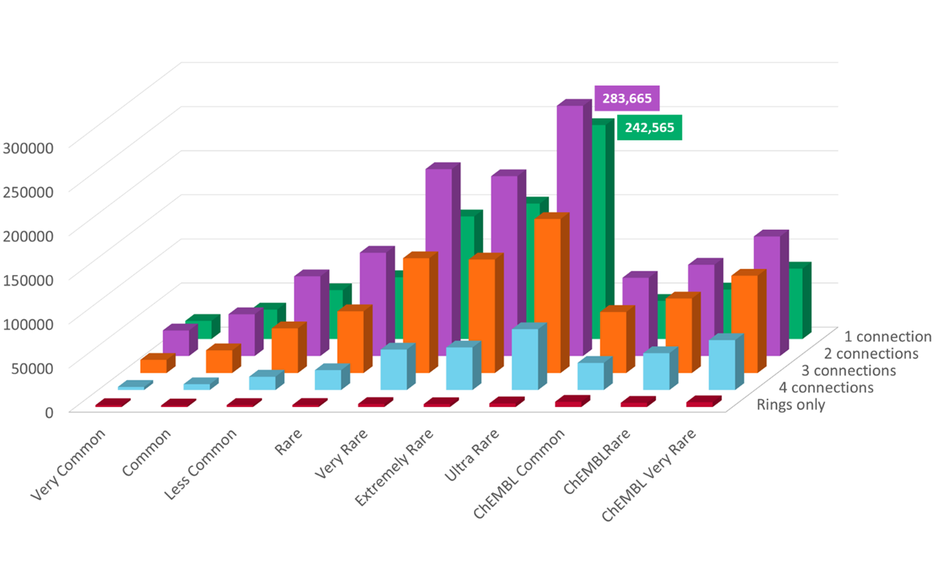
在本次Spark数据库发布中,除了所有片段数据库均已更新之外,还新增一个专注于农业化学品的合集“Agrochemical”。此次Spark片段数据库的更新和扩展,让用户可以访问数百万个构象丰富的片段,从而提供广泛的生物等排体选择,帮助您利用Spark——Cresset的生物等排体替换工具——探索新思路,推动项目进展。表1总结了本次更新后的数据库里的片段分布情况。
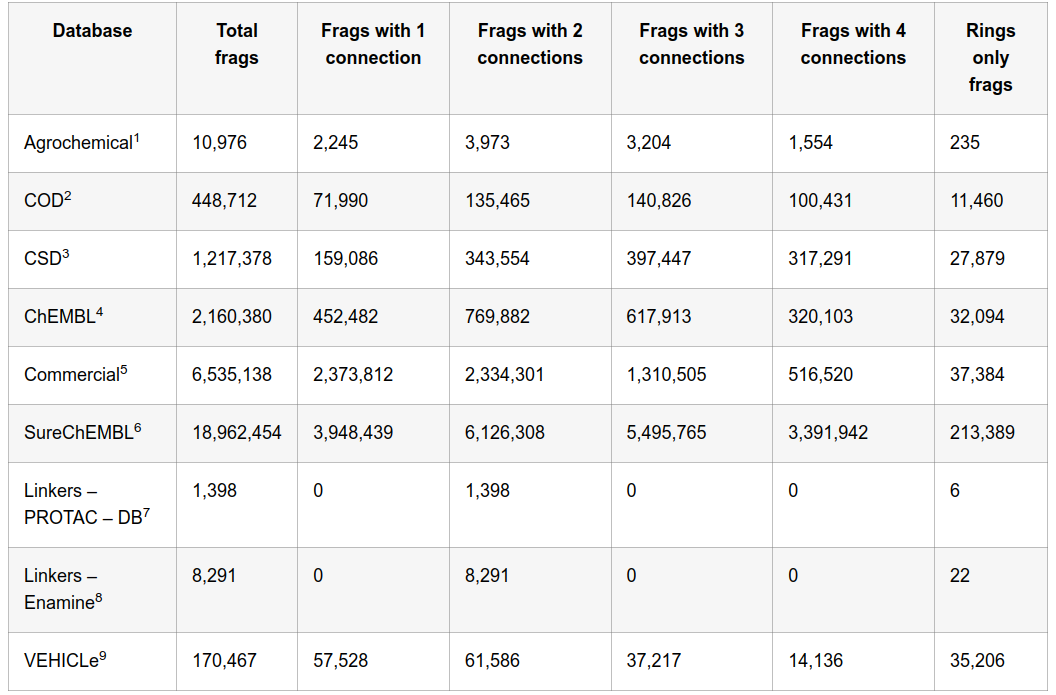
表1. 在本次更新的Spark片段数据库中,片段的总数量(“总片段数”)以及根据片段连接点数量(“含1/2/3/4个连接点的片段”)或连接点模式(“仅环状片段”)进行分类的片段分布情况。
更新的数据库包括仅含环状结构的片段以及具有1到4个连接点的片段,这些片段可用于多种实验,包括骨架跃迁、R-基团探索、片段生长与连接、大环化以及晶体学中水分子的替换等应用。
此外,还有约50万个具有单一连接点的片段,这些片段来源于市售试剂,适用于R-基团探索实验,每月更新一次。根据对试剂进行碎片化所采用的反应类型,目前共有23个“试剂”合集,对应于药物化学中广泛使用的反应。
新增农业化学品数据库
新增的农业化学品专题数据库“Agrochemical”包含了近11,000个构象丰富的片段,这些片段来源于PubChem中的“Agrochemical”类别,涵盖了农业中使用的各类农用化学品,包括化学肥料、杀虫剂等。
使用专注于农业化学品的片段数据库,可确保您的Spark实验结果具有适合应用于农用化学品领域的性质特征,如表2所示。
在对化合物进行片段化之前,PubChem化合物按如下方法进行了预处理:
- 对1,3-二酮类化合物进行枚举并转化为烯醇式结构。
- 去除所有前药型杀虫剂的掩闭基团
- 应用了已知的其它转化反应a-c:
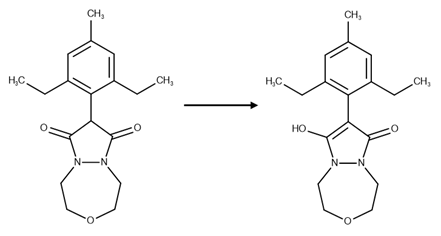
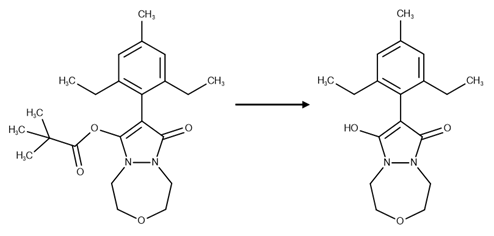
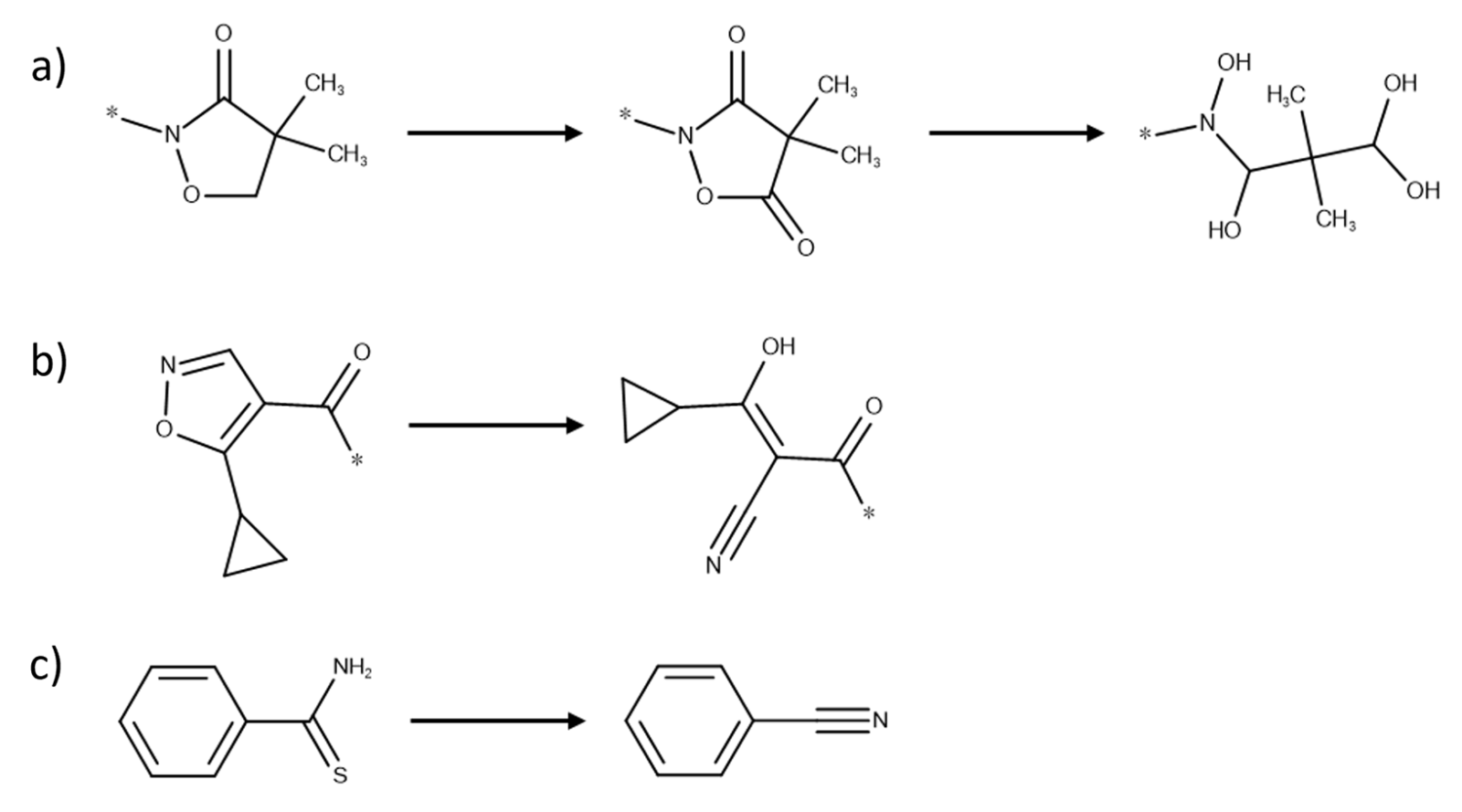
“Agrochemical”数据库中片段的理化性质如下表2所示。
表2. Spark新增“Agrochemical”数据库的片段性质分布。
| Database | Fragments | NC Range | HA Range | MW Range | RB Range |
|---|---|---|---|---|---|
| PubChem | 10,976 | 1 – 30 | 1 – 16 | 12 – 249 | 0 – 5 |
数据库中的片段数量(Fragments)、每个片段的构象数量(NC)、每个片段的重原子数量(HA)、片段的分子量(“MW”)、可旋转键的数量(“RB”)
在你的项目中使用新的Spark数据库
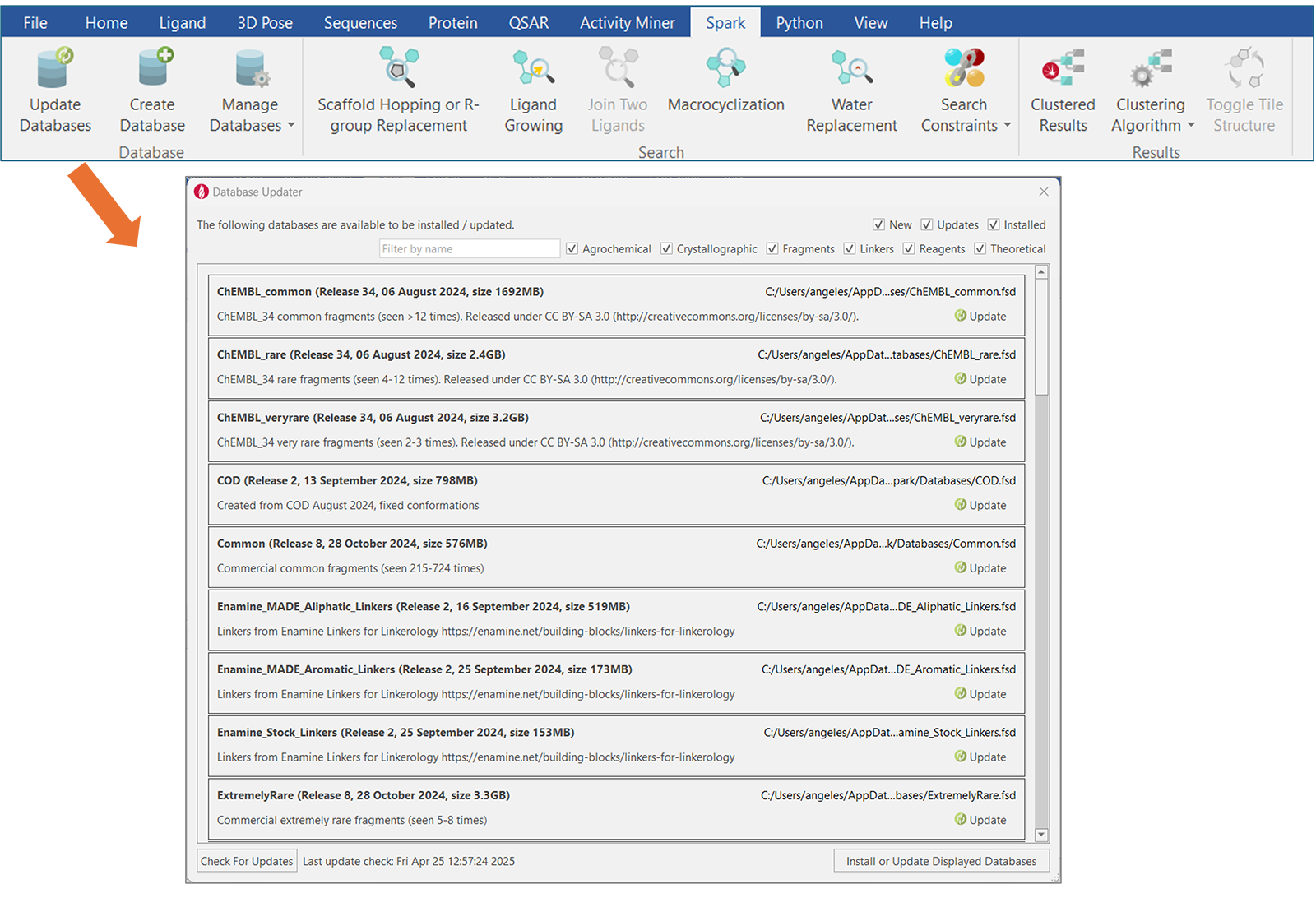
所有Cresset发布的Spark数据库均向所有Spark用户开放,可以从Flare中Spark选项卡下的“数据库更新器”(Database Updater)下载,如图1和图2所示。
图1. 更新的片段和试剂数据库可以从“数据库更新器”(Database Updater)中下载,通过在右上角选择“更新”(Updates)类别进行筛选。
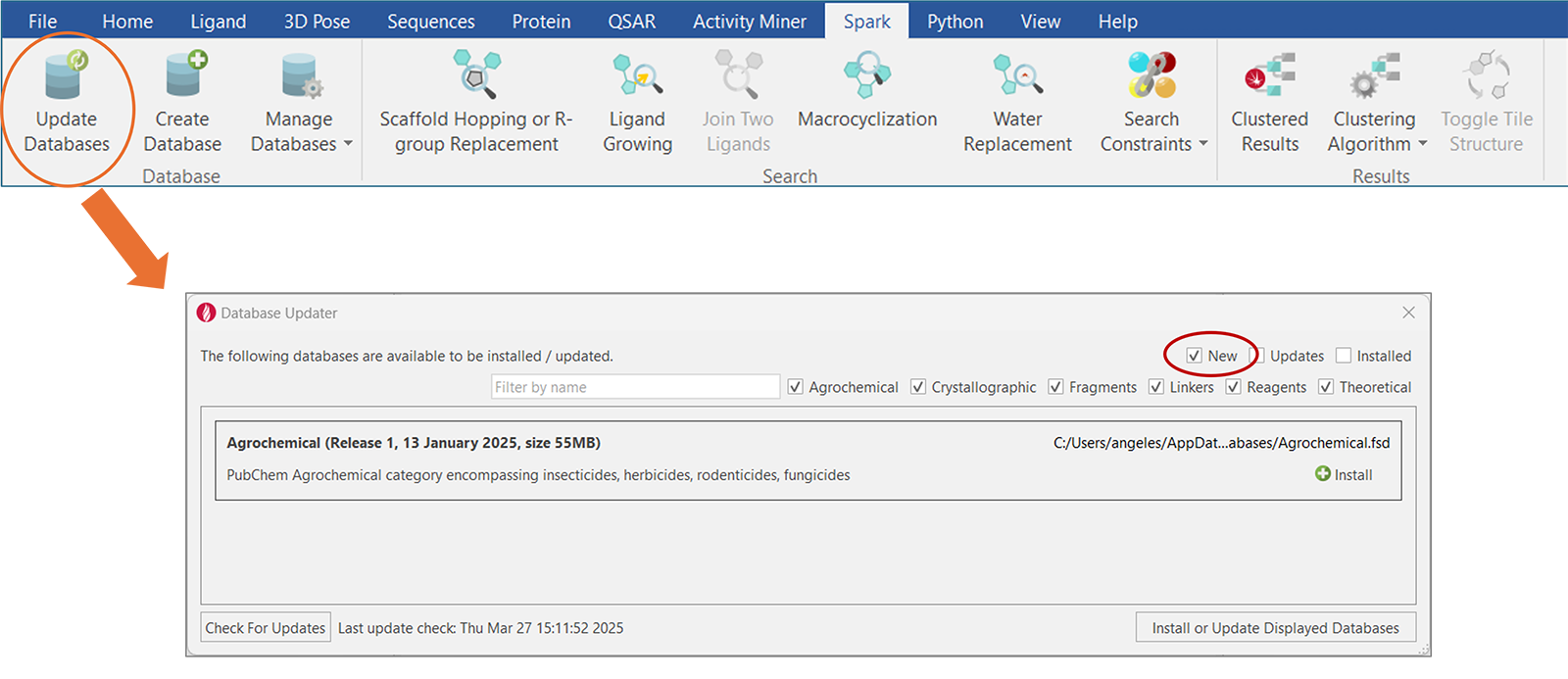
新增的“Agrochemical”数据库也可以从“数据库更新器”中的“New”类别下载,如图2所示。
图2:新增的“Agrochemical”数据库可以从“数据库更新器”中下载,通过在右上角选择“新”(New)类别进行筛选。
如果您在Flare中使用集成的Spark,在采用“配体相似性”打分方法(基于Cresset XED力场)进行计算时,系统将使用与蛋白质计算一致的距离依赖介电函数。为了保持一致性,此次发布的Spark片段和试剂数据库也是在距离依赖介电函数的基础上构建的。
使用更新后的数据库合集时,若采用独立版Spark,需使用Spark 10.8或更高版本。请联系我们以获取最新版本的下载链接。请注意,从Spark 10.8开始,本地Spark中的场点计算也将使用距离依赖介电函数,以保持与Flare中集成的Spark一致。
在你自己的项目中亲自试用Spark
如果您目前尚未使用Spark,但希望尝试了解其功能,请联系我们,了解Spark如何为您的项目带来积极影响。
- 电邮:info@molcalx.com
- 电话:020-38261356
参考文献
- https://pubchem.ncbi.nlm.nih.gov/
- http://www.crystallography.net/cod/
- https://www.ccdc.cam.ac.uk/solutions/csd-system/components/csd/
- https://www.ebi.ac.uk/chembl/
- https://www.emolecules.com/products/screening-compounds
- https://ftp.ebi.ac.uk/pub/databases/chembl/SureChEMBL/
- http://cadd.zju.edu.cn/protacdb/
- https://enamine.net/building-blocks-mob/linkers-for-linkerology
- Pitt, W. R.; Parry, D. M.; Perry, B. G.; Groom, C. R. Heteroaromatic Rings of the Future. J. Med. Chem. 2009, 52 (9), 2952–2963 https://doi.org/10.1021/jm801513z1
- https://www.emolecules.com/products/building-blocks
- Vinter, J.G. Extended electron distributions applied to the molecular mechanics of some intermolecular interactions. J Computer-Aided Mol Des 1994, 8, 653–668. https://doi.org/10.1007/BF00124013



