摘要:Spark V10.6是骨架跃迁和生物等排体替换工具SPARK的最新版。它赋能药物化学家和计算化学家们在药物发现项目中使用新方法产生新思路。新增对接功能使SPARK能够直接从蛋白结合位点出发找到与之相匹配的片段,显著地提高了找到生物等排体的性能,改进了高级搜索选项可以轻松地以设置分子大小标准的定义。
Spark V10.6是骨架跃迁和生物等排体替换工具SPARK最新版本,赋能药物化学家和计算化学家们在药物发现项目中使用新的、改进过的方法产生新思路。
亮点
- 新增对接功能使SPARK能够直接从蛋白结合位点出发找到与之相匹配的片段
- 新的、改进过的计算方法,显著地提高了找到令人感兴趣生物等排体的性能
- 改进的高级搜索选项,可以轻松地设置分子大小标准的定义
SAPRK的分子对接功能
新增的对接功能使Spark可以直接从蛋白活性位点中找到配体-蛋白相互作用相符的片段。用这种方法可以使配体和/或片段往靶蛋白未占据口袋方向生长,因此找到的新化合物与蛋白活性位点的相互作用模式也是现有的起始分子或参考分子未曾有过的。
图1展示了该新工作流的细节。首先,SPARK将起始分子中你希望被替换的部分裁掉(图1,A-B)。然后将新片段连接到在上一步被截短的起始分子上,并在Cresset XED力场计算的蛋白静电势引导下以合理的取向放置到蛋白的结合位点里(图1,C)。 然后,用Lead Finder的“score only”对接模式来优化新分子的pose(图1,D)。最后,用Lead Finder的“Rank score”打分函数对最佳结果进行优先级排序,该打分函数经优化后可以正确重现已知配体的结合模式。
此外,通过设置配体-蛋白质相互作用的对接约束,可以轻松获得强调该约束的结果分子。
Spark新增的分子对接功能作为插件提供,以添加到传统的生物等排体替换方法中。如果您有兴趣,请联系我们。

图1. Spark的Docking工作流(PDB code:6TCU) A)在起始分子中选择要被替换部分;B)删除所选择的片段; C)将新的片段接到在上一步被截短的起始分子上,并以有意义的取向放置好;D)使用 LeadFinder优化新结果分子的pose
改进的计算方法
基于配体场/形状相似性的“传统”Spark方法的改进包括:
- 更多数量的片段进入最后的打分阶段
- 形状相似性打分方法的初始阶段新增“Very Accurate but Slow”的方法
- 从配体-蛋白复合物开始的搜索可以更高效地处理蛋白的排斥体积
这些改进提高了Spark搜索令人感兴趣生物等排体的准确性。例如,在根据盐皮质激素受体调节剂(PDB 5MWP)的X衍射结构进行的骨架跃迁实验中,Spark发现了与其他已知的盐皮质激素配体极为相似的结构(图2)。 结果列表还显示,还富集到高生物等排体因子(BIF%)的其它可选的替换结构。这些改进使得SPARK典型的R-基团替换实验能多识别出30%结果,并具有相当好的BIF%。
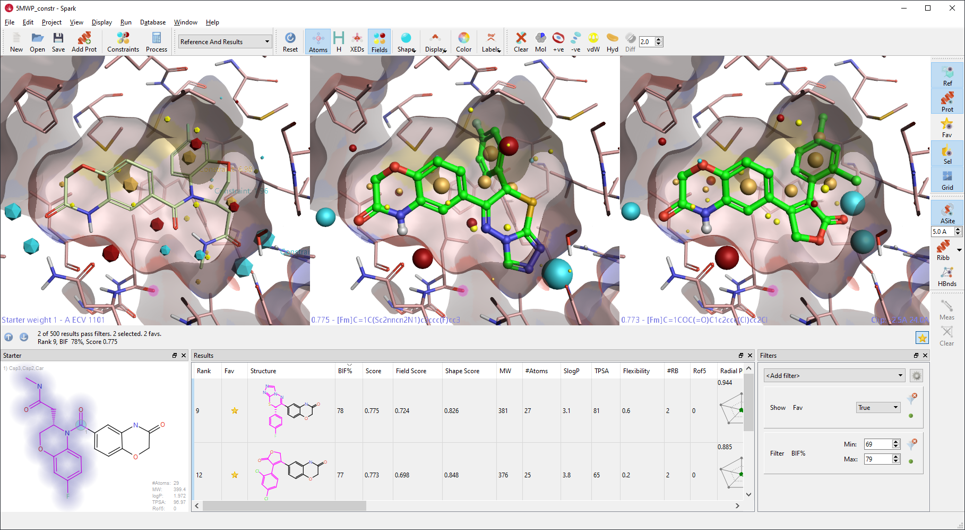
图2. Figure 2: 用PDB 5MWP进行盐皮质激素受体骨架跃迁实验发现一系列结构多样的、BIF%指高的新骨架
改进的高级搜索选项
在搜索过程中进行适当的过滤是一种好方法,它可以减少Spark计算所需搜索的空间、减少计算时间并使实验聚焦于您真正想要的结果上。 这在片段增长实验中或者在使用对接方法时,您希望分子在一定范围内生长时,特别有用。
为了帮助您解决此问题,我们改进了高级Spark搜索选项(图3)的Size Criteria的控制面板。现在,该面板报告了片段和结果分子的重原子数、MW和可旋转键数。将此面板与“Starter”视窗以及“Advanced Filters”中的信息结合使用,可以为Spark实验的结果分子设置理想的理化特性。
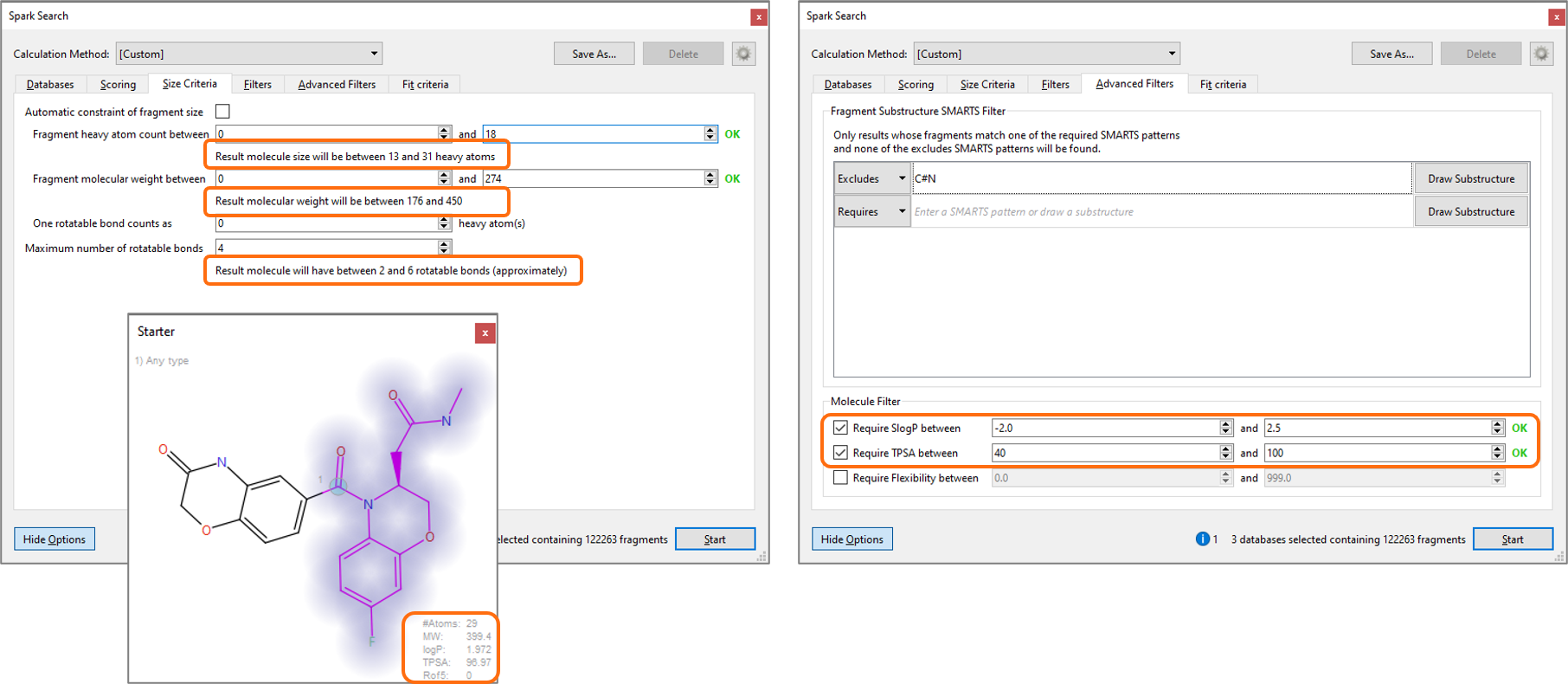
图3:改进的Size Criteria面板(左),与Starter window(中)以及Advanced Filters(右)的信息组合使用,可以非常便利地在SPARK实验中设置结果分子的理化性质特征
其它改进
本版SPARK除了在技术上重大提升之外,在用户界面上也有很多显著地的提升:
- 全新的片段数据库,有690万片段可供搜索
- 增强了结果列表中的2D结构展示
- 能够区分骨架跃迁实验结果中不同的附着点
- 数据库生成功能,新增还原胺化试剂模式和构象搜寻的新选项
- 在搜索和数据库更新程序窗口中更清晰的数据库信息
- 下载数据库更快
- 预定义常见化学基团/官能团的过滤器:比如环、芳环、手性原子、氢键受体/供体等等
- 导入/导出筛选器、自定义属性和径向绘图属性
SPARK 10.6演示:突破现有技术,发现新的IP
SPARK 10.6:分子对接打分特性介绍
想在项目中使用Spark吗?
SPARK现有用户会收到一封关于如何升级到V10.6的电子邮件;若您当前还不是SPARK用户的话,可以联系我们获取试用版。



