摘要:相对自由能微扰(FEP)方法在制药行业中变得越来越流行。 然而,尽管药物发现周期内存在时间限制,但在部署此类方法时应谨慎行事:因为蛋白的准备和系统设置会极大地影响自由能预测的准确度。本文主要给出实例证据来佐证该结论。
作者:硅康科研/硅康SiliconTX
声明:本文经公众号”硅康SiliconTX”徐华锋老师许可转载。
目前,炼金术自由能计算在制药领域越来越受欢迎。值得注意的是,药物靶标蛋白结构的处理及其设置对自由能计算的准确度有着很大的影响。本文主要给出实例证据来佐证该结论。
该项工作选取了NF-Kappa-B-Inducing Kinase (NIK)作为靶标蛋白及其五个结构上相似的小分子配体,如图1(A)所示。图1(B)展示了三种不同的蛋白质处理方法,该三种方法都是经验丰富的计算化学家所使用的。
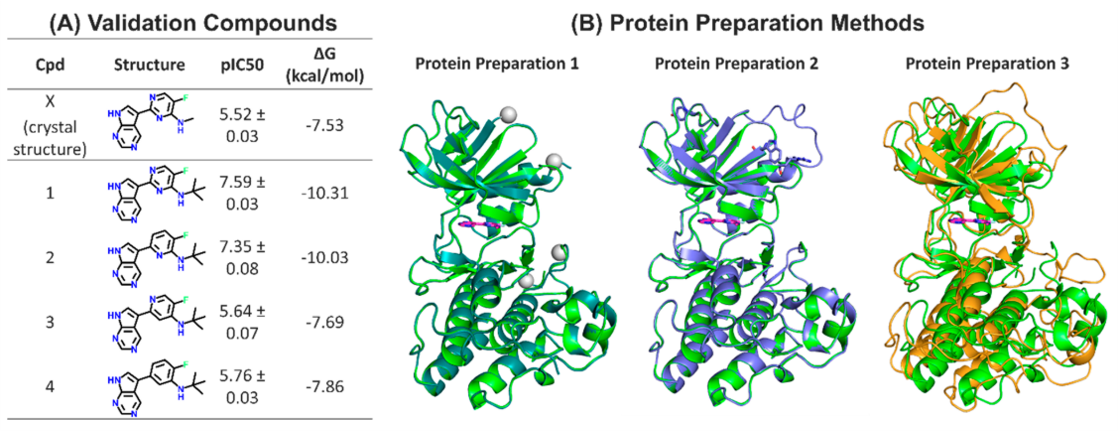
图1:靶标蛋白NIK及其五个小分子配体:(A)实验值pIC50值由三次重复实验取平均得到,及其由该pIC50值转化得到的∆G值;(B) 三种不同的蛋白质结构处理方法,分别由青色、紫色和橙色表示,绿色代表NIK的晶体结构,粉红色代表晶体结构中的配体小分子X。
蛋白质处理方法1(图1B,青色):由具有激酶和分子动力学经验的应用科学家(来自薛定谔公司)独立开发,是处理激酶体系的最佳实践。该方法使用了NIK晶体结构(图1B,绿色),链断裂处加上cap,晶体结构突变保持不变(图1B,白色球体)。
蛋白质处理方法2(图1B,紫色):是由一位在激酶体系方面经验丰富的计算化学家(来自Janssen研发公司)在另一位在分子动力学方面经验丰富的应用科学家(来自薛定谔公司)的指导下开发的,并使用薛定谔软件Prime模块对缺失环进行建模并将突变残基变为野生型残基。
蛋白质处理方法3(图1B,橙色):是由一位Janssen研发公司的计算化学家开发的,他没有激酶靶点的研究背景,但具有丰富的分子模拟经验,涉及到从其他各种公开的NIK晶体结构中模拟缺失环,并将突变残基变为野生型残基以及使用MD来预先平衡体系。
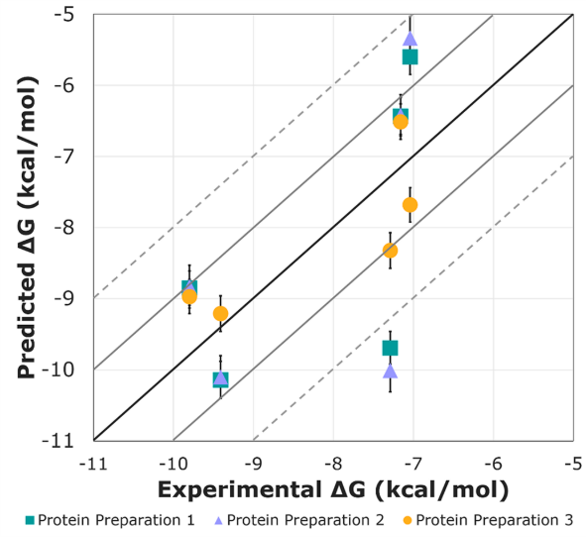
图2:三种蛋白处理方法的FEP计算结果比较
蛋白质处理方法1的FEP+结果表明,该方法所预测的自由能值与实验值的相关性较差,R2为0.40, MUE为1.87±0.36 kcal/mol, RMSE为2.22±0.37 kcal/mol。蛋白质处理方法2与方法1的结果相似,R2为0.34, MUE为2.10±0.41 kcal/mol, RMSE为2.48±0.42 kcal/mol。蛋白质处理方法3的FEP+结果给出,R2为0.64, MUE为1.00±0.17 kcal/mol, RMSE为1.15±0.16 kcal/mol。前两种并不严格的蛋白质处理方法所给出的预测结果中,cpd1-3的预测结果与实验值的偏差在1 kcal/mol范围内;但是cpd4的预测结果并不理想,其误差大于2 kcal/mol。晶体结构中的配体小分子X,由蛋白质处理方法3预测效果良好,准确度在1 kcal/mol以内。
表1. 三种蛋白准备方法对FEP计算结果的影响
| 蛋白准备方法 | R2 | MUE (kcal/mol) | RMSE (kcal/mol) |
|---|---|---|---|
| 处理方法1 | 0.40 | 1.87±0.36 | 2.22±0.37 |
| 处理方法2 | 0.34 | 2.10±0.41 | 2.48±0.42 |
| 处理方法3 | 0.64 | 1.00±0.17 | 1.15±0.16 |
蛋白质处理方法1和2都使用了晶体结构,而蛋白质处理方法3使用了分动力学模拟来预先平衡体系。晶体结构可能存在影响结构坐标的人为因素,主要包括结晶条件和单位细胞堆积。NIK结构还有两个潜在的问题,即缺失环残基和残基突变,尽管它们离配体结合位点较远,但它们都会影响动力学模拟。这些差异可能对NIK特别重要,因为缺失的残基区域组成了激酶激活环。最后,活性位点内的水分子在不同的处理方法中也有所不同。蛋白质处理方法1和2,在做FEP+计算时,晶体结构中保留了一个水分子。在蛋白质处理方法3中,配体周围522Å内的所有水分子都被保留在MD模拟中。这些水分子的存在与否已被证明会影响自由能计算结果,尤其是在激酶体系中。这些差异可能会影响体系的动力学模拟以及由此产生的自由能结果预测;然而,对于不同的蛋白质-配体体系,此类分析的结果会有所不同。
该文作者提醒我们,我们需要在靶标蛋白结构的处理与体系准备上,格外注意,毕竟磨刀不误砍柴功。即使商业软件的可用性使得炼金术自由能计算在体系准备和结果分析方面都变得简单,但是成功地应用该方法仍然需要比较专业的经验。实际的计算机驱动药物研发项目将需要计算化学家(对研究体系和理论方法有着丰富的经验)和药物化学家之间的密切合作。
文献
- Shih, A. Y.; Hack, M.; Mirzadegan, T. Impact of Protein Preparation on Resulting Accuracy of FEP Calculations. J. Chem. Inf. Model. 2020, 60 (11), 5287–5289. https://doi.org/10.1021/acs.jcim.0c00445.



