摘要:本文介绍了最新版本Flare V5的功能与特性,包括:整合了基于配体的方法Forge;基于结构与基于配体方法的协同使用;GIST水分子分析方法;增强的FEP相对结合自由能计算;更快速的分子动力学模拟与增强的结果分析表单;基于场的药效团模拟与生物活性构象预测;全功能Python API。
我们很高兴的宣布,Flare V5现在正式发布。Cresset药物设计综合平台具有新的技术与增强的方法、整合了基于配体和蛋白的方法以及新的和改进的GUI功能,以提高可用性。
前沿的QSAR方法
在此版本中,Flare已从基于结构的解决方案演变为综合药物设计平台。在该平台中,基于配体和基于结构的方法可以完全协同工作。在Flare中,您可以找到所有您喜欢的定性和定量结构-活性关系 (SAR) 方法,但更好的是,这些方法皆可得益于蛋白质信息的使用、改进的GUI和Flare Python API的灵活性。
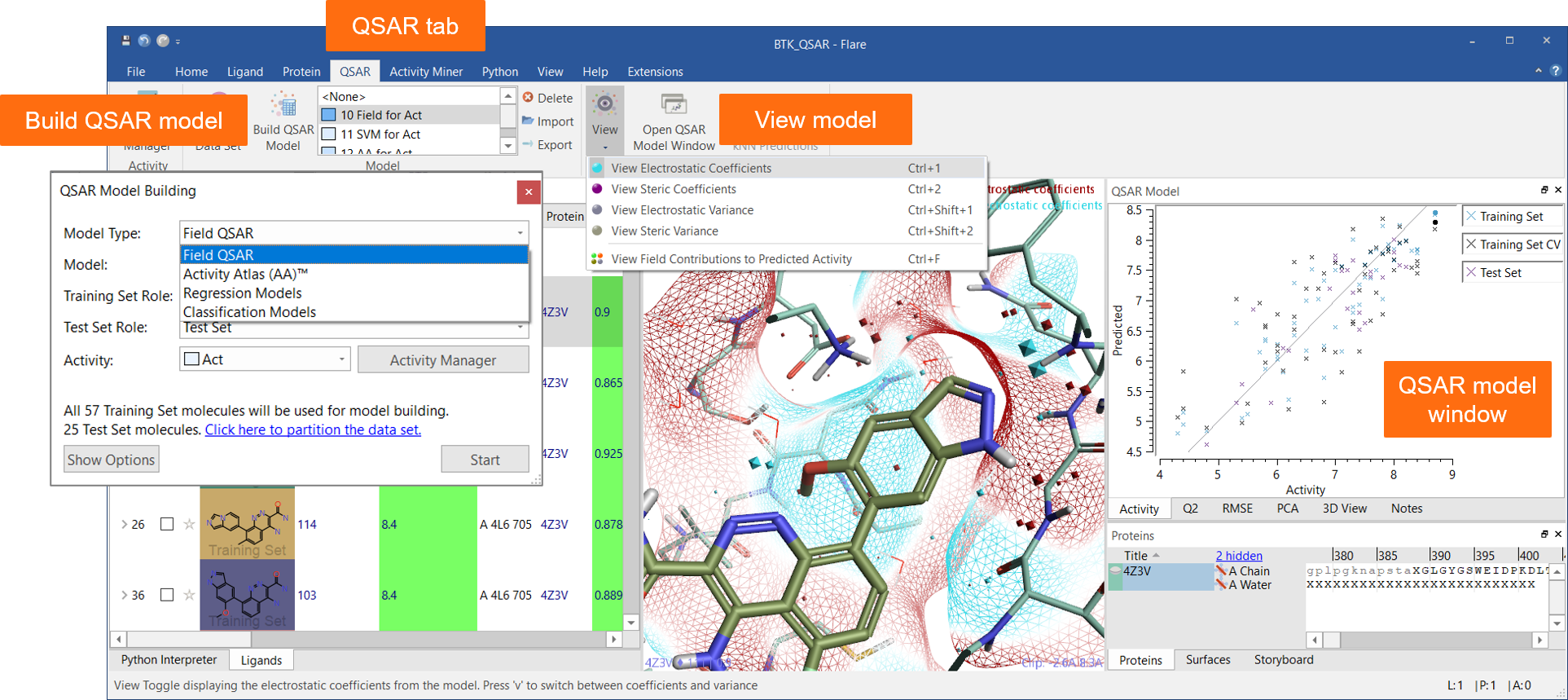
新的“QSAR”选项卡菜单(图 1)可以轻松访问所有功能,以使用多种稳健方法构建定量和定性 SAR 模型:
- 基于场的QSAR对叠合好的分子使用表征形状和静电特性的3D描述符建模,借助信息丰富的可视化,不但提供预测性模型,而且还提供解释性模型。
- 使用支持向量机、相关向量机、随机森林、k 最近邻的机器学习回归和分类,还具有“自动”模型构建选项,可自动运行所有方法并选择具有最佳统计数据的模型。
- Activity Atlas生成定性的3D等值图 ,将您的数据总结为SAR呈现在一张图里。
可以在专用的“QSAR model”窗口中查看相关统计数据和模型信息。
图1:QSAR选项卡和QSAR模型窗口是Flare构建QSAR模型和结果可视化的无缝界面。
Flare Python API可以轻松地将Flare与外部描述符计算器连接起来,并将您喜欢的描述符作为附加列导入到Flare配体表单中,从而促进机器学习模型的开发,以预测相关的ADMET属性。
专用选项卡提供了Activity Miner的增强界面,Flare组件使得基于活性悬崖分析方法能够快速地对复杂SAR进行分析。 数据的多种视图(图 2)可帮助您找到SAR中的关键分子对。对于每一对分子,Activity Miner会向您展示其静电和形状特性的不同之处,从而了解如何设计具有更好特性的更优化合物。
图 2:Activity Miner向您显示数据的多种视图,以帮助您找到SAR中的关键分子对。
在此版本中,Activity Miner完全集成在Flare GUI中,可以访问所有配体和蛋白质可视化选项。多个活性列上的多个Activity Miner项目可以保存在同一个Flare项目中,并在不同蛋白质结构环境中查看。
基于结构与基于配体的方法协同工作
在Flare中基于配体的方法通过展示配体具有活性所需的静电、疏水和形状特征,为您提供宝贵的SAR信息。
当蛋白质结构可用时,在蛋白静电的背景下查看结构-活性信息会提供额外的见解。蛋白相互作用势、静电势 (ESP) 以及静电互补性图将帮助您了解您的配体为何具有活性,让你了解蛋白质对活性的要求,而不仅仅是典型的配体-蛋白相互作用分析。
图 3 显示了两种苯并噁嗪酮RIP1激酶抑制剂。噁唑配体(左)的活性大约是三氮唑配体(右)的 10 倍,尽管这两种配体与蛋白质的活性位点具有相似的配体-蛋白质相互作用模式(图 3)。将蛋白质的ESP图(图 3 – 顶部)与两种配体的ESP图(图 3 – 中间)进行比较,我们可以看到三氮唑配体在蛋白质静电势为正的区域中显示出正静电区域。这种静电冲突被该配体朝向RIP1激酶活性位点的静电互补图所证实(图 3 – 底部,右),这可能是导致活性小幅下降的原因。
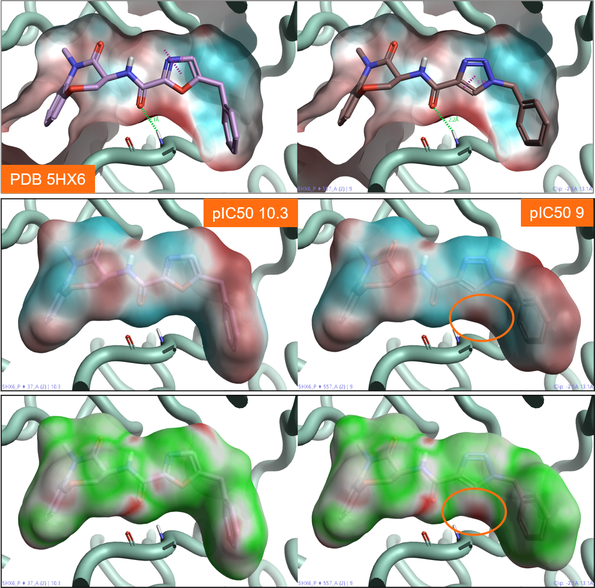
图 3:顶部:RIP1激酶活性位点(PDB:5HX6)的ESP图。 颜色编码:红色 = 正静电,蓝色 = 负静电。 中间:噁唑(右)和三氮唑(左)RIP1激酶配体的ESP图。颜色编码:红色 = 正静电,蓝色 = 负静电。底部:相同RIP1激酶配体的静电互补图。颜色编码:绿色=良好的互补性,红色=静电冲突。
图 4 显示了在RIP1激酶蛋白相互作用势(线框表面)背景下对 RIP1 激酶配体进行Activity Atlas分析的正负静电(固体表面)的活动悬崖总结。这两张地图非常吻合:这两种方法的协同应用让我们相信我们的 SAR模型是稳健的。

图4. 固体表面:RIP1激酶配体静电的活性悬崖总结。线框表面:RIP1 激酶(PDB:5HX6)的蛋白质相互作用势。颜色编码:红色 = 正静电,蓝色 = 负静电。
用GIST分析水分子
了解蛋白活性位点内水分子的行为是药物设计的一个非常重要的方面。
“可成药的”结合位点通常被水分子占据,由于焓和熵的原因,水分子在能量上更倾向于在大量水中。
在蛋白质-配体复合物中,了解桥连水分子的稳定性是决定药物设计策略的关键。如果水分子不是特别稳定,可以设计配体以取代它,并与蛋白质活性位点直接相互作用:这通常会导致配体生物活性的增加。
GIST是一种水分析方法,用于评估结合口袋的水合作用,并通过在分子动力学运行结束时对显式溶剂分布采样来计算相关的水热力学。 GIST分析的结果显示为活性位点水合作用的三维等值面映射“happy”(绿色,与负∆G相关)和“unhappy”(红色,与正 ∆ G相关)区域。unhappy区域映射的是蛋白质活性位点内更可能的可成药区域。happy区域映射的是蛋白结合位点内较低可能成药的区域,在那里水分子更稳定,因此更难置换。
图5-左侧显示了对乙酰辅酶A羧化酶2(PDB:3GID)的apo活性位点进行GIST分析的结果。结果正确地将底部的口袋映射为可成药的,正如预期的那样,该区域被PDB 3GID蛋白-配体复合物中的配体soraphen占据。Gist还将左侧的结合口袋映射为可成药的:这一点也得到了证实,在PDB 5KKN中,它被配体ND-646所占据。
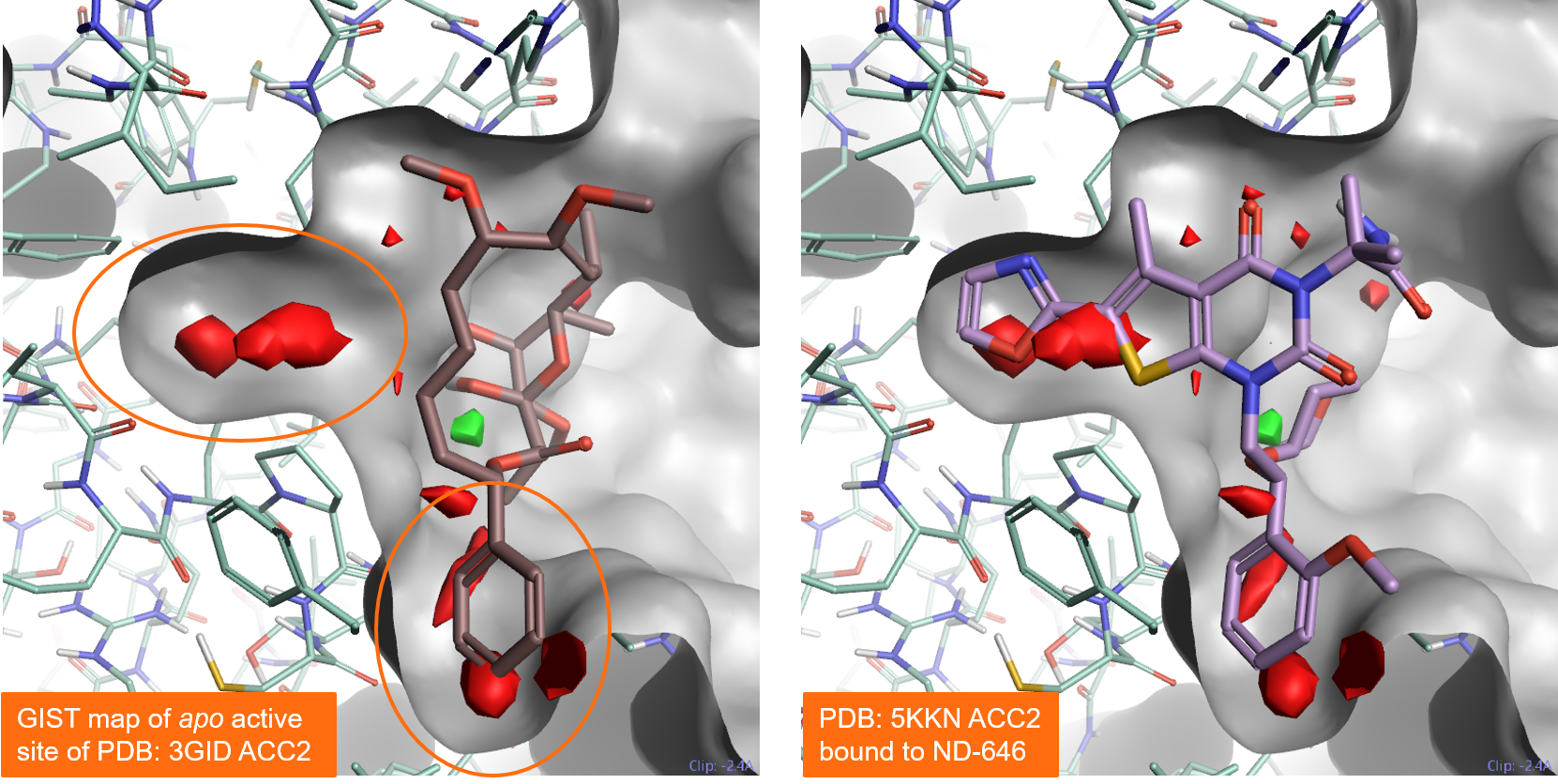
图 5. 对乙酰辅酶A羧化酶 2 (PDB 3GID)的apo活性位点进行的GIST分析结果。左:PDB 3GID配体Soraphen占据底部的结合口袋。右:来自 PDB 5KKN的ND-646配体占据左侧和底部的口袋。GIST预计这两个口袋都可以成药。颜色编码:红色 = unhappy(正 ∆G)水合作用的区域,绿色 = happy(负 ∆G)水合作用的区域。
增强的Flare FEP模拟以准确预测新化合物的活性
Flare V5中的Flare FEP(Free Energy Perturbation)模拟速度得到显著地提升,并且更加自动化。
更快的算法(比之前的版本提高3.5倍,图 6)得益于许多在幕后的系统改进:
- 改进的积分器的实现以及氢质量的再新分配,确保获得稳定的4fs时间步长
- 模拟使用更高效的截断八面体水盒以及更小的溶盒箱缓冲区,在不影响结果质量的情况下加快了计算速度
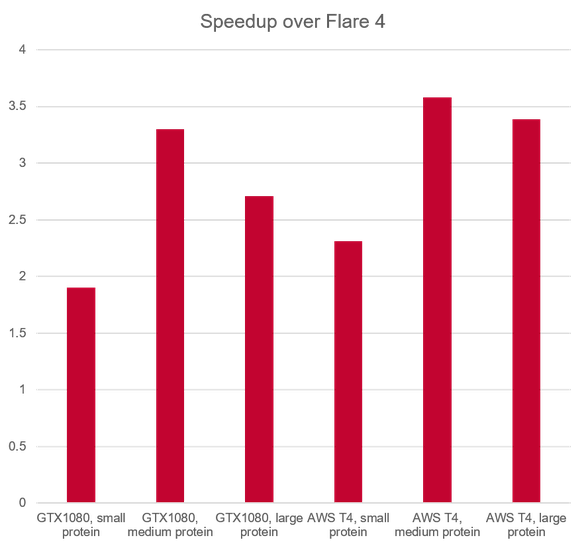
图 6:Flare V5 与 Flare V4中FEP计算的相对性能。
此版本中的Flare FEP也明显更加自动化。 增强功能包括:
- 自动生成中间结构以保证配体之间足够平滑的转换,优化 FEP 模拟的成功率
- 自动确定每个转换的最佳 lambda规划。设置lambda规划现在仅需要少量的用户交互,却获得额外30%的整体速度提升。
将生产模式计算中对微扰网络没有贡献的配体全部排除之后,可以获得更高的计算效率。
当使用星图时,这些变化还使Flare FEP能够在高达500个配体的更大规模上高效地工作。
更快速的分子动力学模拟实验、增强的接触可视化分析
由于为Flare FEP实施了相同的系统改进,Flare V5中的分子动力学模拟受益于明显更快的算法,并且还有额外的高级选项来微调动力学模拟实验的设置:
- 改进的积分器j加上氢质量重新分配以获得稳定的 4fs时间步长
- 更高效的截断八面体水盒
- 显式水模型的扩展选择
- 高级选项可设置温度、压力、长程静电处理、非键截断
Flare V5还具有增强的Contacts表单以监测有利的配体-蛋白质相互作用:
- 交互式:单击 Contacts 表中的配体/蛋白质原子标签就会在3D视窗中会突出显示对应的原子,双击将3D视图聚焦在该原子上
- 表单创建的速度显著地提升了
- 表单可以保存在Flare项目中,也可以导出
其他增强功能包括改进了对肽配体的支持以及对翻译后修饰氨基酸的支持。
为你的配体生成有意义的结合模式
我们显着地扩展了Flare的能力,通过FieldTemplater准确的场药效团为您的系列配体生成合理的结合假设(图 7)。
FieldTemplater使您能够在您靶标的蛋白质结构信息不可用时模拟配体的静电和形状等3D性质。在GPCR、离子通道和新靶点等情况下,FieldTemplater是重建配体生物活性构象的最佳方法,可获得简单易懂的解释性结果。
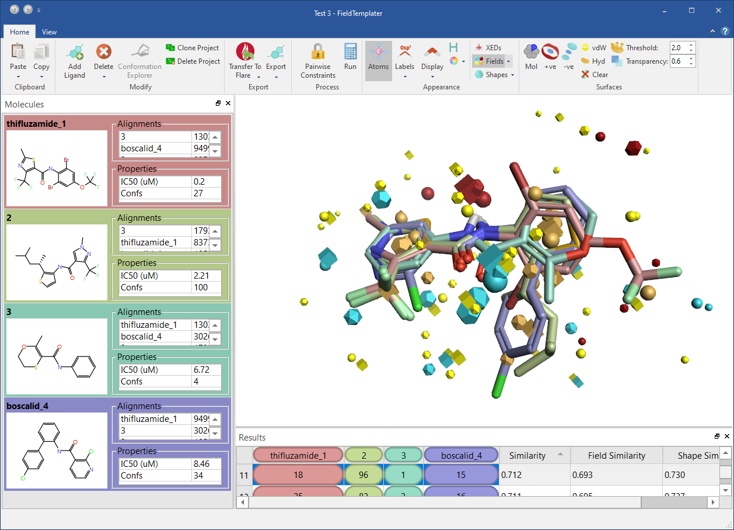
图 7:FieldTemplater使您能够在您靶标的蛋白质结构信息不可用时模拟配体的静电和形状等3D性质。
在Flare V5中使用LeadFinder算法进行对接和打分具有扩展的对接约束选择功能。你可以将这些约束应用于蛋白质原子/残基,以确保配体的结合模式与相关的蛋白质-配体相互作用匹配。完整的列表包括:
- 从蛋白原子定义的氢键
- 金属
- 阳离子-pi相互作用
- 芳香-芳香相互作用
- 盐桥相互作用
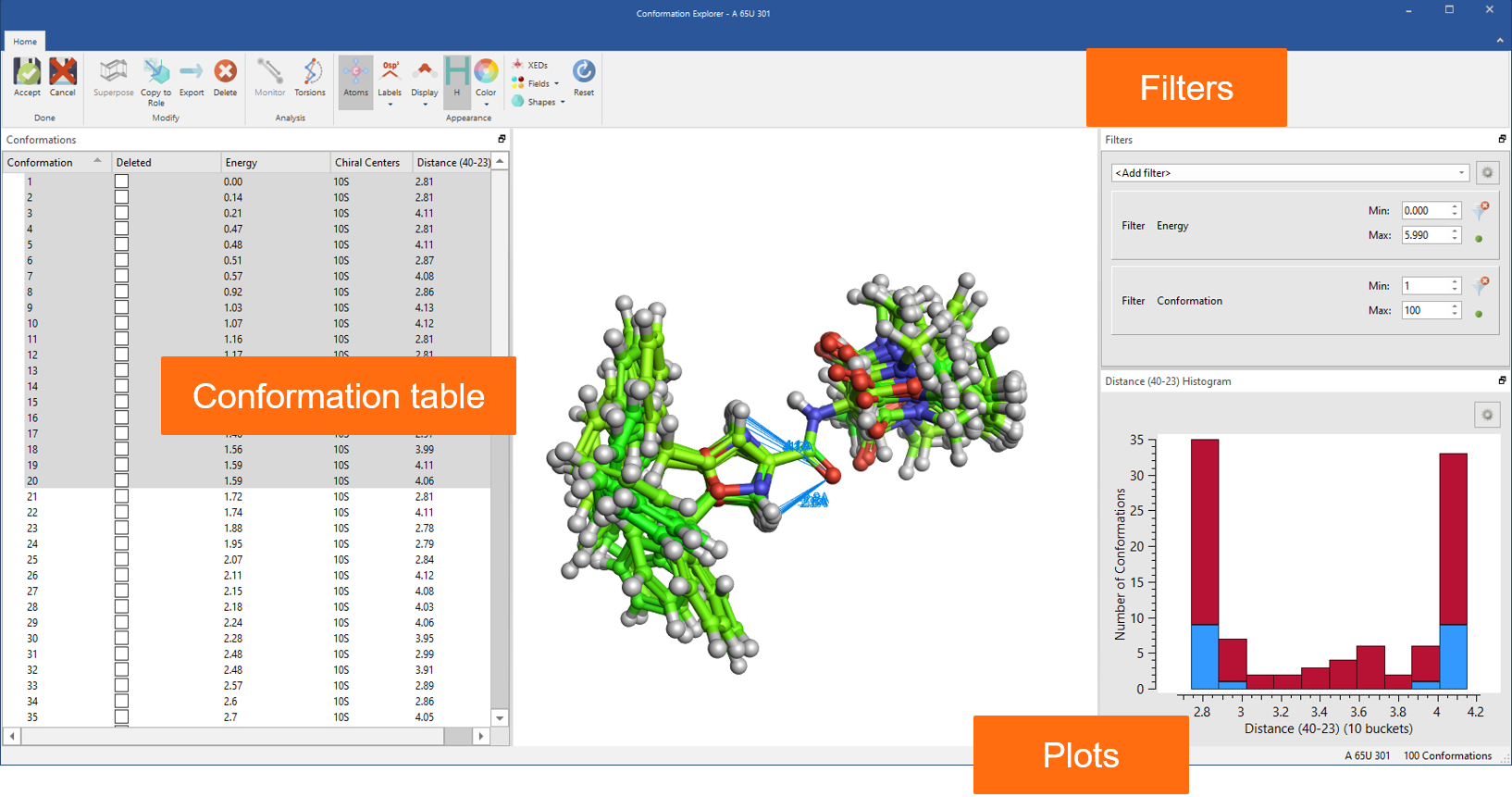
最后,来自Forge的所有基于配体的叠合高级选项现在都可以在Flare 中与Conformation Explorer一起使用,以检查和分析构象分布(图 8)。
图 8:使用Conformation Explorer来检查和分析Flare中生成的构象分布。
Flare Python API提供支持
从最早的版本开始,Flare就可以使用Python API,并且多年来不断改进,使您可以完全访问所有Cresset方法。 作为Forge与Flare整合的一部分,所有基于配体的方法都可以通过单个pyflare二进制文件和 Cresset开发的Python脚本从命令行访问,这些脚本直接与软件一起安装。
为了促进从Forge到Flare的过渡,python脚本使用与原始Forge可执行文件具有相同的命令行选项。
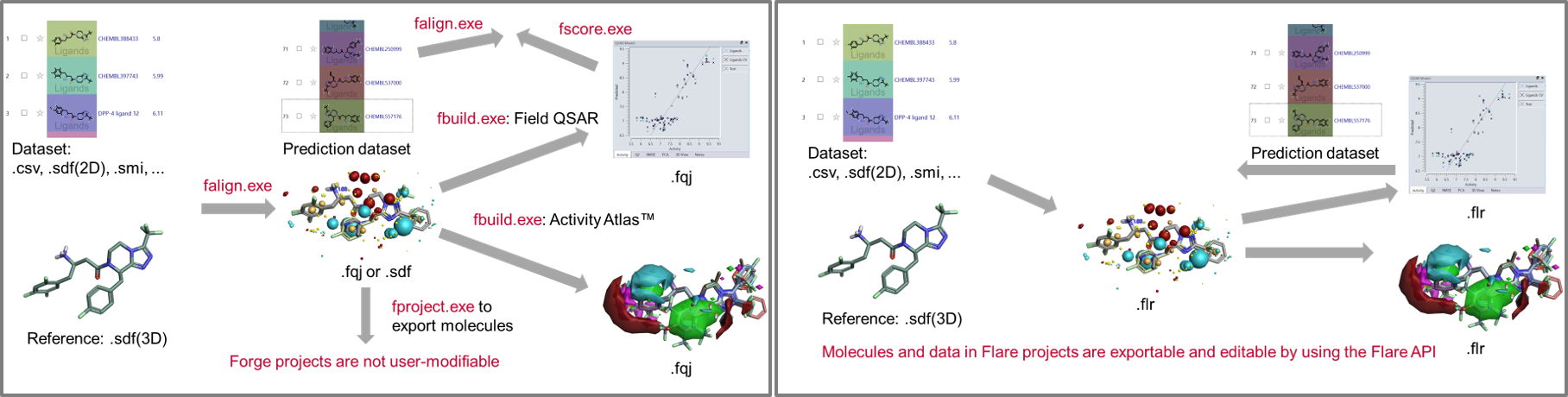
这显着简化了集成工作流的创建。 在图9(左)中,我们展示了一个Forge工作流来生成QSAR模型以预测新化合物的活性并了解它们的SAR。在图9(右)中,通过编写一个执行所有必要步骤的Python脚本来简化相同的工作流。
图 9:左:生成 QSAR 模型以预测新化合物的活性并了解其SAR的 Forge工作流程。 右:在 Flare 中可以编写一个执行所有必要步骤的Python脚本来简化相同的工作流。
灵活的授权与免费测试版
无论你是从事计算化学还是实验化学,无论是在工业企业还是在学术单位,我们提供的授权选项里总有一款适合你的。想亲自在项目中测试Flare V5,请联系我们。