摘要:本文以BCR-ABL1激酶为例,演示了如何用LigandScout预测潜在的结合位点,接着用GIST预测结合位点的成药性,最终识别出BCR-ABL1激酶的别构位点。在本算例中。LigandScout正确地识别出别构位点,GIST正确地确认该位点为成药性位点;进一步的分子对接表明,Flare分子对接成功地重现了结合模式。这说明将LigandScout的结合位点识别与GIST的成药性判断组合可以用于蛋白结合位点的预测,为基于结构的设计提供思路。
肖高铿/2021-07-23
1. 前言
了解蛋白活性位点内水分子的行为是药物设计的一个非常重要的方面。“可成药的”结合位点通常被水分子占据,由于焓和熵的原因,水分子在能量上更倾向于在大量水中。在蛋白质-配体复合物中,了解桥连水分子的稳定性是决定药物设计策略的关键。如果水分子不是特别稳定,可以设计配体以取代它,并与蛋白质活性位点直接相互作用:这通常会导致配体生物活性的增加。
在最近发布的Flare V5中引入了一个新的模块GIST[1]。GIST是一种水分析方法,用于评估结合口袋的水合作用,并通过在分子动力学运行结束时对显式溶剂分布采样来计算相关的水热力学。 GIST分析的结果显示为活性位点水合作用的三维等值面映射“happy”(绿色,与负∆G相关)和“unhappy”(红色,与正 ∆ G相关)区域。unhappy区域映射的是蛋白质活性位点内更可能的可成药区域。happy区域映射的是蛋白结合位点内较低可能成药的区域,在那里水分子更稳定,因此更难置换。
适逢AI领域在预测蛋白3D结构的突破,AlphaFold2[2,3]与RossetaFold[4]两大预测方法分别在Natue与Science上发表并开放其源代码。它们在预测蛋白三级结构上获得惊艳的效果。其中,AlphaFold2还对人类基因组蛋白进行了结构预测建立AlphaFold Protein Structure Database[5],通过欧洲分子生物学实验室共享预测结果。很多人认为,在AlphaFold预测的蛋白3D结构基础之上,挖掘潜在成药结合位点,可为药物开发提供新的思路与方向。
因此,本文的目的之一是,如何利用现有的结合位点探测与结合位点成药性评价方法,提供一个可靠的蛋白结合位点预测方案。为此,我以BCR-ABL1激酶为例,进行了一次回溯性分析以演示如何挖掘蛋白的潜在别构位点。选择BCR-ABL1激酶作为算例,主要考虑到几个实事:1)靶向BCR-ABL1正构位点的上市药物选择性不够好且出现耐药,寻找选择性更好、抗耐药的新型药物是大家关注的;2)靶向别构位点可克服选择性与耐药问题;3)BCR-ABL1激酶目前已确定了其别构位点,已有别构抑制剂Asciminib(ABL001)在临床研究中;4)别构抑制剂Asciminib比起竞争性抑制剂Imatinib晚了很17年才出现,说明该别构位点的发现与利用并不容易,但有共晶结构解释出来可供验证,适合于回溯性分析。
2020年2月,诺华宣布其Asciminib被FDA授予突破性疗法资格[6];2021年8月25日,诺华宣布该要获得FDA优先审评资格[8];2021年10月29日诺华宣布FDA批准其用于治疗慢性粒细胞白血病(CML),商品名为Scembix[9]。
2. 详细流程
首先,我们以STI-ABL1激酶复合物结构(PBD 1FPU)为起点,用LigandScout预测可能的结合位点;然后用Flare对预测的结合位点进行GIST分析,以判断该位点是否具备成药特征。然后,将Asciminib对接到1FPU预测到的别构结合位点里,并与X-Ray解释的Asciminib-ABL1复合物结构(PDB 5MO4)进行比较,考察对接能否重现Asciminib的结合模式。
2.1 用LigandScout预测结合位点
我在之前的教程里已经详细描述过如何用LigandScout预测结合位点[7],在本算例中我们用同样的方法,下载PDB 1FPU,删除B链,预测A链的结合位点,结果如图1所示。
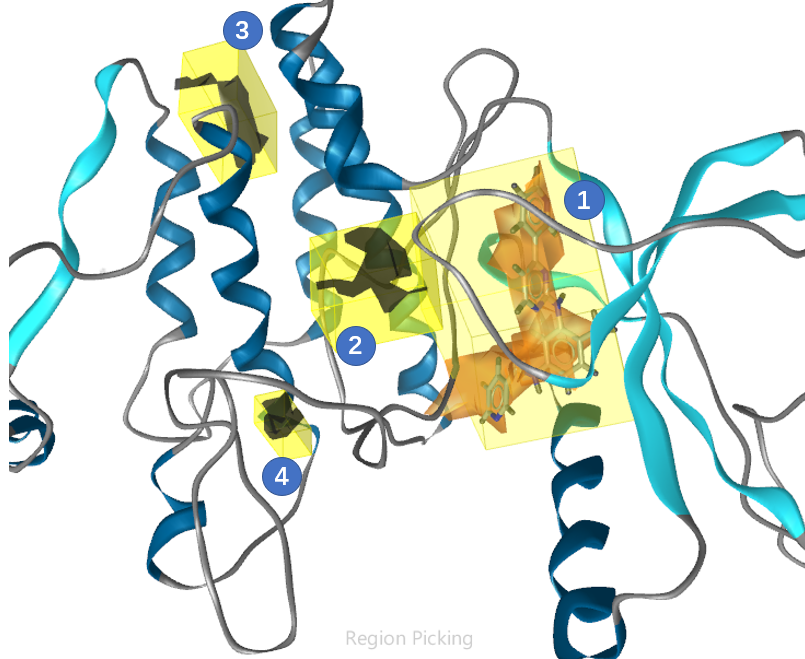
图1. PDB 1FPU的结合位点预测结果
如图1所示,Ligandscout的结合口袋预测方法探测到4个可能的结合位点。其中位点1是配体STI的结合位点;位点4看着体积很小,而位点2、3的体积相对较大。其中位点3就是已知别构抑制剂Asciminib的结合位点。接下来我开始用Flare GIST分析这些结合位点。
2.2 对结合位点进行GIST分析
本步骤的目的是对上一步获得的潜在位点进一步筛选,挑选出其中具有成药性的结合位点。我们可以从水分子的能量学角度来看待结合位点的成药性。我们假设配体与蛋白结合的过程是配体与水分子竞争结合到结合位点的过程。如果一个结合位点里的水大部分为unhappy水,则在理论上这些unhapp水可以被一个分子通过竞争性结合而替换掉,也就是具有成药性。相反,如果一个结合位点里的水都是happy水,那么就不存在一个分子可以竞争替换这些水的机会,也就是说该结合位点没有成药性。因此,我们可以借助GIST对结合位点计算水分子的能量特征来评估其的成药性。
Flare GIST是基于分子动力学模拟的方法。下载PDB 1FPU,删除B链,仅保留A链。在进行GIST分析时,全部采用默认值。分别对结合位点1与3进行GIST计算。
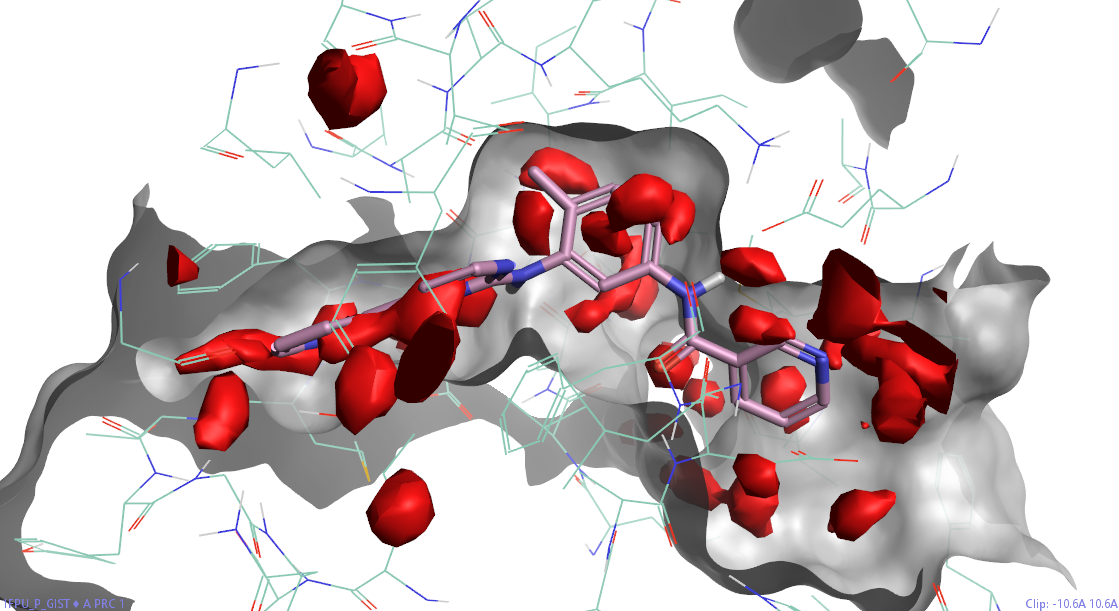
图2. PDB 1FPU结合位点1的GIST计算结果
图2显示了对BCR-ABL1激酶(PDB 1FPU)的结合位点1进行GIST分析的结果。GIST预测该结合位点具有成药性:只有红色的unhappy水出现,而没有绿色的happy水出现。结果正确地将该口袋映射为可成药的,正如预期的那样,该区域被抑制剂STI占据。
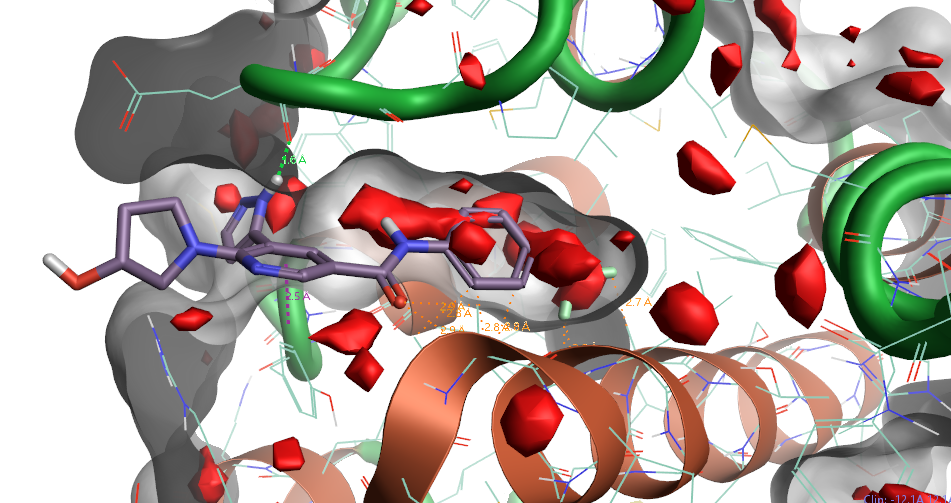
图3. PDB 1FPU结合位点3的GIST计算结果
图3显示了对BCR-ABL1激酶(PDB 1FPU)的结合位点3进行GIST分析的结果。GIST预测该结合位点具有成药性:只有红色的unhappy水出现,而没有绿色的happy水出现。结果正确地将该口袋映射为可成药的,在PDB 5MO4中,该区域被别构抑制剂Asciminib占据。
2.3 分子对接计算
在Flare中,用Flare Dock将Asciminib对接到1FPU的结合位点3,图4展示了对接计算的pose与PDB 5MO4共晶配体的比较,发现两者除了暴露于溶剂中的羟基取代的四氢吡咯环片段(红色圆圈高亮显示)具有较大差异外,其它部分叠合的相当好。这说明,在预测的结合位点基础上,使用Flare Docking可以正确预测出结合模式。这说明,在缺乏该别构位点与配体复合物的情况下,分子对接虚拟筛选也可以有效地发现潜在的配体,具有进一步进行虚拟筛选的实用价值。
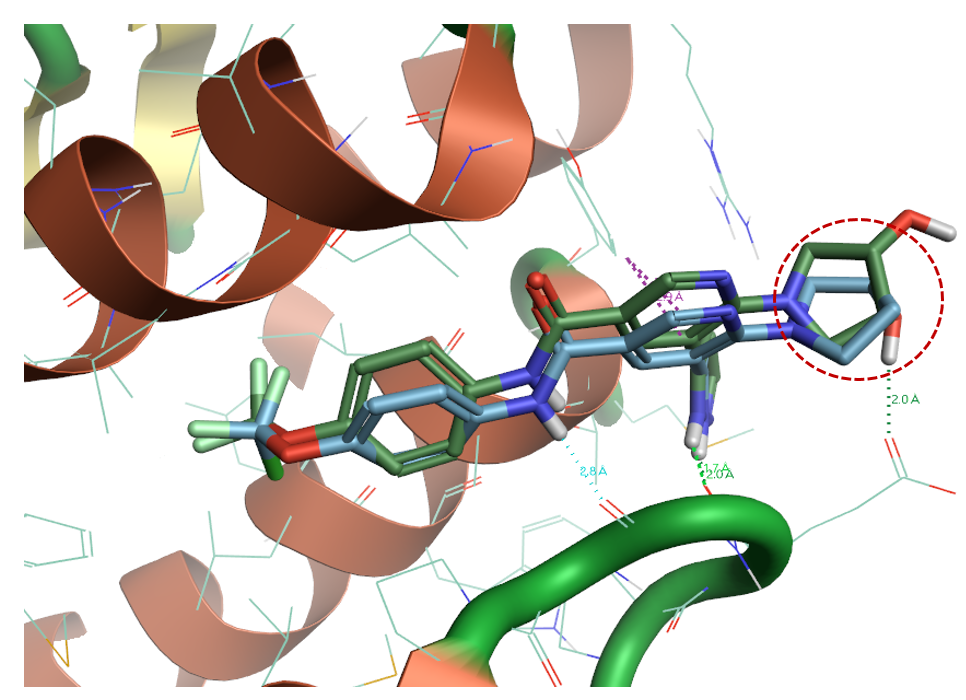
图4. 用Flare将Asciminib对接到PDB 1FPU结合位点3的结果。绿色:5MO4配体Asciminib;青灰色:Flare 计算的pose
3.拓展:GIST用于基于结构的设计
在1FPU算例中,我们用GIST分析一个潜在位点的成药性。我们也可以用GIST来分析已知结合位点亚结合位点的成药性,并用于指导基于结构的设计。
图5-左侧显示了对乙酰辅酶A羧化酶2(PDB:3GID)的apo活性位点进行GIST分析的结果。结果正确地将底部的口袋映射为可成药的,正如预期的那样,该区域被PDB 3GID蛋白-配体复合物中的配体soraphen占据。Gist还将左侧的结合口袋映射为可成药的:这一点也得到了证实,在PDB 5KKN中,它被配体ND-646所占据。
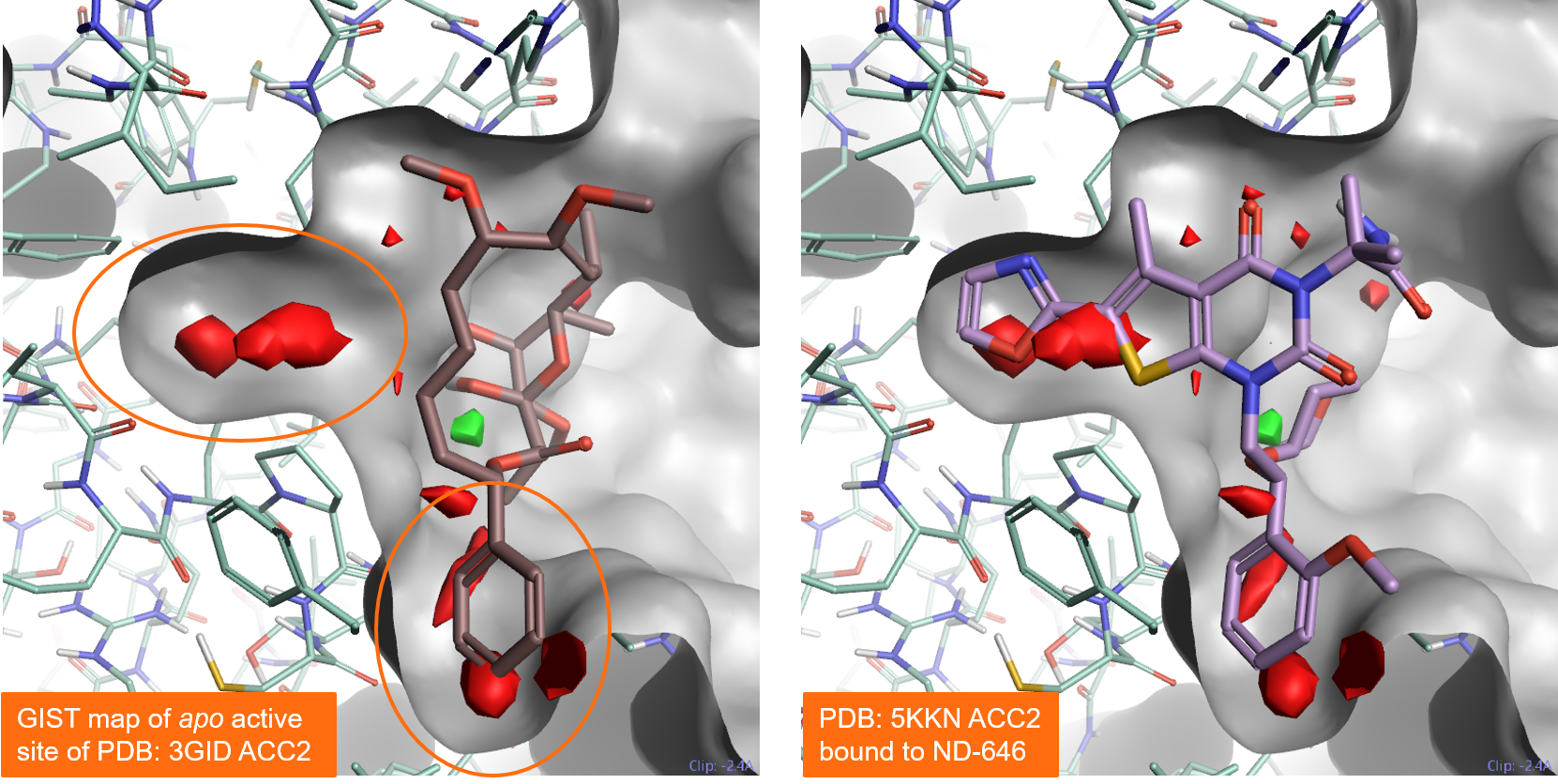
图 5. 对乙酰辅酶A羧化酶 2 (PDB 3GID)的apo活性位点进行的GIST分析结果。左:PDB 3GID配体Soraphen占据底部的结合口袋。右:来自 PDB 5KKN的ND-646配体占据左侧和底部的口袋。GIST预测这两个口袋都可以成药。颜色编码:红色 = unhappy(正 ∆G)水合作用的区域,绿色 = happy(负 ∆G)水合作用的区域。
在基于结构的设计中,该计算可以帮助我们决策是否往一个子口袋“生长”片段而优化现有化合物的活性,因此GIST与SPAKR的分子生长策略具有很好的协同使用前景。
4.文献
- Nguyen, C.; Gilson, M. K.; Young, T. Structure and Thermodynamics of Molecular Hydration via Grid Inhomogeneous Solvation Theory. 2011. arXiv:1108.4876v1. https://arxiv.org/abs/1108.4876
- Jumper, J.; Evans, R.; Pritzel, A.; Green, T.; Figurnov, M.; Ronneberger, O.; Tunyasuvunakool, K.; Bates, R.; Žídek, A.; Potapenko, A.; et al. Highly Accurate Protein Structure Prediction with AlphaFold. Nature 2021, 1–11. https://doi.org/10.1038/s41586-021-03819-2.
- Callaway, E. DeepMind’s AI for Protein Structure Is Coming to the Masses. Nature 2021. https://doi.org/10.1038/d41586-021-01968-y.
- Baek, M.; DiMaio, F.; Anishchenko, I.; Dauparas, J.; Ovchinnikov, S.; Lee, G. R.; Wang, J.; Cong, Q.; Kinch, L. N.; Schaeffer, R. D.; et al. Accurate Prediction of Protein Structures and Interactions Using a Three-Track Neural Network. Science (80-. ). 2021, 8754 (July), eabj8754. https://doi.org/10.1126/science.abj8754.
- AlphaFold Protein Structure Database. https://alphafold.ebi.ac.uk
- Novartis receives FDA Breakthrough Therapy designations for investigational STAMP inhibitor asciminib (ABL001) in chronic myeloid leukemia. Retrieved from https://www.novartis.com/news/media-releases/novartis-receives-fda-breakthrough-therapy-designations-investigational-stamp-inhibitor-asciminib-abl001-chronic-myeloid-leukemia
- 肖高铿.LigandScout教程–VINA分子对接.墨灵格的博客.http://blog.molcalx.com.cn/2016/04/04/ligandscout-vina-tutorial.html
- FDA accelerates review of Novartis STAMP inhibitor asciminib (ABL001) for patients with chronic myeloid leukemia (CML) (2021-08-25).Retrieved from https://www.novartis.com/news/fda-accelerates-review-novartis-stamp-inhibitor-asciminib-abl001-patients-chronic-myeloid-leukemia-cml
- FDA approves Novartis Scemblix® (asciminib), with novel mechanism of action for the treatment of chronic myeloid leukemia. (2021-10-29). Retrieved from https://www.novartis.com/news/media-releases/fda-approves-novartis-scemblix-asciminib-novel-mechanism-action-treatment-chronic-myeloid-leukemia



