摘要:本文演示了如何利用Flare水分析工具3D-RISM用来分析大环抑制剂BRD-810与靶标的共晶结构、理解大环的结合模式,并利用这些信息来帮助设计新的化合物,并讨论如何使用水分析来辅助设置后续的其它高级计算。
作者:Jenny Brookes
编译:肖高铿
在本篇博文中,我们将探讨Flare水分析工具3D-RISM1在研究大环抑制剂与人类髓细胞白血病蛋白1(Myeloid cell leukemia 1, MCL1)活性位点结合机制中的应用价值。MCL1作为一种关键的抗癌靶点,近期Rauh等人2的研究介绍了一种高效且具有选择性的抑制剂BRD-810,该抑制剂能够促使肿瘤细胞凋亡。这项研究公布了BRD-810与MCL1靶点形成的共晶结构。通过深入分析这一共晶结构,运用3D-RISM技术来理解大环抑制剂的结合模式,并利用这些信息指导新化合物的设计。此外,还讨论如何利用水分析结果来辅助设置其他计算流程,从而为药物设计提供有力的支持。
结合位点探索
首先,我下载了包含电子密度图的共晶结构PDB 8T6F2。我从检查拟合后的电子密度图(图1,紫色网格,2fo-fc图)开始,并显示差示图(fo-fc图),以检查模型中是否存在过度拟合或电子密度未充分支持的原子。我还检查了观察到的替代氨基酸侧链位置,考虑到观察到的占据率并查看晶体接触,以找出任何由于晶体堆积(crystal packing)而导致的异常信号(artifacts)。
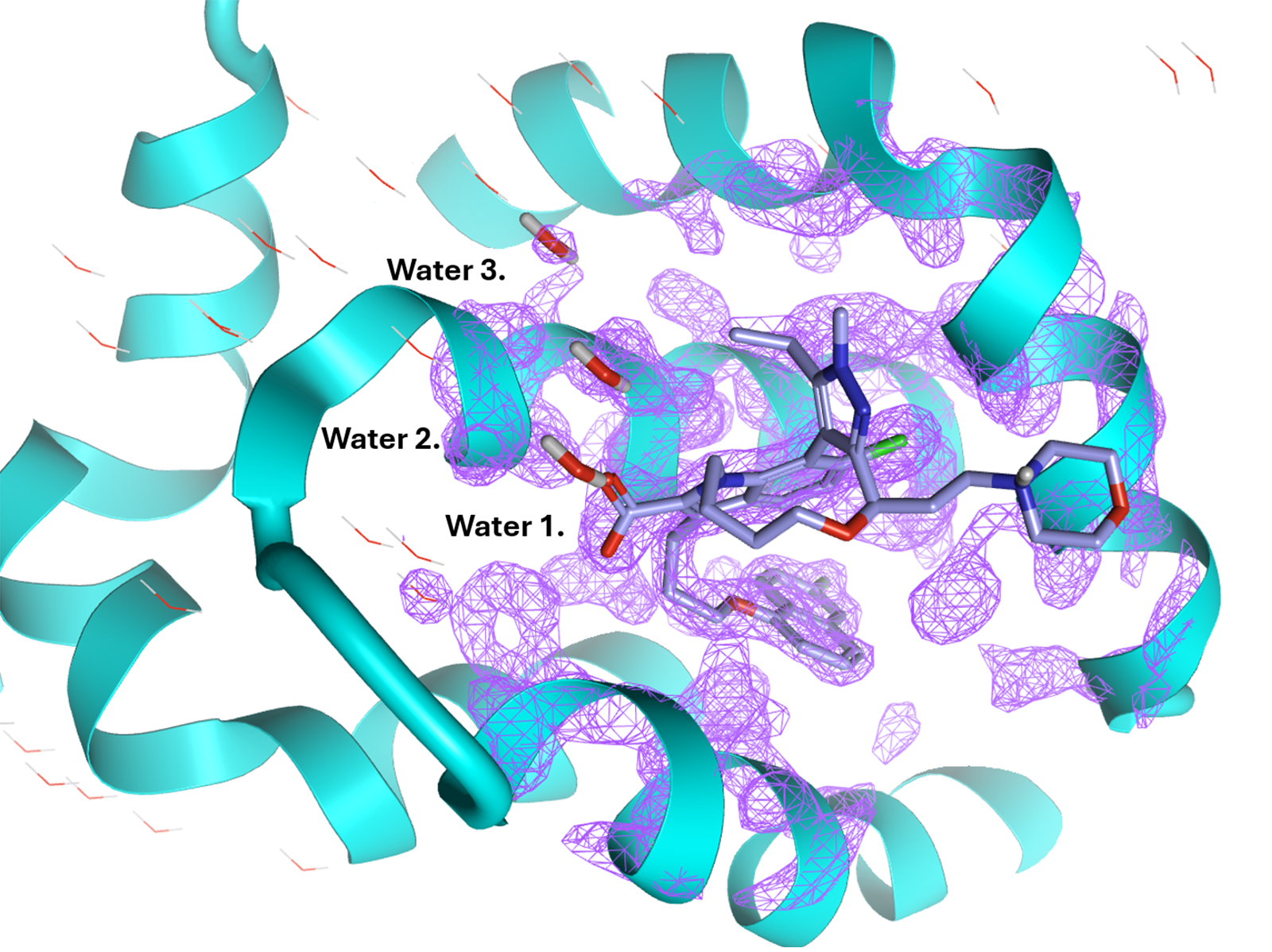
图1. Flare可视化PDB 8T6F2,显示了大环抑制剂BRD-810与MCL1的BH3沟槽结合的电子密度图(2fo-fc图)。我专注于检查配体和活性位点残基以及水的位置以对拟合电子密度进行信心评估。结合位点部分暴露于水中,特别是三个水分子(标记为1-3)靠近大环。
如图1所示,注意到三个靠近结合位点的水,它们都具有1.5σ的电子密度,并且可能发生潜在的重要相互作用。在检查了电子密度后,我用集成在Flare中的两个工具“Protein Preparation”和“Protein structure Checker”——这两个工具旨在优化并验证蛋白质结构——对蛋白结构进行准备与验证。前者用于保持所有水分子、添加质子并进行能量最小化,后者用来检查碰撞、无效价键以及末端是否被封闭。对配体的质子化状态和互变异构体状态进行了检查,然后进行了一次10ns分子动力学模拟(使用了GCNCMC,设置150mM NaCl浓度)。该分子动力学模拟旨在释放蛋白质在结晶过程中可能引入的结构张力。
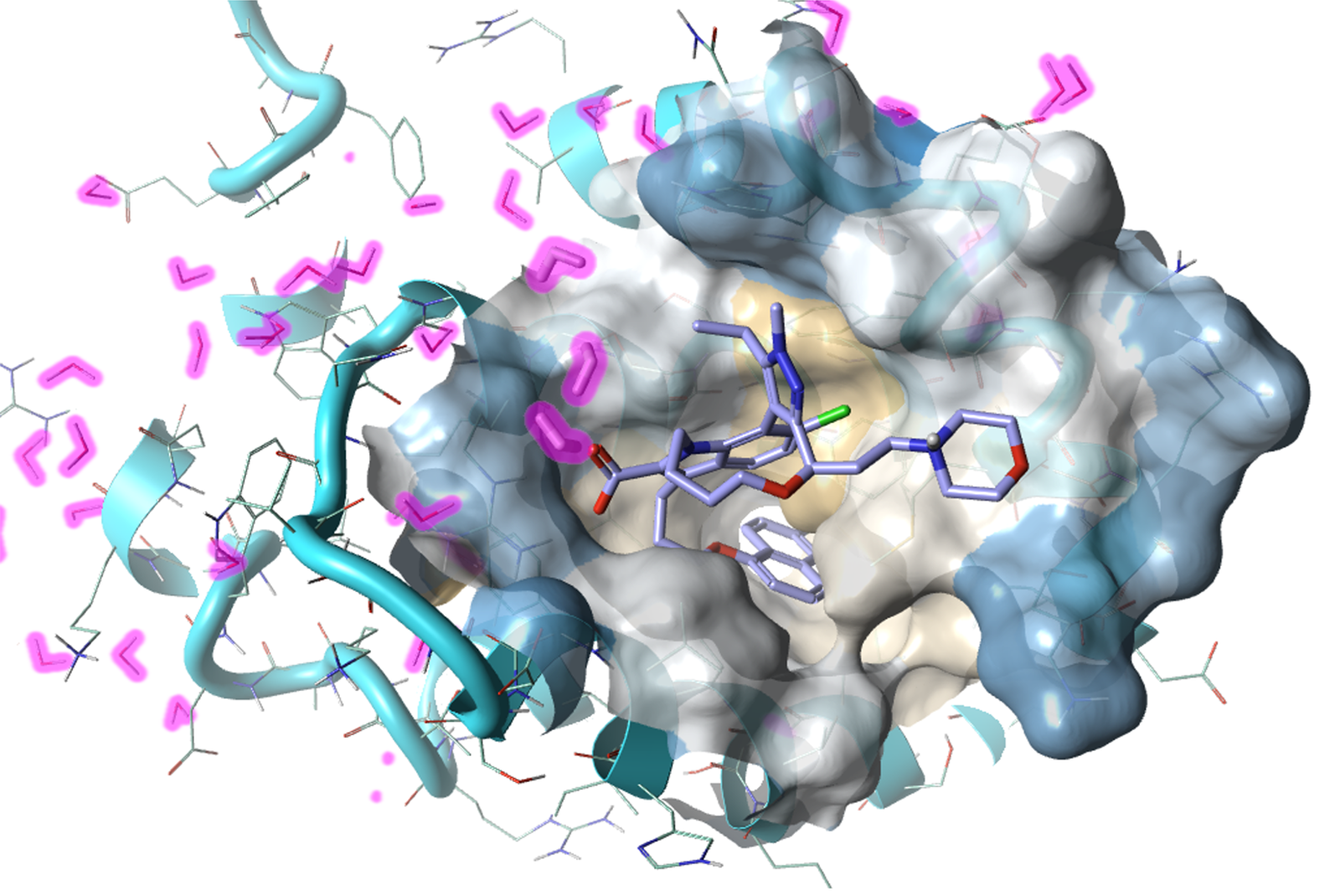
图2. 从准备好的PDB 8T6F2进行的MD中截取的一张快照,显示了结合位点的疏水表面(黄色表示疏水性,蓝色表示亲水性)和原始的晶体水(用粉红色高亮显示),叠加在分子动力学模拟的快照帧上。三个感兴趣的水分子渲染为“棍状”(靠近结合位点),其它X-衍射的水分子渲染为“线状”。
图2显示了蛋白质的疏水表面,以帮助理解结合环境并与原始的结晶水(用粉红色高亮显示)进行了比较。在本文中,我们研究了与亲水表面(蓝色表面)上大环化合物靠近的三个水分子的热力学性质。活性位点中没有其它来自晶体结构的水分子,该位点大部分是疏水性的(黄色表面)并且被抑制剂占据。结合槽有一个深的疏水口袋,大环化合物的双环延伸进入其中,而大环化合物的羧基和吗啉取代基则分别突入蛋白表面的水可及区域(图中蓝色表面,左侧和右侧),这些区域暴露于溶剂,可与水分子接触。
结合口袋的水合位点分析(作为后续模拟的准备工作)
为了获得更深入的见解并更准确地模拟结合口袋的水合位点,使用了3D-RISM(Three-Dimensional Reference Interaction Site Model)方法对结合口袋进行分析。3D-RISM是一种计算方法,用于预测生物分子结构周围溶剂分子的分布和占据,通过基于分子相互作用计算粒子密度网格来实现。我运行了一个完整的holo 3D-RISM计算,并利用Cresset XED力场3进行出色的静电模拟(图3),并与MD快照和原始结晶水位置进行了比较。Holo 3D-RISM是在蛋白质和配体的背景下运行的(见图3中的3D-RISM面板),并且没有利用已知结晶水的信息。3D-RISM模拟的输出包含了粒子密度(水的O和H密度)网格,从而可以预测水分子的占据区域(以表面形式呈现)和具体位点(以球形“3DR”表示)。在本文中,我们关注三个感兴趣的水,但请注意,3D-RISM可以提供氧位点(3DR),这些位点很容易转化为水以对整个靶标进行精确的溶剂化,并且因此准备好了进一步建模的输入。如果你计划将模型用于药物发现工作后续阶段,比如用MM/GBSA和FEP进行定量测结合亲和力模拟,建议运行完整的3D-RISM以准确地对你的体系进行溶剂化处理,然后运行MD以检查体系的稳定性和结合稳定性。BRD-810及其几个取代的大环吲哚衍生物4的实验结合亲和力已知,这使其适合于进行基准FEP计算,随后进行生产FEP,以测试新的设计想法。
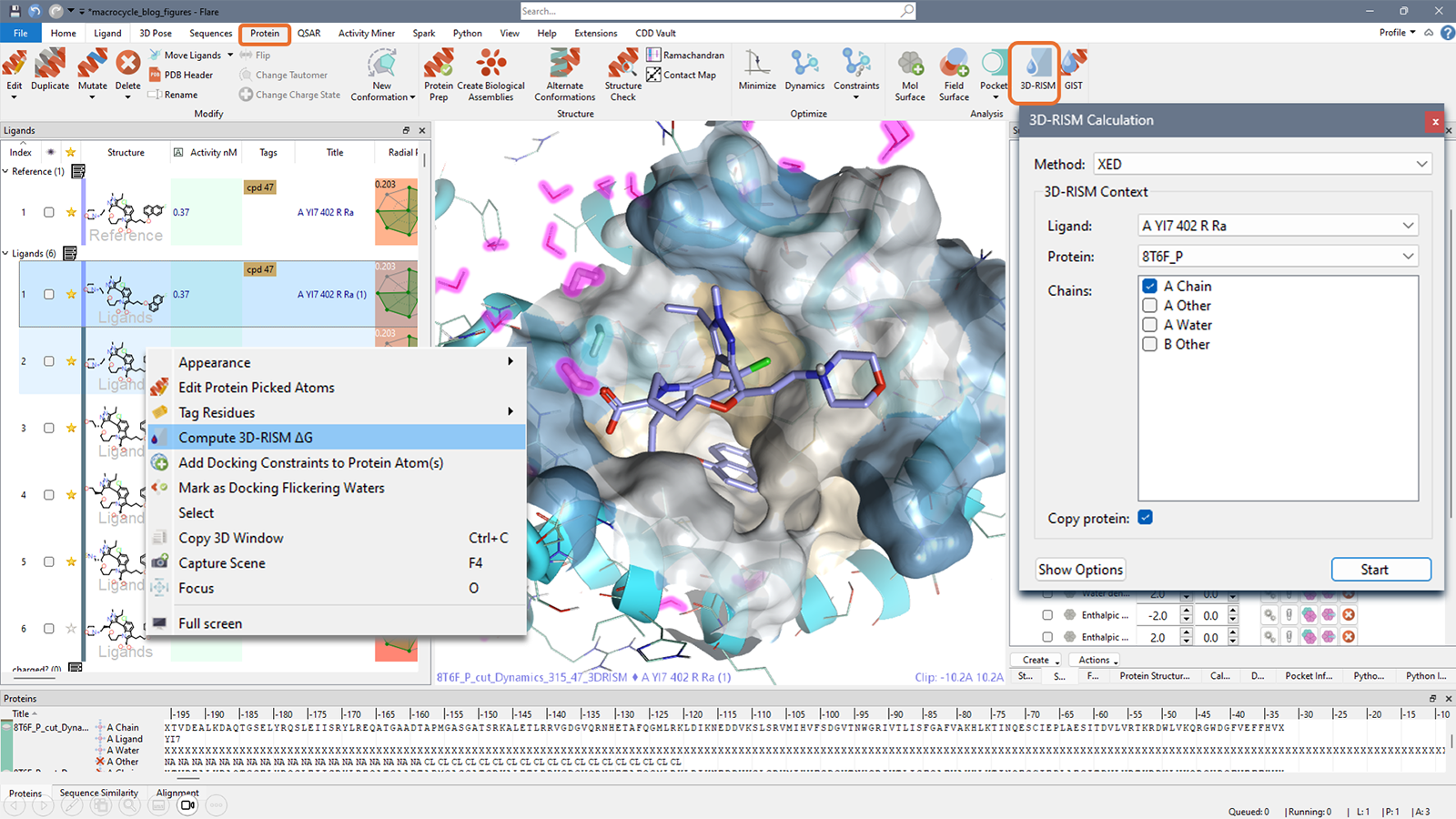
图3. 在Flare中进行holo 3D-RISM水分析计算(橙色方框):选择配体和蛋白质链以包括蛋白质-配体复合物作为背景,并确认使用XED力场3。计算结果会出现在“Surface”视窗,然后可以右键单击选定的水分子(这里为蓝色的结晶水2)以计算预期的3D-RISM ΔG贡献值,单位为kcal/mol。
图3显示了使用3D-RISM计算的每个原始结合水位点的水∆G贡献:以kcal/mol为单位,并分别用绿色表示“Happy”水,用红色表示“Unhappy”的水(或有利/不利结合能贡献)。结果如图4所示,左侧是原始结合水位置(由于无法分辨而未显示质子);右侧是在MD快照中看到的水相互作用。检查水1-3的3D-RISM值,晶体结构中水1–3的ΔG值分别为 -0.8、-1.6 和 -1.5 kcal/mol,而MD快照中对应值为 -0.4、-2.0 和 0.3 kcal/mol。两个水合位点(左图为晶体模型,右图为MD快照)的Water 1、2 和 3 的 RMSD 分别为 1.1 Å、0.7 Å 和 0.2 Å,表明晶体水和MD模型之间的氧位置具有良好一致性。这说明,在没有这三个结晶水“在先知识”的情况下,3D-RISM可以准确地预测了它们的位置。
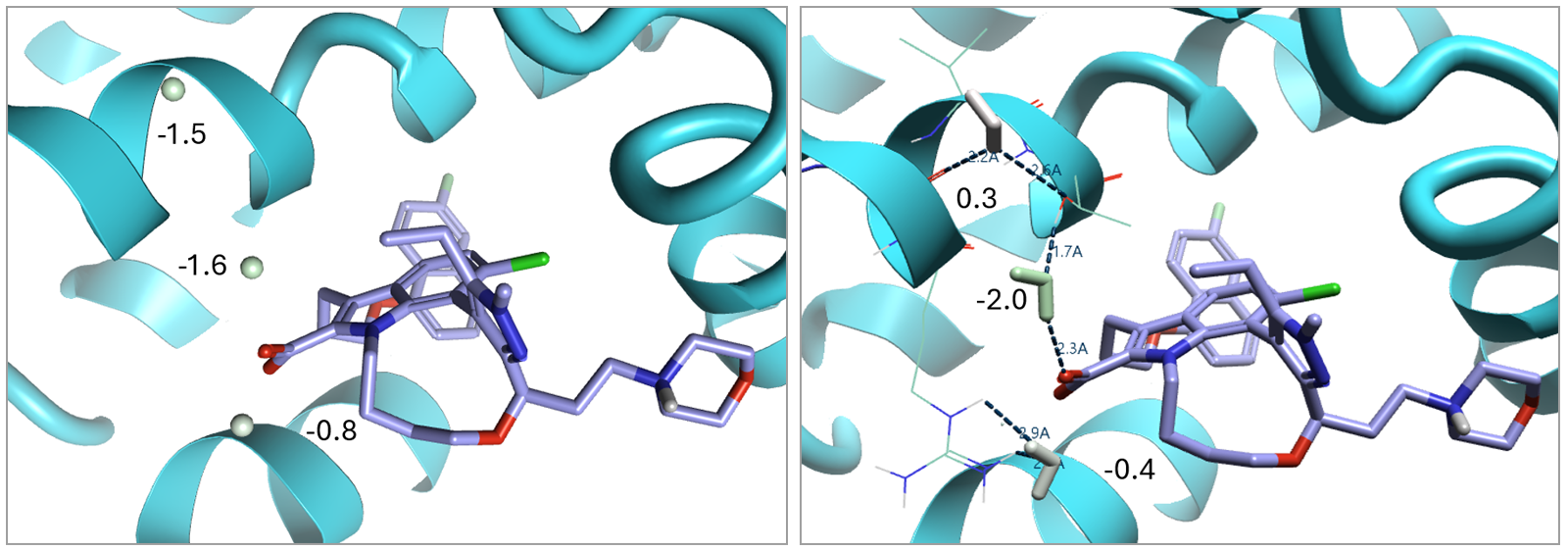
图4. 左图为原始结合水位点(因为无法分辨而未显示质子),右图为精选的MD快照水位点,均按计算的3D-RISM ΔG着色,单位为 kcal/mol。三个感兴趣的水分子的ΔG贡献值分别为左:-0.8、-1.6、-1.5 和 右:-0.4、-2.0 和 0.3 kcal/mol。水分子用绿色表示“Happy”或红色表示“Unhappy”。晶体模型与MD模型快照的水位置之间的RMSD (左侧与右侧氧原子的位置)分别为1.1Å、0.7Å 和 0.2Å 分别对应于Water 1-3。
从MD分析(图4-右)中可以看出频繁的水相互作用发生:
- 在溶剂可及的Water 1和ARG263以及大环的羧基之间。
- 在96%的帧中,桥接水Water 2向大环的羧基提供氢键,并接受来自THR266的氢键,表现出“DA”模式。
- Water 3向THR266和GLY262的骨架氧提供氢键,表现出“DD”模式并稳定蛋白质。
晶体结构与MD轨迹之间的一致性增强了我们对模型可靠性的信心。Water 1 作为“happy”水分子,介导ARG263与大环羧酸基团之间的水桥相互作用,提供“水合“作用,与周围其他水分子相互作用的同时与溶剂水进行交换。Water 2是最“Happy”的水,这符合预期:Water 2是桥接水,有助于大环与靶标之间的相互作用。这在MD轨迹上表现为高频发生的或强烈相互作用(96%),并与晶体结构中原始结合水的∆G预测值(kcal/mol)相吻合。我们确定在进一步模拟中应该将这些水包括在内,特别是那些在结合位点进行定量能量计算的模拟。事实上,我们可以可靠地使用3D-RISM来给结合口袋(图5)和整个蛋白计算水合位点,并识别重要的水分子。3D-RISM是一种准确的方法,可以验证晶体中存在的水分子4,并且可以适当给整个体系加溶剂水。最后,Water 3 虽可通过“DD”模式稳定蛋白骨架,但因其位于亲水表面,与体相水频繁交换,更接近溶剂水行为。
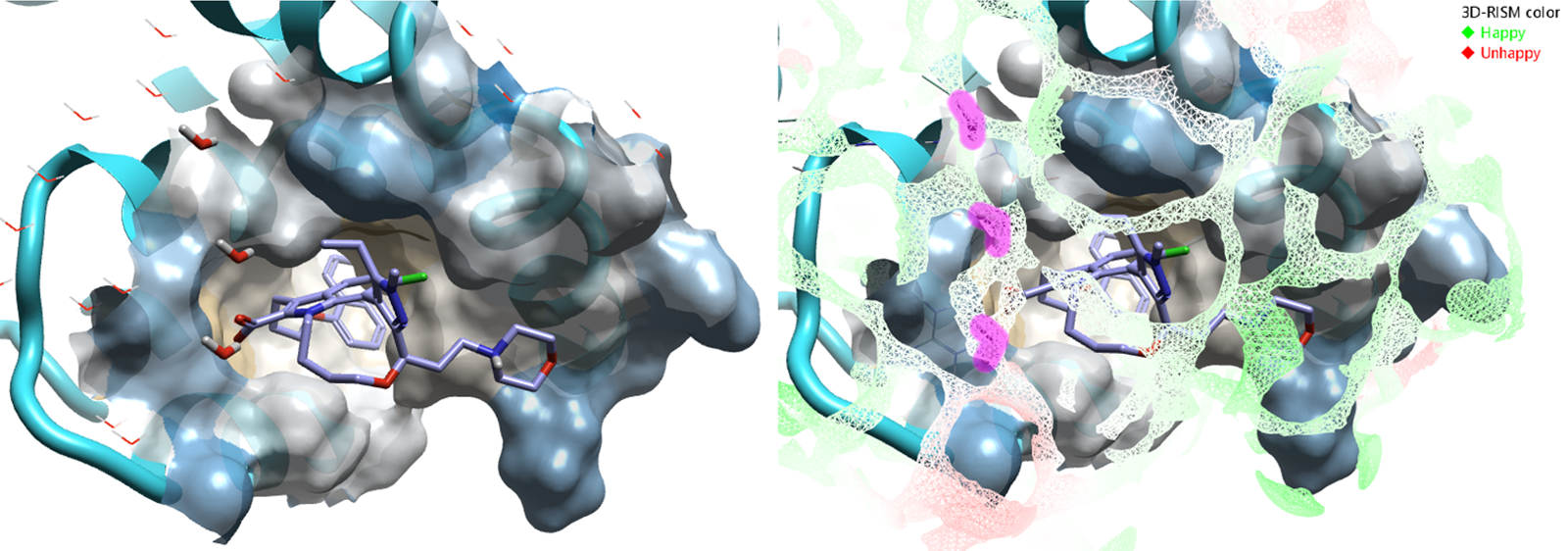
图5. 左:原始晶体水位置(红色和白色,“棍棒状”);右:在准备、动力学模拟和3D-RISM计算的模型(没有“实验性”水的知识)。所有的水(为清晰起见,仅显示其中三个正在研究中的水,即粉红色高亮显示的那些)都来自3D-RISM预测的“3DR”氧密度,并进行编辑和预处理以展示真实的水相互作用。还显示了从3D-RISM计算中获得的氧密度表面,并用绿色网格表示“Happy”,用红色网格表示“Unhappy”。这三个水如预期地位于网格表面上,我们可以从网格表面对整个体系预测其溶剂化自由能。
图5显示了从3D-RISM获得的氧密度表面(网格),绿色表示“Happy”,红色表示“Unhappy”。3D-RISM不仅准确识别出与晶体结构⁵中观察到的水分子位置一致的位点,还能分析整个体系,并且可以给出在晶体结构中未观察到的位置以便用于后续模拟。这对于高级的基于结构的设计(SBD)模拟至关重要,在没有或实验数据不完整的地方(例如同源建模模型、AlphaFold模型和冷冻电镜模型)尤其有价值。水分子在蛋白结合位点中起着至关重要的作用,而3D-RISM可以在药物发现过程中快速(在单个CPU上约20分钟)、准确地预测它们。
驱动药物设计
Flare可以使用分子表面和3D-RISM的组合来促进药物设计。通过检查疏水性表面并选择“3DR”:3D-RISM氧位点显示为球状,用绿色表示“Happy”(\(ΔG < -1.5 kcal/mol\)),用红色表示“Unhappy”(\(ΔG > 1.5 kcal/mol\)),我们可以研究结合区域,并寻找可能替换“Happy”水或置换“Unhappy”水的地方(图6)以添加新的基团。这样我们就可以产生新的药物设计想法,在这个案例中,在BRD-810基础上进行简单的修饰可增加结合强度,然后进行计算模拟以验证其有效性。
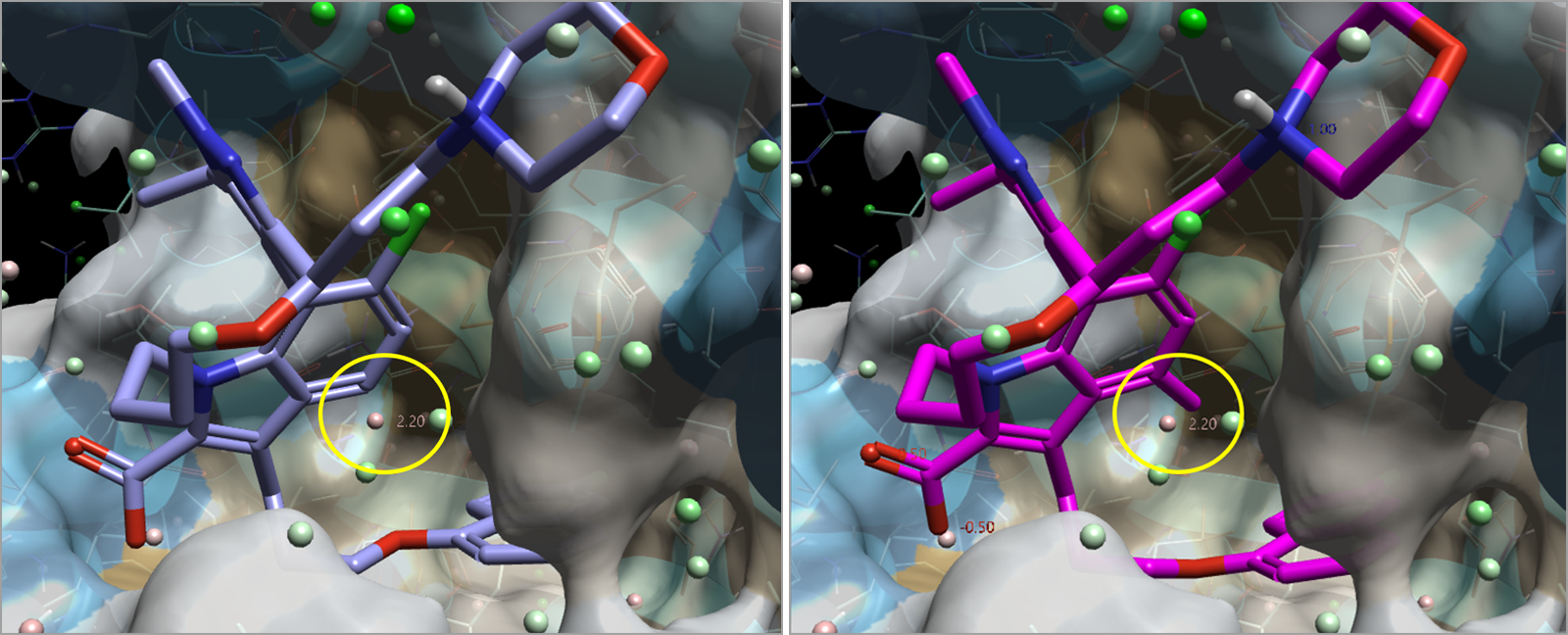
图6. 使用疏水表面并选择“3DR”:3D-RISM氧位点(球状,绿色表示“Happy”,红色表示“Unhappy”),其值分别小于-1.5和大于1.5 kcal/mol。例如,一个显示∆G为2.20 kcal/mol的3DR(黄色高亮显示)是“Unhappy”的,或具有不利的结合自由能贡献,并位于疏水区域中,添加甲基(右:粉色配体)可以“置换”出这个“Unhappy”的水分子,从而增加亲和力。
如图6所示,基于Flare的3D-RISM分析,我们可能会建议研究BRD-810的衍生物,在疏水区域中简单但明智地放置一个疏水基团以去除“Unhappy”水。在本例中,置换此类水可能增加约2 kcal/mol结合自由能。因此,通过使用3D-RISM水分析可以帮助产生设计想法,如添加甲基(图6中的粉色配体),或更大的卤素取代基,比如用Cl来置换“Unhappy”的水以提高活性。
同样,如果有机会替换一个“Happy”水分子,例如将极性官能团放置在更亲水的区域以取代与蛋白相互作用的水并释放到溶剂中,则这也可以增加结合亲和力。
请注意,这不是对3D-RISM的能力和用途的最终证明。这里的重点是分析结合配体的结构,确保体系加了正确的水,并为后续的高级计算提供保证,同时提供信息以设计新化合物来置换或替代水分子。
还可以对apo-结构(无配体结合的结构)进行3D-RISM计算。这些计算可以帮助您从水置换的观点了解为什么配体会与之结合,以及在结合位点未知时评估假定的结合位点(检查口袋的成药性)。这对于生长片段或创建大环化合物尤其有用,例如从非环状化合物出发设计大环化合物。通过观察氧表面绘制出的水占据情况可以指导化合物设计的形状和结构,以指示您在哪生长以及生长什么。如果您的新化合物设计难以实现或成本高昂,那么使用3D-RISM来查看水占据情况将为您提供信心,以便做出正确的设计。配体结合主要取决于活性位点的去溶剂化所带来的成本或收益,3D-RISM让您能快速、经济地探索并利用药物设计中的这些去溶剂化效应。
作者简介

Jenny Brookes
Jennifer Brookes 博士拥有逾15年的科研经验,研究背景涵盖理论与计算化学及生物物理学。她的科研贡献涉及多个领域:从探索嗅觉过程中可能存在的量子效应,到量化光合作用中的高效能量传递机制,再到利用抗体(纳米抗体)-抗原对中的蛋白质-蛋白质相互作用。Jennifer 的研究足迹遍及伦敦国王学院、伦敦大学学院(UCL)、麻省理工学院(MIT)和哈佛大学,并于近期重返伦敦大学学院,任职于伦敦纳米技术中心(London Centre for Nanotechnology),担任英国工程与物理科学研究理事会(EPSRC)投入1100万英镑设立的“传染病早期预警传感系统”跨学科研究中心(IRC)的首席计算与理论生物物理学家。
参考文献
- T. Luchko, et al. Three-dimensional molecular theory of solvation coupled with molecular dynamics in Amber. J. Chem. Theory Comput. 2010, 6, 3, 607–624. https://doi.org/10.1021/ct900460m
- U. Rauh, et al. BRD-810 is a highly selective MCL1 inhibitor with optimized in vivo clearance and robust efficacy in solid and hematological tumor models. Nat Cancer. 2024. https://doi.org/10.1038/s43018-024-00814-0
- J. G. Vinter. Extended electron distributions applied to the molecular mechanics of some intermolecular interactions. J Computer-Aided Mol Des 8, 653–668 1994. https://doi.org/10.1007/BF00124013
- US11891404B2 – Substituted macrocyclic indole derivatives – Google Patents
- L. Fusani, et al. Optimal water networks in protein cavities with Gasol and 3D-RISM. Bioinformatics. 2018 Jun 1;34(11):1947-1948. https://doi.org/10.1093/bioinformatics/bty024
联系我们
想在自己的项目中使用Flare,请联系我们获取免费的试用版。你还可以采购软件或委托我们进行项目合作:info@molcalx.com。
原创文章,作者:小墨,如若转载,请注明出处:《使用Flare™水分析3D-RISM在大环抑制剂BRD-810结合位点进行探索、准备与药物设计》http://blog.molcalx.com.cn/2024/12/02/macrocycle-inhibitor-water-analysis-3d-rism.html



