摘要:目前已经有非常多的ADMET性质预测工具,包括商业软件、开源软件。本文介绍了一个开源、免费的工具ADMET-AI,具有较快的计算速度、良好的预测精度,容易部署、易于使用与整合。此外,本文还演示了如何在python界面里对两个同系物进行ADMET性质预测,并比较感兴趣的性质。
肖高铿/2024-12-05
ADMET-AI简介
对苗头化合物进行快速且准确的筛选,以获得理想的药物特性,对于推进成功率更高的分子至关重要1。具体而言,一个小分子要想成为治疗策略的核心并从发现阶段推进到临床试验阶段,该化合物必须具备优秀的吸收(Absorption)、分布(Distribution)、代谢(Metabolism)、排泄(Excretion)和毒性(Toxicity)等ADMET属性。为了解决这个问题,Swanson等人开发了一种简单、快速、精确的ADMET预测平台ADMET-AI2。

图1. ADMET-AI工作原理
ADMET-AI使用一种名为Chemprop-RDKit的图神经网络(图1)框架在 Therapeutics Data Commons (TDC) 的41个 ADMET 数据集上训练而得。在速度和准确性方面,ADMET-AI 超过了现有的大多数 ADMET 预测工具(见图2)。
图2. 在准确性和速度方面,ADMET-AI超过了现有的大多数 ADMET 预测工具
ADMET-AI还提供了其它特性,例如使用已批准上市药物作为参比进行 ADME 预测,将预测结果作为背景,以了解新化合物在上市药物背景下所处的位置。比如,你开发抗生素,那么用上市的抗生素比用上市的抗肿瘤药物作为毒性预测的背景参比是更有意义的。为了更直观地展示这些信息,ADMET-AI创建了一个散点图,该散点图基于用户选择的两个ADMET属性(图3)比较输入分子与DrugBank参比分子。参比数据集提供的背景表明,该输入分子相对抗肿瘤药而言具有可接受的毒性,但相对抗生素而言则毒性很高。

图3. ADMET-AI生成的散点图显示了输入分子(红色星号)与DrugBank批准药物参比数据集中的抗肿瘤药(左)以及抗生素(右)相比的毒性及吸收情况。参比数据集提供的背景表明,该输入分子相对抗肿瘤药而言具有可接受的毒性,但相对抗生素而言则毒性很高。
ADMET-AI的获取与使用方法
ADMET-AI可从软件仓库github下载3,根据说明文档在本地部署之后可以通过命令行、python界面或Web界面使用。
算例1. GSK3β抑制剂1与14的hERG抑制活性预测
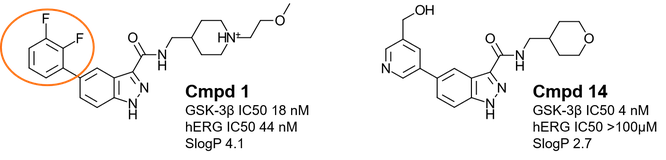
图4. 左:Cmpd 1是一种强效、选择性的GSK3β抑制剂,但却受到hERG不利的影响;右:优化后的Cmpd 14的GSK3β活性得以提高,并且降低了hERG的不利作用。
接下来,以Ombrato等人4报道的两个GSK3β抑制剂Cmpd 1与14(图4)为例,演示如何在Python界面里用ADMET-AI进行性质预测,并比较其中的tpsa、AMES、BBB与hERG等性质。
1 2 3 4 5 6 7 8 9 | import pandas as pd from admet_ai import ADMETModel model = ADMETModel() struct_1 = "Fc1c(F)cccc1-c2ccc3c(c(n[nH]3)C(=O)NCC4CC[NH+](CC4)CCOC)c2" struct_14 = "OCc1cncc(-c2ccc3c(c(n[nH]3)C(=O)NCC4CCOCC4)c2)c1" structs = [struct_1,struct_14] results = model.predict(smiles=structs) df = pd.DataFrame(results) props = ['tpsa','AMES', 'BBB_Martins','hERG'] |
其中第8行将预测结果保存为Pandas的Data Frame。然后将自己感兴趣4个性质作为一个数组,单独列出这4个性质:
1 2 3 4 | >>> df[props] tpsa AMES BBB_Martins hERG Fc1c(F)cccc1-c2ccc3c(... 71.45 0.314828 0.798180 0.975997 OCc1cncc(-c2ccc3c(c(... 100.13 0.120690 0.456809 0.726630 |
用Data Frame的columns可以查看ADMET-AI预测的性质种类:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 | >>> df.columns Index(['molecular_weight', 'logP', 'hydrogen_bond_acceptors', 'hydrogen_bond_donors', 'Lipinski', 'QED', 'stereo_centers', 'tpsa', 'AMES', 'BBB_Martins', 'Bioavailability_Ma', 'CYP1A2_Veith', 'CYP2C19_Veith', 'CYP2C9_Substrate_CarbonMangels', 'CYP2C9_Veith', 'CYP2D6_Substrate_CarbonMangels', 'CYP2D6_Veith', 'CYP3A4_Substrate_CarbonMangels', 'CYP3A4_Veith', 'Carcinogens_Lagunin', 'ClinTox', 'DILI', 'HIA_Hou', 'NR-AR-LBD', 'NR-AR', 'NR-AhR', 'NR-Aromatase', 'NR-ER-LBD', 'NR-ER', 'NR-PPAR-gamma', 'PAMPA_NCATS', 'Pgp_Broccatelli', 'SR-ARE', 'SR-ATAD5', 'SR-HSE', 'SR-MMP', 'SR-p53', 'Skin_Reaction', 'hERG', 'Caco2_Wang', 'Clearance_Hepatocyte_AZ', 'Clearance_Microsome_AZ', 'Half_Life_Obach', 'HydrationFreeEnergy_FreeSolv', 'LD50_Zhu', 'Lipophilicity_AstraZeneca', 'PPBR_AZ', 'Solubility_AqSolDB', 'VDss_Lombardo', ... |
其中”…”表示被省略的部分,这部分显示了上述预测值在DrugBank上市药物预测值百分位分布的位置。每个性质可以在TDC网站5上查询到数据集及其含义。
以hERG为例,可以从TDC网站的Toxicity数据集里知道这是一个分类模型,因此预测值表示概率。预测结果表明,Cmpd 1具有极高的概率(0.98)为hERG抑制剂,这与实验hERG IC50 = 44 nM一致;虽然Cmpd 14预测为hERG抑制剂的概率(0.73)有所降低,但依然分类为hERG抑制剂,这与实验值hERG IC50大于100μM不一致。
算例2. MET抑制剂13及其N-去甲基衍生物的P450酶抑制活性预测

图5. 辉瑞公司报道MET抑制剂13
Pfizer的Bumpers等人6发现了一种新型的脑渗透性MET抑制剂,其中化合物13(图5)表现出高选择性、克服了耐药性并且拥有良好的药代动力学特性。初步的CYP450混合试验未显示化合物13具有抑制CYP450酶的活性。然而,在研究时间依赖性抑制时,发现13对CYP3A4表现出高水平的时间依赖性抑制(time-dependent inhibition,TDI)活性,达到不可接受的水平。最终,这一活性被认为不适合进一步开发,导致了化合物13以及这一系列化合物的开发终止。
根据Bumpers等人6的研究,13对CYP3A4的TDI与其吡唑环的N-去烷基化代谢物有关,13及其代谢物对CYP3A4的Ki分别为44μM与105nM。很可能是N-去烷基化作用使得化合物13的吡唑氮暴露出来,从而使代谢物能够与CYP3A4的血红素铁形成强配位键。
用ADMET-AI预测13及其吡唑N-去甲基衍生物对几种常见的P450酶(CYP1A2、CYP2C19、CYP2C9、CYP2D6、CYP3A4)的抑制活性,从TDC网站的介绍可知是分类模型:
1 2 3 4 5 6 7 8 9 | import pandas as pd from admet_ai import ADMETModel model = ADMETModel() struct_13 = "Fc1cc(NC(=O)C2=CC=NN(c3ccc(F)cn3)C2=O)c(F)cc1N(C4CC(C4)c5ccn(n5)C)c6c7ccnn7ccn6" struct_13_met = 'Fc1cc(NC(=O)C2=CC=NN(c3ccc(F)cn3)C2=O)c(F)cc1N(C4CC(C4)c5cc[nH]n5)c6c7ccnn7ccn6' structs = [struct_13,struct_13_met] results = model.predict(smiles=structs) df = pd.DataFrame(results) props = ['CYP1A2_Veith','CYP2C19_Veith','CYP2C9_Veith','CYP2D6_Veith','CYP3A4_Veith'] |
结果如下(请拉滚动条,查看更多内容):
1 2 3 4 | >>> df[props] CYP1A2_Veith CYP2C19_Veith CYP2C9_Veith CYP2D6_Veith CYP3A4_Veith Fc1cc(NC(=O)C2=... 0.010217 0.036683 0.136446 0.019011 0.720289 Fc1cc(NC(=O)C2=... 0.037174 0.135819 0.463873 0.046976 0.849260 |
预测结果表明,化合物13(第3行)及其代谢物(第4行)为CYP1A2、CYP2C19、CYP2C9与CYP2D6抑制剂的概率较低,但为CYP3A4抑制剂的概率很高,尤其是去甲基代谢物。进一步与DrugBank中上市药物相比:
1 2 3 4 | >>> df[['CYP3A4_Veith_drugbank_approved_percentile']] CYP3A4_Veith_drugbank_approved_percentile Fc1cc(NC(=O)C2=... 88.134936 Fc1cc(NC(=O)C2=... 92.710353 |
预测分析显示,化合物13及其N-去甲基代谢物作为CYP3A4抑制剂的可能性分别高于DrugBank中88%和93%的已上市药物的预测值。这意味着,与大多数已上市药物相比,化合物13及其特定代谢产物更有可能是CYP3A4抑制剂,尤其是其代谢物。
表1. 化合物13在不同ADMET预测工具上的CYP450抑制剂预测结果
| Items | CYP1A2 | CYP2C19 | CYP2C9 | CYP2D6 | CYP3A4 |
|---|---|---|---|---|---|
| ADMET-AI2 | 0.010 | 0.037 | 0.136 | 0.019 | 0.720 |
| ADMETLab 3.07 | 0 | 1 | 1 | 0 | 0 |
| SwissADME8 | No | No | Yes | No | No |
| admetSAR 3.09 | 0 | 1 | 1 | 0 | 1 |
同时,在几个在ADMET预测服务器上也对化合物13及其去N-甲基代谢物进行了CYP450抑制剂的预测,结果分别见表1与表2。
表2. 化合物13的N-去甲基衍生物在不同ADMET预测工具上的CYP450抑制预测结果
| Items | CYP1A2 | CYP2C19 | CYP2C9 | CYP2D6 | CYP3A4 |
|---|---|---|---|---|---|
| ADMET-AI2 | 0.037 | 0.135 | 0.464 | 0.047 | 0.849 |
| ADMETLab 3.07 | 0 | 1 | 1 | 0 | 0 |
| SwissADME8 | No | No | Yes | No | No |
| admetSAR 3.09 | 0 | 1 | 1 | 0 | 1 |
ADMETlab 3.07均将化合物13及其N-去甲基衍生物的预测为非CYP3A4抑制剂,同时还将化合物预测为CYP2A9的抑制剂。SwissADME8也没有将化合物预测为CYP3A4的抑制剂,将化合物预测为CYP2C9与CYP2C19的抑制剂。admetSAR 3.09在将化合物13及其N-去甲基代谢物预测为CYP3A4抑制剂的同时,还预测为CYP2C9与CYP2C19的抑制剂。这些预测与Bumpers等人6对实验的描述不一致:初步CYP450混合试验未显示化合物13具有抑制CYP450酶的活性。总体上讲,在这个例子里,ADMET-AI2表现出最佳的性能。
算例3. 钙离子通道阻断剂的代谢稳定性:质子化状态影响预测结果
需要注意的是,提供给ADMET-AI预测性质的结构需要进行标准化处理,否则质子化状态会影响ADMET-AI的预测结果。这里以钙离子通道阻滞剂非洛地平(Felodipine, 图6)与氨氯地平(Amlodipine,图6)为例来说明这个问题。

图6. 降低钙离子通道阻断剂的亲脂性可降低肝代谢清除率,图片来源于文献[10]
降低亲脂性是改善清除率的重要策略之一10,这是因为大多数代谢酶都具有与亲脂性基团相结合的活性口袋,对于具有相同骨架或相似结构的化合物,其清除率往往与亲脂性正相关。如图6所示,非洛地平具有较高的亲脂性(cLog P = 5.30),非洛地平在犬体内的系统清除率较高,CL = 38 mL·(min·kg)-1,血浆半衰期仅为1小时。氨氯地平的亲脂性相比非洛地平降低约2个Log单位,犬体内系统清除率降低至11 mL·(min·kg)-1,仅为非洛地平的1/4,血浆半衰期延长至30小时。
其中氨氯地平的氨基侧链具有碱性,在生理条件下携带正电荷,因此在分子设计中通常表示为带正电的质子化状态(amlodipine_ionized),而在大多数性质预测软件中可能需要进行标准化处理表示为中性的分子(amlodipine_norm)。用ADMET-AI对非洛地平与氨氯地平的两个不同质子化状态结构进行性质预测:
1 2 3 4 5 6 7 8 9 10 11 | import pandas as pd from admet_ai import ADMETModel model = ADMETModel() felodipine = 'Clc1c(Cl)cccc1C2C(=C(NC(=C2C(OCC)=O)C)C)C(OC)=O' amlodipine_ionized = 'Clc1ccccc1[C@@H]2C(=C(NC(COCC[NH3+])=C2C(OCC)=O)C)C(OC)=O' amlodipine_norm = 'Clc1ccccc1C2C(=C(NC(=C2C(OCC)=O)COCCN)C)C(OC)=O' structs = [felodipine,amlodipine_ionized,amlodipine_norm] results = model.predict(smiles=structs) df = pd.DataFrame(results) df['Structures']=['felodipine','amlodipine_ionized','amlodipine_norm'] props = ['Structures','logP','Clearance_Hepatocyte_AZ','Clearance_Microsome_AZ','Half_Life_Obach'] |
查看预测结果(请拉动滚动条查看更多):
1 2 3 4 5 | >>> df[props] Structures logP Clearance_Hepatocyte_AZ Clearance_Microsome_AZ Half_Life_Obach felodipine 3.9643 90.972043 122.384993 0.421901 amlodipine_ionized 1.5495 103.991292 107.029545 -0.696006 amlodipine_norm 2.2663 84.927830 86.145041 17.916490 |
可以看到,质子化状态的比中性的氨氯地平计算得到更低的logP值、更高的人肝细胞与人肝微粒体清除率、同时也具有更低的半衰期。相比之下,标准化处理的氨氯地平结构预测值相当合理,氨氯地平比非洛地平的logP降低了1.7个对数单位,清除半衰期预测值也由0.42小时提高到17.9小时,这与实验值(狗)从1小时提高到30小时具有相同的趋势。总的来说,结构的标准化预处理对ADMET-AI的预测是非常重要的。
表3给了三个不同方法t1/2的预测结果,可以发现,除了ADMET-AI2之外,ADMETlab 3.07与admetSAR 3.09的预测结果也受输入分子结构的质子化状态影响。此外,ADMETlab 3.07与admetSAR 3.09都未能预测出Amlodipine比Felodipine具有更长的半衰期,这与实验结果不一致。
表3. Felodipine与amlodipine两个不同质子化状态的t1/2预测结果
| Items | Felodipine | Amlodipine-ionized | Amlodipine-norm |
|---|---|---|---|
| ADMET-AI2 | 0.42 | -0.70 | 17.9 |
| ADMETLab 3.07 | 0.51 | 0.57 | 0.48 |
| admetSAR 3.09 | -0.78 | -0.43 | -0.55 |
| Exp. Dog | 1 | 30 | |
单位:小时
算例4. 药物诱导肝损伤(DILI)的预测
药物诱导的肝损伤(Drug-induced liver injury,DILI)是由于药物引起的致命性肝脏疾病。在过去50年中,DILI一直是与安全相关的药品退市的主要原因之一 ,例如iproniazid、ticrynafen和benoxaprofen。ADMET-AI包含了一个DILI预测模型,该模型是在一个包含475化合物(物种:人类)的数据上训练而来,模型的AUCROC = 0.87(AUCPC=0.86)。接下来用这个模型预测减肥药Azelaprag(图7)潜在的DILI风险。

图7. Azelaprag的化学结构式
Apelin(艾帕素)是一种多肽,通过与骨骼肌、心脏以及中枢神经系统的相互作用调节新陈代谢,促进肌肉再生。Azelaprag是BioAge公司开发的一款口服的Apelin受体激动剂,当与肠促胰岛素类药物结合应用时,能显著促进体重下降并改善身体成分。在临床前研究中,Azelaprag能使通过肠促胰岛素治疗实现的体重减轻效果加倍,并进一步改善身体成分与肌肉功能。在1期临床试验中,Azelaprag已被证明能够在健康老年志愿者进行卧床休息期间,促进肌肉代谢,增加能量消耗,从而有效防止肌肉萎缩。BioAge公司与礼莱合作启动了一项2期临床试验,旨在评估Azelaprag与Tirzepatide联合使用,治疗肥胖症的效果。
用ADMET-AI对Azelaprag进行ADMET性质预测:
1 2 3 4 5 6 7 8 | import pandas as pd from admet_ai import ADMETModel model = ADMETModel() azelaprag = 'CC1=CC(=CN=C1)C2=NN=C(N2C3=C(C=CC=C3OC)OC)NS(=O)(=O)[C@@H](C)[C@H](C)C4=NC=C(C=N4)C' structs = [azelaprag] results = model.predict(smiles=structs) df = pd.DataFrame(results) props = ['DILI','DILI_drugbank_approved_percentile'] |
其中DILI预测结果如下:
1 2 3 | >>> df[props] DILI DILI_drugbank_approved_percentile CC1=CC(=CN=C1)C2=NN=C(... 0.971994 95.036836 |
可以看到,Azelaprag预测为DILI的概率高达97.2%,高于DrugBank里95%药物预测的DILI概率值。2024年12月6日,BioAge宣布终止azelaprag与tirzepatide联用治疗肥胖症的STRIDES二期临床试验12。根据报道13,主要原因是:在二期研究STRIDES中,接受azelaprag治疗的十一名受试者显示出了肝酶水平升高,这可能预示着潜在的器官损伤。研究者在使用BioAge药物作为单药治疗或与礼来公司销售的Zepbound(tirzepatide)联用的受试者中发现了肝酶升高,即肝转氨酶升高。仅接受Zepbound治疗的参与者中没有人出现肝转氨酶升高的情况。在临床上观察到的肝酶水平升高与DILI预测结果相符,Azelaprag自身很可能存在DILI导致的安全性风险。这是一个前瞻性的预测,让人看到计算方法在控制药物开发过程中潜在风险的价值。
避免误解的说明:本文只是讨论ADMET预测的技术问题,根据临床前的数据14,AMG 986(也就是azelaprag临床前开发代码)具有可接受的安全性特征,我不认为临床前存在对安全性评估不足的问题。
Azelaprag预测为DILI是科学还是巧合?
虽然ADMET-AI的DILI模型报道的性能相当不错,ROC AUC=0.87,但这只是综合性能(overall performance),真实性能需要看到ROC曲线图才能了解。以ATC中全部的2579个Drugbank上市药物(ATC code “all”)为参照背景,观察ADMET-AI的DILI预测值与HIA预测值(这只是我为了绘图而随便选了一个性质,当它不存在),结果如图8所示。可以看到,尽管Azelaprag预测为DILI的概率比95%的参照药物高,但是参照药物几乎以纵坐标0.5为中心对称分布,没有看到明显偏好,这一点从右边的纵坐标上的直方图上可以直观地看到,大约有50%的参比药物被预测为DILI。这显然不太可能,因此我认为模型的假阳性率过大,将azelaprag预测为DILI只是个巧合。相比之下,横坐标表示小肠吸收,从直方图可以看到绝大部分药物属于口服可吸收的,这是符合预期的分布。

图8. Azelaprag与2579个药物的ADMET-AI预测值分布图
这再次说明了,使用机器学习方法进行决策时非常有必要了解预测的不确定性, 无论预测结果是否符合自己的预期与喜好。
小结
目前已经有非常多的ADMET性质预测工具,包括商业软件、开源软件。本文介绍了一个开源、免费的工具ADMET-AI,具有较快的计算速度、良好的预测精度,容易部署、易于使用与整合。此外,本文还演示了如何在python界面里对一个或多个化合物进行ADMET性质预测,比较感兴趣的性质。需要注意的是,这是一个新工具,还缺乏与商业软件的性能比较,有待进一步在更大数据上的使用、验证与完善。
接下来可以作什么?
- 作为ADMET预测工具,对化合物进行排序
- 与Flare/Spark整合,对化合物进行优先级排序
- 与生成式AI整合,比如作为RL的打分函数成分,控制生成化合物的性质
- 投资者也可以用来对感兴趣的项目进行ADMET风险评估
文献
- Feinberg, E.N. et al. (2020) “Improvement in ADMET Prediction with Multitask Deep Featurization,” Journal of Medicinal Chemistry, 63(16), pp. 8835–8848. Available at: https://doi.org/10.1021/acs.jmedchem.9b02187.
- Swanson, K. et al. (2024) “ADMET-AI: a machine learning ADMET platform for evaluation of large-scale chemical libraries.” Available at: https://doi.org/10.5281/zen.
- https://github.com/swansonk14/admet_ai
- Ombrato, R.; Cazzolla, N.; Mancini, F.; Mangano, G. Structure-Based Discovery of 1 H -Indazole-3-Carboxamides as a Novel Structural Class of Human GSK-3 Inhibitors. J. Chem. Inf. Model. 2015, 55 (12), 2540–2551. https://doi.org/10.1021/acs.jcim.5b00486.
- https://tdcommons.ai/single_pred_tasks/overview
- Bumpers, Q.A. et al. (2024) “Discovery of Pyrazolopyrazines as Selective, Potent, and Mutant-Active MET Inhibitors with Intracranial Efficacy,” Journal of Medicinal Chemistry, 67(16), pp. 14466–14477. Available at: https://doi.org/10.1021/acs.jmedchem.4c01232.
- ADMETLab 3.0. https://admetlab3.scbdd.com/server/evaluationCal. Accessed 6 December 2024.
- SwissADME. http://www.swissadme.ch. Accessed 6 December 2024.
- admetSAR3.0. https://lmmd.ecust.edu.cn/admetsar3. Accessed 6 December 2024.
- 张蕊,王江,朱浩然,柳红. 先导化合物结构优化策略(九)——改善药物清除率. 药学学报 Acta Pharmaceutica Sinica. 2021, 56(11):3030−3046.
- ADMET-AI. https://admet.ai.greenstonebio.com. Accessed 6th December 2024.
- BioAge Labs Announces Discontinuation of STRIDES Phase 2 Clinical Trial Evaluating Azelaprag in Combination with Tirzepatide for the Treatment of Obesity. 2024 Dec 6th. . Available at: https://ir.bioagelabs.com/news-releases/news-release-details/bioage-labs-announces-discontinuation-strides-phase-2-clinical
- Gwendolyn Wu. BioAge shares tumble after decision to halt obesity drug study. 2024 Dec 7th. Available at: https://www.biopharmadive.com/news/bioage-azelaprag-tirzepatide-study-2-stop-obesity/734931
- Winkle P, Goldsmith S, Koren MJ, Lepage S, Hellawell J, Trivedi A, Tsirtsonis K, Abbasi SA, Kaufman A, Troughton R, Voors A, Hulot JS, Donal E, Kazemi N, Neutel J. A First-in-Human Study of AMG 986, a Novel Apelin Receptor Agonist, in Healthy Subjects and Heart Failure Patients. Cardiovasc Drugs Ther. 2023 Aug;37(4):743-755. doi: 10.1007/s10557-022-07328-w. Epub 2022 Apr 23. PMID: 35460392.
原创文章,作者:小墨,如若转载,请注明出处:《开源软件ADMET-AI的介绍》http://blog.molcalx.com.cn/2024/12/05/admet-ai.html




