摘要:Pat Walters的这篇博客探讨了生成式 AI 在药物设计中的实际应用,尤其是针对媒体上关于《生成式 AI 将在不久的将来独立设计新药》的夸张报道提出了质疑。作者指出,尽管生成式 AI 展现了一定潜力,但其操作远比媒体报道复杂,当前技术尚未达到完全自主设计新药的程度。

原文:Pat Walters. May 22, 2024. Generative Molecular Design Isn’t As Easy As People Make It Look. Practical Cheminformatics. https://practicalcheminformatics.blogspot.com/2024/05/generative-molecular-design-isnt-as.html
编译:肖高铿
我被最近一篇 CNBC 的文章《生成式 AI 将在不久的将来独立设计新药》所震惊。我知道不应该关注大众媒体上关于人工智能的文章,但我认为即使是从事药物发现的科学家们也可能对生成式 AI 能做什么和不能做什么的看法存在偏差。为了准确理解其中涉及的内容,逐步讲解一个典型的生成分子设计工作流程,并指出一些关键点可能会有所帮助。首先,这些程序远非自主运行。即使面对定义明确的问题,生成算法也会产生大量无意义的结果。其次,在筛选生成算法产生的分子时,领域专业知识是必不可少的。没有深厚的药物化学背景,人们无法理解这些结果。第三,虽然生成模型的输出中确实存在一些有价值的信息,但要提取它们仍需要大量的工作以及传统的化学信息学。
在这个特别的练习中,我检查了 DiffLinker 的输出,这是一个由 Ilia Igashov 及其同事最近发表的程序。DiffLinker 使用扩散模型来生成可以连接两个或更多化学片段的链接臂(linkers)化学结构。这类方法在药物发现中有广泛的应用,包括基于片段的设计、PROTAC 优化以及肽骨架的替换。DiffLinker 的一个令人兴奋的特点是它能够在三维空间中工作。与一些其他生成一维结构(SMILES)然后转换为三维进行打分的模型不同,DiffLinker 直接对三维结构进行操作。此外,如果提供了结合位点,DiffLinker 还可以在蛋白质结合位点的背景下执行片段链接,并避免与蛋白质发生空间冲突。DiffLinker 的输入是一组化学片段和连接原子。下图说明了这一点,显示了两个结合到 HSP90 的片段实验结构,HSP90 是一种与肿瘤生长有关的分子伴侣。图中洋红色高亮显示的两个原子就是 DiffLinker 将要连接的原子。
将我们维系在一起的键
重要的是要记住,DiffLinker 并不是直接在分子上操作,而是在点云上进行处理。这个点云简单来说就是一组空间中的点,每个点都有一个关联的原子类型。扩散模型使用从实验晶体结构中学习到的分布来拟合一组具有定义原子类型的点。因此,DiffLinker 没有“键”或“键级”的概念。它的输出是一组 x, y, z 坐标和元素类型。为了能够与分子建模软件一起使用,DiffLinker 的输出必须经过转换,这包括归属键和键级。这比看起来要复杂得多。
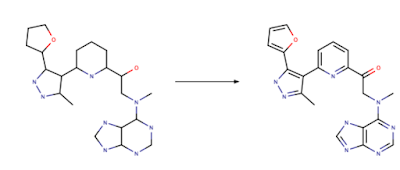
键的归属相对较为简单。我们可以遍历所有原子对,并在任意两个原子之间的距离小于共价半径之和加上一定的容差(通常是 0.4-0.5 Å)时归属一种键。然而,键级的归属则棘手得多。乍一看,这似乎很简单:双键和三键的键长更短。我们应该能够创建一个查询表,将键长与键级关联起来并归属键级。但当我们考虑到带有交替单键和双键的共轭π体系时,问题就不那么简单了。从下面左边的结构到右边的结构的转变并非轻而易举。Roger Sayle 在 2001 年的一份白皮书中提供了关于键和键级归属问题的更完整描述。
当然,敏锐的读者现在可能会想,“我们有可靠的开源化学信息学工具包可以解决这个问题,对吧?”不幸的是,答案是“并不是这样”。RDKit 和 Open Babel 都可以进行键和键级归属。但是,这两个程序主要是设计用来处理包含氢原子连接到相应重原子的量子化学计算输出的。如果我们给 RDKit 一个来自 DiffLinker 的 XYZ 文件,它会搞得一团糟。

这并不是说 RDKit 中的程序是错误的;只是这不是它们被设计来执行的任务。OpenBabel 的表现稍微好一些,但仍存在一些问题。下面是 OpenBabel 对同一 XYZ 文件的转换结果。
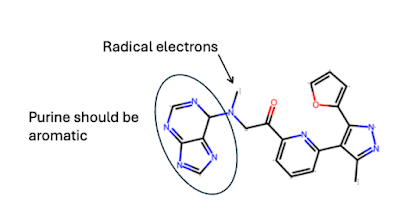
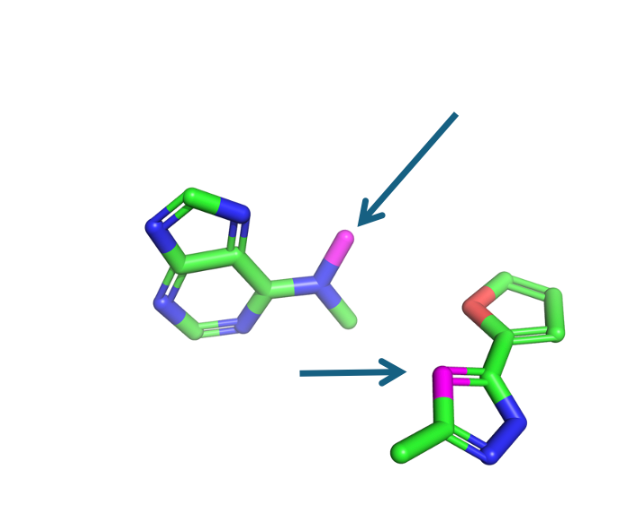
键级归属几乎正确,但请注意,在应该存在氢原子的位置出现了自由基。我们可以使用一些方法来填充这些氢原子,但是 OpenBabel 仍然存在一些问题。在上述例子中,DiffLinker 将左侧的嘌呤与右侧的吡唑-呋喃连接起来。请注意,嘌呤的键级归属是不正确的。这种错误的键级归属会使得生成分子的进一步处理极大地复杂化。
经过几次尝试开源软件失败后,我转向了商业软件解决方案。我发现解析 XYZ 文件并归属键级的最佳工具是 OpenEye Scientific Software 的 OEChem 工具包。使用 OEChem,我可以解析 XYZ 文件,并正确地归属键级。
发现重复的结构
在运行 DiffLinker 时,用户可以指定要生成的解决方案数量。由于我希望有多个选择,我使用 “-output”标志指定了 1,000 个输出分子。上述 OEChem 工作流程使我可以解析 DiffLinker 的输出并生成基本正确的结果。我首先要做的是去除重复的分子设计。为了实现这一点,我为每个分子生成了一个 InChI Key,并统计了每个 InChI Key出现的次数以识别重复项。也可以使用 isomeric SMILES 来识别重复项,但我选择了 InChI Key,因为它能够规范化互变异构体( normalizes tautomers ),提供更稳健的去重方法。
下面是根据 InChI Key 统计得到的出现频次最高的 10 个分子及其生成次数的表格(别担心,我们稍后会查看这些结构):
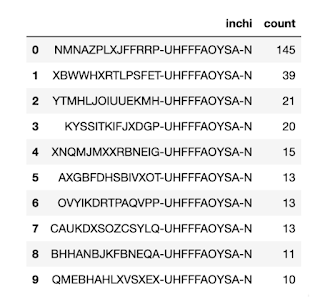
如表格所示,第一个结构被生成了 145 次。当然,比较 InChI key仅能告诉我们相同的分子图被多次生成。有可能同一个分子在结合位点可以采取不同的姿态和/或构象。一种调查方法是计算所有结构对之间的均方根偏差(RMSD),并检查 RMSD 分布。如果我们这样做,最终会得到如下所示的分布图。从图中可以看出,RMSD 值小于 0.3 Å,这表明所有副本具有相似的构象和姿态。

为了确认这些数字,我写出了所有 145 个副本的结构并在 PyMol 中进行了可视化。确实,这些副本的姿态和构象是相同的。细心的读者可能会注意到下面的分子在化学上并不稳定;我们稍后会讨论这一点。
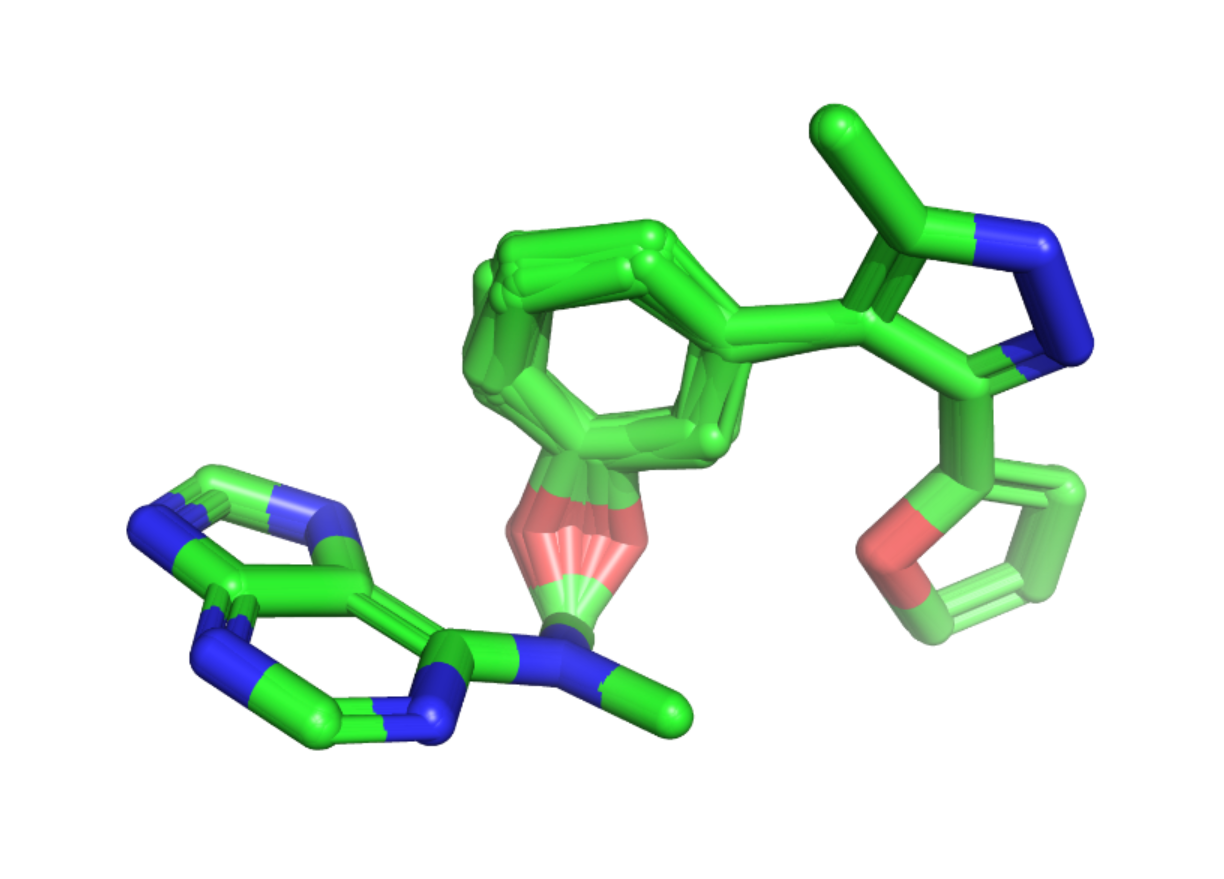
经过几次抽样检查后,我感到只保留一份重复结构的副本是安全的。由于数据存储在一个 Pandas 数据框中,使用 drop_duplicates 方法很容易从表中删除具有相同 InChI Key的行。删除重复项后,我们剩下 581 个独特的分子。
给它加个环吧
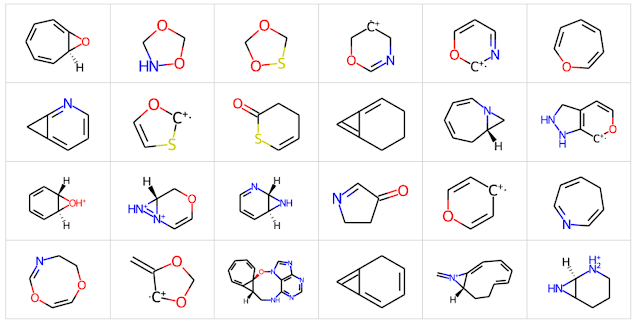
分子生成算法的一个令人担忧的方面是它们倾向于产生包含不稳定环系的分子。这种不稳定性有时可归因于环张力。然而,在许多情况下,这是由于化学上不稳定的键或部分取代的芳香系统,这些系统可能氧化为更稳定的芳香形式。为每个由生成模型想象出来的奇特环系创建评估其稳定性的规则是非常困难的。与其试图过于聪明,我设计了一个简单的方法,并在之前的帖子中详细描述了这个方法。简而言之,我编写了一种算法来从分子中提取环系。一旦提取出来,每个环系与包含 ChEMBL 数据库中所有环系及其频次的查找表进行比较。如果一个环系不出现在 ChEMBL 中,那么它可能是不稳定的或者难以合成。在这两种情况下,它可能不会引起药物化学家的兴趣。
为了评估去重后剩余的 537 个分子中的环系,我使用了 useful_rdkit_utils 工具包中的 RingSystemLookup 类。有 144 个生成的分子包含在 ChEMBL 中出现次数少于 100 次的环系。下图展示了一些不在 ChEMBL 中出现的环系的例子。即使是一瞥也能看出,这些不是任何人能够合成的分子。有人可能会问,是否要求环系在 ChEMBL 中出现 100 次才被视为有效是一个过于严格的标准。相反,正如我们将在下文看到的,100 可能还不够严格。
过滤器,过滤器,过滤器
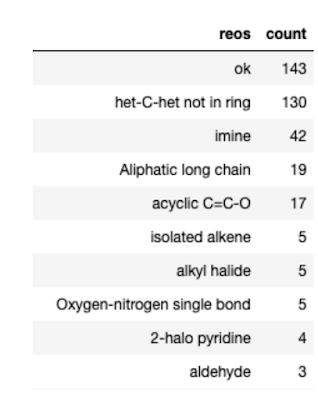
当我开始查看 DiffLinker 的结果时,我注意到许多分子包含化学上不稳定的官能团。为了识别并移除这些分子,我转向了 useful_rdkit_utils 工具包中的 REOS(快速淘汰废物,Rapid Elimination of Swill)功能。REOS 包含了几组在药物发现中存在问题的官能团集合,其中包括已知具有反应性、毒性或干扰生物测定的基团。REOS 类提供了多个过滤器合集。“Dundee”过滤器集对我来说最为熟悉,也成为了我的默认选择。应用 Dundee 过滤器后,剩余的 537 个分子中只有 143 个符合标准并被保留了下来。下表显示了淘汰最多分子的规则。
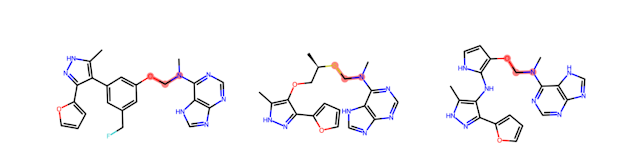
‘het-C-het’ 规则代表了 SMARTS 模式 “[NX3R0,NX4R0,OR0,SX2R0][CX4][NX3R0,NX4R0,OR0,SX2R0]”,该模式匹配由两个杂原子夹在中间的非环状脂肪族碳,如缩醛、缩酮、氨基甲酸酯、半缩醛和二醇等功能团。这些功能团在酸性条件下容易水解,可能导致分解。下图展示了几个带有高亮显示的‘het-C-het’ 功能团的例子。
然而,这种过滤可能过于严格。Wu 和 Meanwell 在 2021 年《J Med Chem》的一篇论文中指出,并不是所有的‘het-C-het’ 功能团都不稳定。他们提到超过 90 种上市药物含有这些连接键,并强调了稳定这些键的几种策略。正如本文详细描述的许多策略一样,实际情况要复杂得多。
高的构象张力能
使用上述过滤器,我将最初的 1,000 个分子列表缩减到了 143 个。在检查剩余的分子时,我注意到有几个几何错误,其中键长或键角不是标准值,导致高张力能的结构。一个例子如下图所示,芳香环取代基不在平面上,从环上出来的键角远非理想的 120 °。
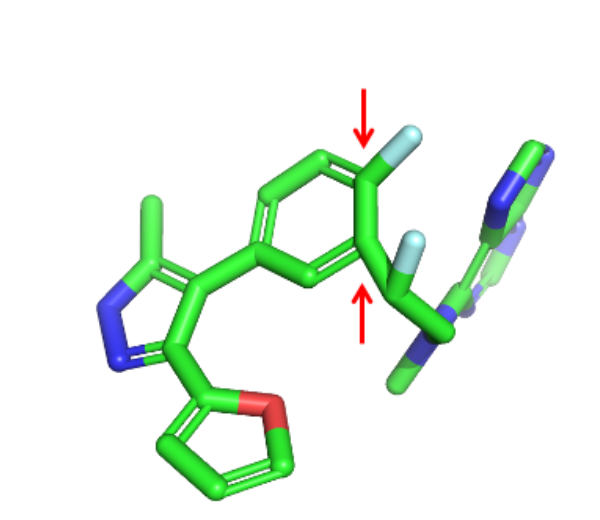
幸运的是,有一个开源软件库可以用来识别这些类型的不规则性。Martin Buttenschoen 及其同事发布的 PoseBusters 软件有 19 项测试来检测结构错误,包括键长、键角、内部空间立体冲突以及蛋白质与配体之间的冲突。对剩余的 143 个分子运行 PoseBusters 测试又淘汰了 59 个,剩下 88 个通过了所有过滤器的分子。除了 PoseBusters 之外,对结构进行扭转张力能过滤可能也是一个好主意。这个任务可以通过开源 MayaChem Tools 套件中提供的软件轻松完成。
我们现在处于什么位置?
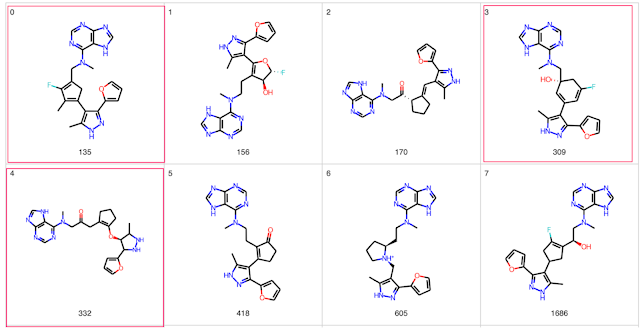
在这个阶段,查看剩余的 88 个分子以确定哪些可能进行实验性跟进相对容易。然而,在查看通过了之前过滤器的分子时,我对使用环系过滤器时设定的 ChEMBL 示例 100 次阈值产生了疑问。下表展示了剩余 88 个分子中具有最低 ChEMBL 环系频次的 8 个分子。结构下方的数字表示链接臂环系在 ChEMBL 中出现的频次。许多出现次数少于 500 次的环系并不稳定。令人惊讶的是,经典 Diels-Alder 加成物环丁二烯出现在 135 个 ChEMBL 分子中。我也对下面高亮显示的饱和杂环和部分饱和芳香链接臂不太满意。
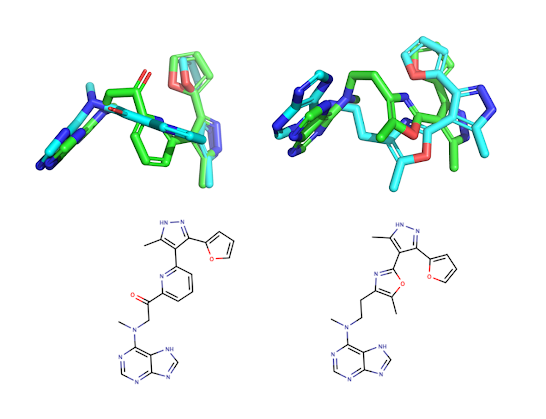
到目前为止,本次研究出现了两个有趣的主题。它们都是简单的模式,将一个五元或六元杂环与一个短的非环状连接臂结合在一起。下图展示了两个例子。为了评估 DiffLinker 构象的稳定性,我使用 RDKit 中实现的 ETKDG 方法为每个分子生成了 50 个构象。每个构象都用 MMFF 力场进行了最小化,并与相应的 DiffLinker 结构进行了叠合。下图展示了一个 RMSD 最低的构象叠合图。DiffLinker 结构用绿色表示,最接近的低能量构象则用蓝色叠合显示。可以看出,即使经过 PoseBusters 过滤后,这些结构仍然存在张力。右边的绿色结构包含一个明显重叠的单键,看起来不怎么稳定。如果这是一个“真实”的项目,我会转向基于物理的方法,并执行分子动力学模拟来检查配体姿态在结合位点中的稳定性。
通过这个冗长的练习,我试图证明生成式分子设计不是一个无缝的过程。我们不会简单地将生成模型指向一个蛋白质结合位点并等待药物自行产生。需要大量的领域专业知识和耐心来筛选生成模型的输出,并识别出有希望合成的分子。即便是在上述工作之后,仍有更多的事情要做。请注意,我们在过程中没有整合任何关于可合成性的评估。虽然有自动设计有机合成的方法,但这些工具尚未获得主流认可。最终,如何合成一个分子的最佳仲裁者是有经验的有机化学家。
上面的过滤工作流程提供了一种处理生成模型输出的方法。另一种方法是使用人机协作机器学习来开发能够区分理想和不理想分子的模型。2023 年,微软和诺华的一个团队描述了 MolSkill 方法,该方法使用人类反馈来建模分子的药物相似性。作为这项练习的一部分,35 名诺华药物化学家被要求对分子对进行排名并选择他们偏好的分子。此输入随后用于训练机器学习模型,以识别理想的分子特征。对于那些想了解更多 MolSkill 的人,请参考我在 2023 年写的一篇文章。
ACD 0-1-2-3..4-5
2022 年,我和同事们在《J Med Chem》上发表了一篇论文,定义了沿两个坐标轴的自动化化学设计(ACD,Automated Chemical Design)框架:
- 想法 – 是由人还是机器提出分子的想法?
- 选择 – 是由人还是机器决定合成哪些分子?
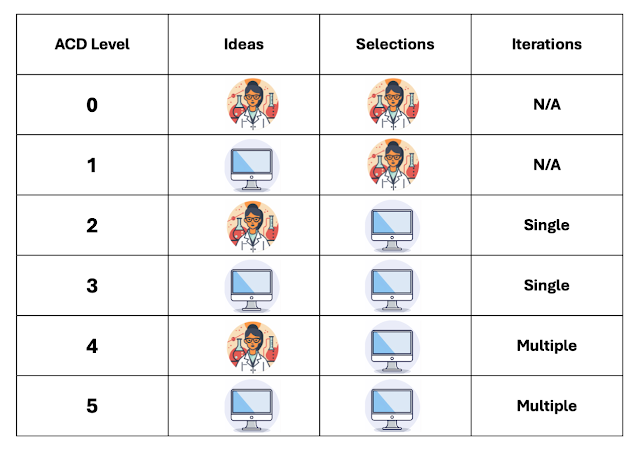
我们根据这两个维度定义了六个 ACD 级别,如下面的图表所示。
- 0. 想法和选择均由人定义。
- 1. 机器提供想法,然后由人选择。
- 2. 人定义可用的化学空间(例如反应),选择由机器做出。
- 3. 机器提供想法并且也由机器优先排序。
- 4. 与级别 2 相同,但有多次迭代。
- 5. 与级别 3 相同,但有多次迭代。
我们看到的大多数大众媒体和科学新闻报道似乎表明生成模型操作在 ACD 级别 3 或更高,但这并不是实际情况。就像本帖前面详细描述的例子一样,大多数生成分子设计论文可以归类为 ACD 级别 1。在这些出版物中,算法生成符合某些标准的新分子,例如适应结合位点。然后由人工手动筛选生成的分子,并决定合成哪些化合物。像“通过机器学习发现XX靶标抑制剂”之类标题的文章,当最终的选择是由拥有数十年药物发现经验的科学家驱动时,感觉有些不真诚。
需要注意的是,我在这里强调的问题并非 DiffLinker 所独有。我试验过的许多生成算法都表现出类似的问题。生成式 AI 有用,但它仍处于初级阶段。正如我的一位同事所说:“这些模型需要很多‘儿童防护’。” 尽管可以通过设置安全门来移除一些模型产生的愚蠢分子,但仍需要人工策展。Gary Marcus 曾说大型语言模型(LLMs)“响应的是语料库相似性,而非概念。” 我们也应该以同样的方式思考分子生成。这些模型不是在学习化学;它们只是在学习 SMILES 字符串或空间点中的模式。虽然生成模型提供了重要的创意来源,但我们距离能够独立设计分子的算法还有很长的路要走。
致谢
感谢 Jacob Gora、Hakan Gunaydin、Ilia Igashov、Steven Kearnes、Mark Murcko、Patrick Riley 和 Rebecca Swett 的有益讨论。
代码获取
本分析中使用的代码可在 GitHub 上获取:filter_difflinker.ipynb



