摘要:本文介绍了Flare MD模拟对膜蛋白模拟支持的一些特性。
作者:Keverne Louison/2023-01-10
编译:肖高铿
前言
膜蛋白的分子动力学(Molecular Dynamics,MD)是Flare最近引入的一个新功能。膜蛋白包括GPCR和离子通道。膜蛋白不仅在细胞生物学中发挥着关键作用,而且约20%的人类基因组编码膜蛋白。就治疗靶点而言,膜蛋白具有重要意义1。
膜蛋白的MD模拟可以表征和识别蛋白和脂质双层之间的相互作用,并深入了解它们在天然环境中的功能2。在含有膜结合蛋白的模拟中将膜考虑在内有利于重现实验蛋白的环境,这可以积极影响蛋白的一些三级结构特征,例如稳定性。
为什么在Flare中跑分子动力学模拟
Flare消除了命令行界面的技术障碍,使得用户能够使用直观的对话框和3D查看器来设置分子体系。在整个设置过程中用户可以使用3D查看器监控体系,并使用简单的文本框和下拉菜单修改参数。
Flare还整理了几个PDB资源,允许在应用程序中下载和准备所需的蛋白结构。MD轨迹可保存为广泛使用的文件格式,可以在应用程序中轻松查看和分析。通过这种方式,Flare可以帮助您将MD实验的各个方面都放在一个易于导航的地方。
在Flare中设置分子动力学模拟作业
由于用户友好的GUI,设置和启动MD模拟既简单又直观。首先从OPM(Orientation of Membrane Proteins)服务器3下载PDB文件,这将给出一个指示,说明上膜和下膜相对蛋白质的位置。
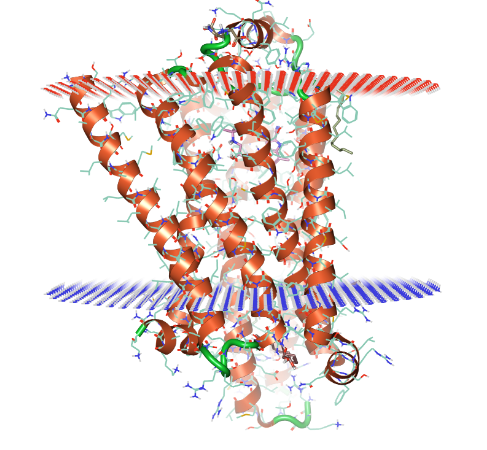
图1. 禽类β1-肾上腺素受体(PDB 5a8e)与OPM膜方向示意图。
一旦蛋白下载、准备并进行任何手动修改后,可以从GUI的蛋白选项卡中选择动力学。
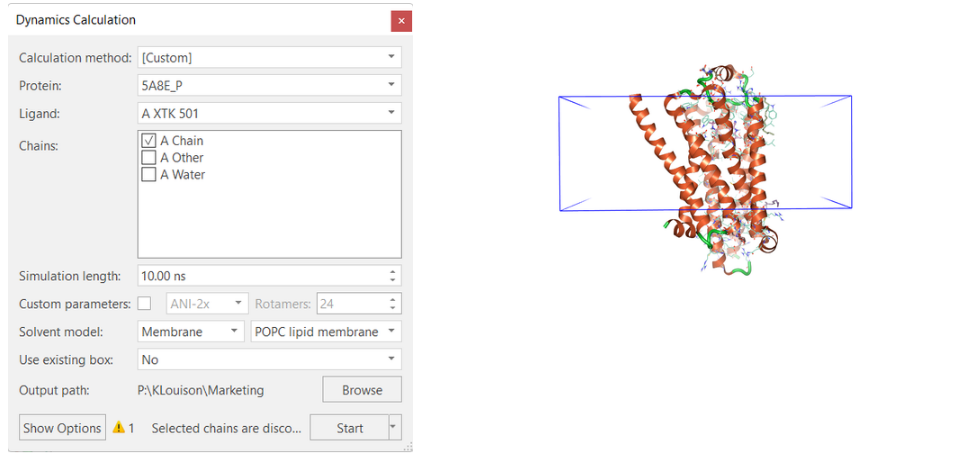
图2. 左:Flare中MD计算的示例对话框。右:蛋白以及用蓝色方框来指示的膜。
图2-左显示了在Flare中运行MD模拟的初始对话框。由此,用户可以选择他们想要在哪个蛋白、配体、链和溶剂模型上运行MD模拟,以及以纳秒为单位的模拟长度。单击对话框左下角的“Show Options”可以访问高级选项。高级选项允许用户修改参数,如力场、盒子尺寸和时间步长。Flare用户手册中提供了各种可自定义参数。
点击“Start”后,Flare开始配体参数化。如果使用OpenFF和力场初始化,则允许实现用户定义的自定义参数。Cresset目前支持OpenFF和AMBER力场。一旦这些步骤完成,Flare将启动MD计算。
在Flare中分析分子动力学模拟结果
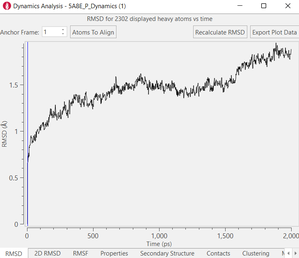
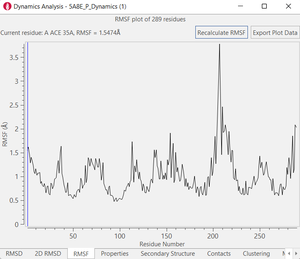
MD模拟完成后,可以在Flare GUI中分析轨迹.dcd文件。常见的分析工具包括各种诸如RMSD、RMSF、势能、用户定义的特定的原子/残基之间距离等性质的时间序列图的绘制,还有接触、二级结构和聚类分析等等。

 |
 |
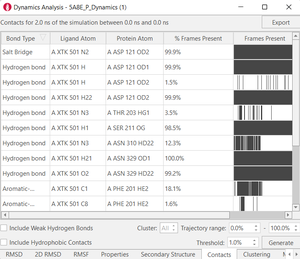 |
图3. 计算后分析的例子。上:目视检查;左下:RMSD图;中下:用户指定残基的RMSF;右下:蛋白-配体接触。
文献
- Chavent, M.; Duncan, A. L.; Sansom, M. S. P. Molecular Dynamics Simulations of Membrane Proteins and Their Interactions: From Nanoscale to Mesoscale. Current Opinion in Structural Biology. Elsevier Ltd October 1, 2016, pp 8–16. https://doi.org/10.1016/j.sbi.2016.06.007.
- Stansfeld, P. J.; Sansom, M. S. P. Molecular Simulation Approaches to Membrane Proteins. Structure. November 9, 2011, pp 1562–1572. https://doi.org/10.1016/j.str.2011.10.002.
- Lomize, M. A.; Pogozheva, I. D.; Joo, H.; Mosberg, H. I.; Lomize, A. L. OPM Database and PPM Web Server: Resources for Positioning of Proteins in Membranes. Nucleic Acids Res 2012, 40 (D1). https://doi.org/10.1093/nar/gkr703.



