摘要:本文以选择性CDK7抑制剂CT7001为例,来说明如何用基于配体的蛋白叠合方法来比较CT7001与CDK7/CDK2的结合模式差异与靶标选择性,并与基于蛋白结构的叠合方法进行了比较。总的来说,基于配体的蛋白叠合比其基于结构的蛋白叠合在结合位点上具有更好的叠合结果,可以捕捉住结合位点的局部特征,并用来解释CT7001对CDK7/CDK2的SAR与激酶选择性。
肖高铿/2023-04-26
在CT7001的CDK7/CDK2激酶靶标选择性分析一文中1,我们介绍过CT7001以一个优势的构象与CDK7(PDB 7B5Q)结合,而以一个高能的构象与CDK2(PDB 5JQ5)结合,这是从配体的张力能角度来解释CT7001的靶标选择性。
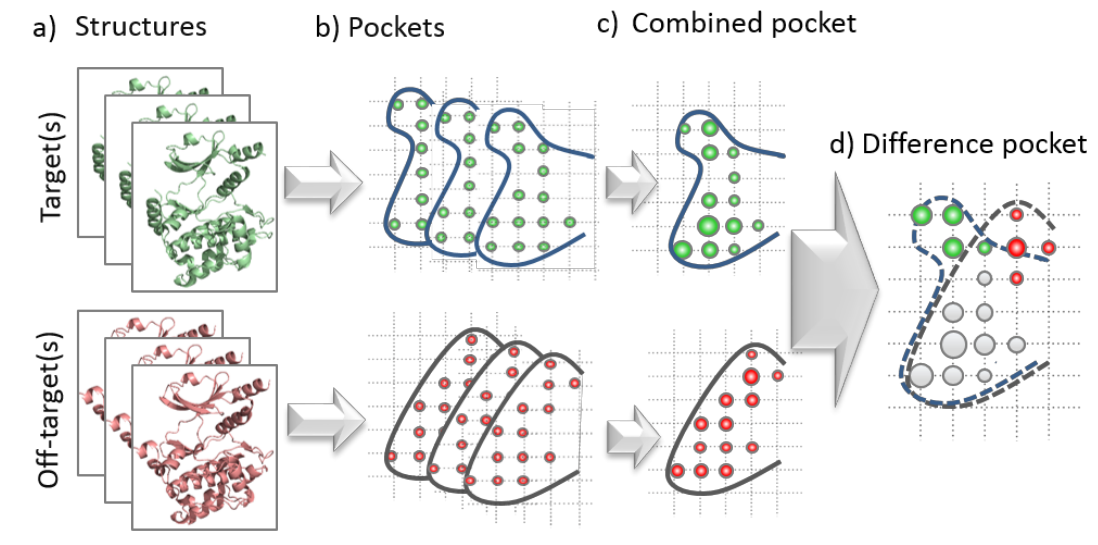
图1. 选择性子口袋分析法示意图2
通过比较CT7001与CDK7/CDK2结合位点的差异是另一种理解靶标选择性的方法,比如常用的选择性子口袋分析方法2,3,这需要用到蛋白叠合以便进行子口袋比较,如图1所示。目前有两种叠合方法:1)基于蛋白3D结构的叠合,比如按照Cα原子进行偏最小二乘拟合,然后结合位点的配体跟随着蛋白移动而叠合;2)基于配体的蛋白叠合,将配体进行叠合,然后蛋白跟随着配体移动过来而得以得叠合。基于蛋白结构的叠合是一种全局比较法,其缺点是通常不能照顾到更加保守的结合位点局部特征,导致局部叠合很差。为了克服这个问题,还经常采用基于结合口袋残基的叠合方法,而叠合的效果通常取决于残基的选择。而基于配体的蛋白叠合则刚好相反,可以捕捉到结合位点局部特征,通常更容易用来解释SAR、蛋白功能与靶标选择性。
常用的可视化软件都提供基于蛋白结构的叠合方法,比如Pymol就提供了多种方法进行蛋白叠合,其中align命令先进行序列比对、再进行结构叠合4,适合于序列一致性达到30%以上的场景。PDB 7B5Q与5JQ5的蛋白序列一致性在44.86%,因此我们先用align命令进行蛋白叠合:
1 2 3 4 5 | >align 7b5q 5jq5, \ cycles=5, \ cutoff=2.0, \ mobile_state=0, \ target_state=0 |

图2. align命令进行蛋白叠合之后结合位点里的CT7001的构象比较
可以看到蛋白叠合之后,叠合位点里的配体也跟着蛋白而叠合过来。可以发现结合位点的相关关键残基叠合不佳,比如CDK7的绞链区关键残基MET94与CDK2的LEU83的氢键供体与受体就叠合的不好(没有呈现)。除此之外,CDK7与CDK2的结合位点里的配体叠合结果也不佳:如图2所示,与绞链区发生氢键相互作用的片段在3D空间上是分开的,难以用来解释SAR。
蛋白叠合CE算法适合于蛋白序列相似性低时的蛋白叠合任务,因此尝试用Pymol实现的CE算法(cealign)来进行叠合5:
1 2 3 | >cealign 7b5q 5jq5, \ mobile_state=0, \ target_state=0 |
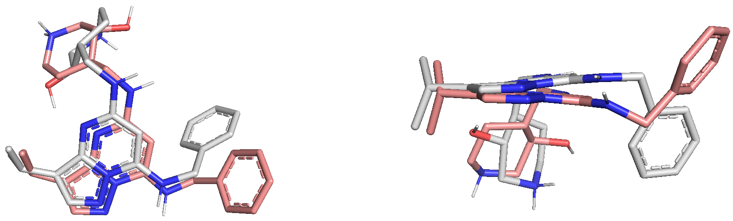
图3. cealign命令进行蛋白叠合之后结合位点里的CT7001的构象比较
叠合结果如图3所示,可以看到配体的叠合有所改善,部分原子叠合地更加紧密了,但是依旧并不佳:与绞链区发生氢键相互作用的片段在3D空间上还是分开的。
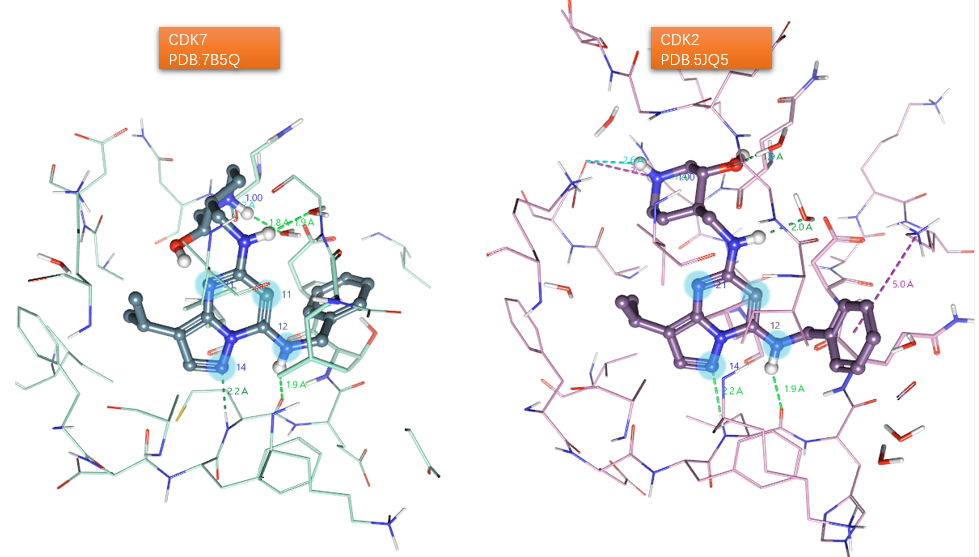
图4. 高亮的4个配体原子用来指导基于配体的蛋白叠合
在Flare的GUI提供了两种蛋白叠合方法:1)序列比对,再按Cα叠合;2)序列比对,再按选定的残基进行拟合。前者可自动叠合,后者需要手动选择残基然后再叠合。除了使用GUI界面进行蛋白叠合之外,Flare还提供了API可以自定义叠合方法。此前,Scott Midgley演示了如何用Flare的Python API进行基于配体的蛋白叠合5。这里,我选择了CT7001的四个配体原子(如图4所示)用Scott Midgley的方法进行了基于配体的蛋白叠合。
其中配体部分叠合的效果如图5所示,可以看到与CDK7/CDK2绞链区发生氢键相互作用的片段几乎完全重合在一起(图5-右),主要差异是苄基与哌啶环的取向不同。同时可以看到CDK7/CDK2蛋白与CT7001发生关键相互作用的残基在空间上的位置也更具可比性(图5-左)。
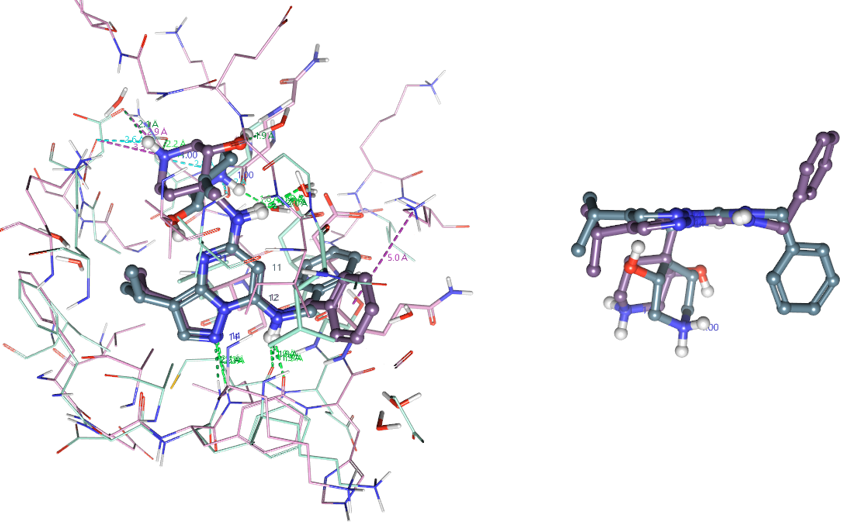
图5. CT7001与CDK7/CDK2的结合构象在结合位点里的叠合结果
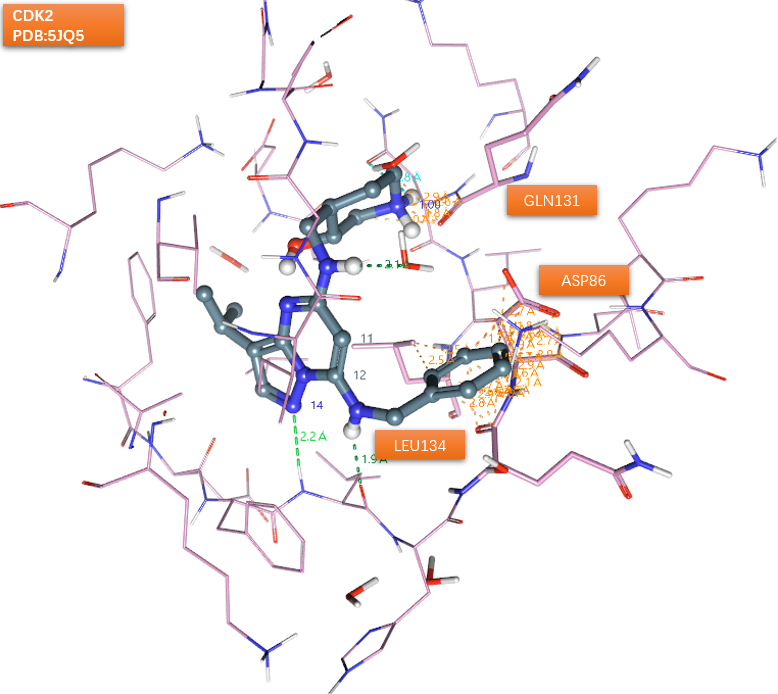
现在,将CT7001的CDK7活性构象与CDK2的结合位点进行比较,结果如图6所示,可以发现CT7001哌啶环的质子化氨基部分与CDK2的GLN131发生了严重的立体冲突,这可以解释为什么CT7001与CDK2结合时会不惜破环分子内氢键、母核与哌啶环之间连接臂采取了高能构象也要改变哌啶环取向。CT7001苄基上的苯环也与CDK2的ASP86、LEU134残基与发生严重的立体冲突。这可解释与CDK2结合时为什么苄基的取向会发生变化。
图6. CT7001的CDK7活性构象与CDK2的结合位点比较
图7是基于蛋白序列比对再进行结构叠合的结果,可以看到叠合结果有更多的噪音。一方面,由于结合位点残基局部的叠合偏差大,导致CT7001的CDK7结合构象不能与CDK2结合位点里的铰链区发生氢键相互作用,这不能解释SAR。另一方面,由于配体出现的位置不同,立体冲突也不同。其中,CT7001的母核与CDK2残基GLY11、VAL13、ILE10、ALA31发生严重的立体冲突;苄基上的苯环与LYS89发生立体冲突;而哌啶环没有发生任何立体冲突。这个叠合结果不能解释为什么CT7001的哌啶环与CDK2结合时需要改变取向,因而不能正确解释SAR与选择性。

图7. CT7001的CDK7活性构象与基于蛋白3D结构叠合的CDK2结合位点比较
总的来说,基于配体的蛋白叠合比其基于结构的蛋白叠合在结合位点上具有更好的叠合结果,可以捕捉住结合位点的局部特征,并用来解释CT7001对CDK7/CDK2的靶标选择性。
文献
- 肖高铿. CT7001的CDK7/CDK2激酶靶标选择性分析. 墨灵格的博客. 2022-05-31. http://blog.molcalx.com.cn/2022/05/31/cdk7-inhibitor-ct7001.html
- Volkamer, A.; Eid, S.; Turk, S.; Rippmann, F.; Fulle, S. Identification and Visualization of Kinase-Specific Subpockets. J. Chem. Inf. Model. 2016, 56 (2), 335–346. https://doi.org/10.1021/acs.jcim.5b00627.
- Czodrowski, P.; Mallinger, A.; Wienke, D.; Esdar, C.; Pöschke, O.; Busch, M.; Rohdich, F.; Eccles, S. A.; Ortiz-Ruiz, M.-J.; Schneider, R.; et al. Structure-Based Optimization of Potent, Selective, and Orally Bioavailable CDK8 Inhibitors Discovered by High-Throughput Screening. J. Med. Chem. 2016, 59 (20), 9337–9349. https://doi.org/10.1021/acs.jmedchem.6b00597.
- https://pymolwiki.org/index.php/Align.
- https://pymolwiki.org/index.php/Cealign
- Scott Midgley. 用Flare Python API实现根据配体叠合来调整蛋白的取向. 墨灵格的博客. 2023-04-13. http://blog.molcalx.com.cn/2023/04/23/protein-transformation.html